Archive
- Home
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
キンギョのシングルセル遺伝子発現解析で進化の謎に迫る
-全ゲノム重複後の遺伝子発現パターンの進化をシングルセルレベルで解析-
プレスリリース

Single-cell transcriptomics of the goldfish retina reveals genetic divergence in the asymmetrically evolved subgenomes after allotetraploidization
Tetsuo Kon, Kentaro Fukuta, Zelin Chen, Koto Kon-Nanjo, Kota Suzuki, Masakazu Ishikawa, Hikari Tanaka, Shawn M. Burgess, Hideki Noguchi, Atsushi Toyoda, Yoshihiro Omori
Communications Biology (2022) 5, 1404 DOI:10.1038/s42003-022-04351-3
長浜バイオ大学の大森義裕教授・今鉄男特任助教(現在:ウィーン大学シニアリサーチフェロー)の研究グループは、国立遺伝学研究所(豊田敦特任教授)、データサイエンス共同利用基盤施設(野口英樹特任教授、福多賢太郎研究員)、愛知県水産試験場弥富指導所、ウィーン大学および米国国立衛生研究所(NIH)と共同でフナを原種とするキンギョの眼球の網膜組織から約2万3千個の細胞を分離し、それぞれの細胞に発現する数万個の遺伝子発現など(シングルセルRNA-seq解析とシングルセルATAC-seq解析)を測定することに成功しました。キンギョの網膜において、全遺伝子が同時に倍加する進化上まれな現象である全ゲノム重複によって倍加した遺伝子のうち、306ペアの遺伝子対の発現が進化し新たな発現パターンを獲得したことを明らかにしました。これは全ゲノム重複後の1400万年という比較的短い時間に細胞レベルで遺伝子の発現パターンの進化が起こることを具体的に示した世界初の報告となります。また、全ゲノム重複後に重複した遺伝子対の発現が重複前の片方のゲノムに偏っているという「非対称サブゲノム進化」がシングルセルレベルで進行していることが証明されました。これらの発見は、現在も謎の多い全ゲノム重複という現象の全体像の解明に向けた重要な一歩となります。また、キンギョをモデルとしたヒトの網膜関連疾患の研究に繋がると期待されます。
本研究成果は、2022年12月26日(月)19:00(日本時間)に国際科学誌「Communications Biology」(オンライン)に掲載されました。
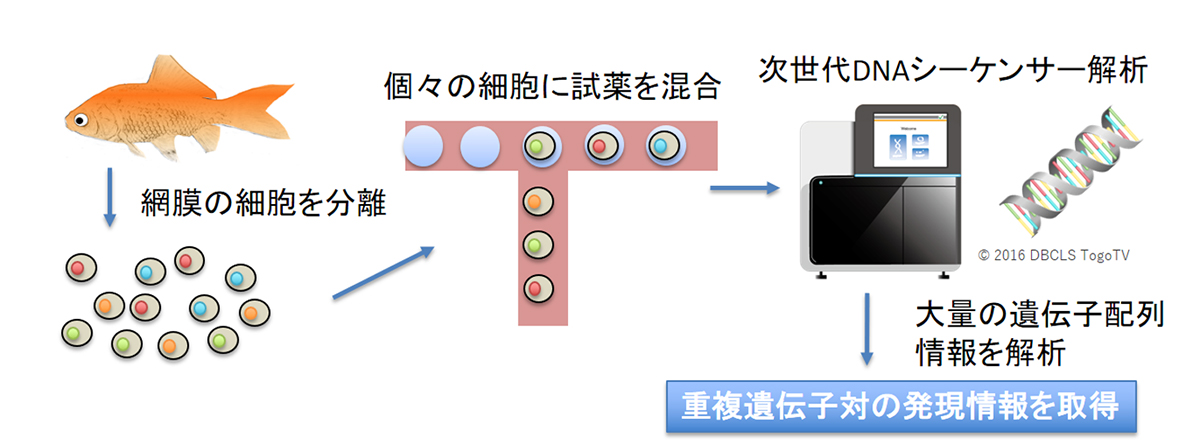
▶ 本研究は先進ゲノム支援(PAGS)の支援を受けておこなわれました。
陸上植物と近縁な接合藻類ヒメミカヅキモのゲノム解読および接合型決定遺伝子の同定に成功
プレスリリース
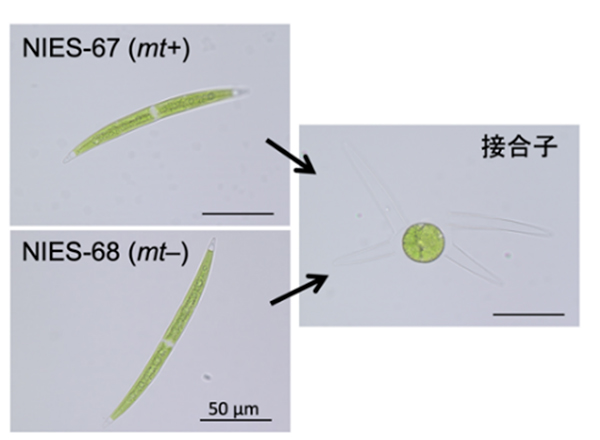
A divergent RWP-RK transcription factor determines mating type in heterothallic Closterium
Hiroyuki Sekimoto, Ayumi Komiya, Natsumi Tsuyuki, Junko Kawai, Naho Kanda, Ryo Ootsuki, Yutaka Suzuki, Atsushi Toyoda, AsaoFujiyama, Masahiro Kasahara, Jun Abe, Yuki Tsuchikane, Tomoaki Nishiyama
New Phytologist 2022 Dec 19 DOI:10.1111/nph.18662
日本女子大学 化学生命科学科 関本弘之教授,情報・システム研究機構 国立遺伝学研究所 豊田敦特任教授,藤山秋佐夫特命教授,および金沢大学 疾患モデル総合研究センター研究基盤支援施設 西山智明助教らの共同研究チームは,接合藻類のヒメミカヅキモの雌雄にあたる2種の接合型のゲノムを解読して比較することにより,ヒメミカヅキモの接合型を決定する遺伝子を特定しました。
さらに,本グループが確立したヒメミカヅキモのゲノム編集技術を用いて,この遺伝子が接合型を決定する遺伝子の本体であることを示しました。
この遺伝子は,陸上植物の有性生殖に重要な遺伝子から接合型決定遺伝子に進化したと考えられます。また,ヒメミカヅキモは陸上植物と最も近縁な藻類の一つであり,本研究は,陸上植物が祖先的な藻類からどのように進化して陸上に適応したのか,その謎の解明への貢献が期待されます。
本研究は、日本学術振興会 科学研究費助成事業(20017013, 23770277, 24247042, 24370038, 25304012, 26440223, 26650147, 15H05237, 16H02518, 16H04836, 16H06378, 18H06367, 18K19365, 19K06827, 19K22446, 19K22448, 20K21451, 21H02549, 22H05177)の支援を受けて行われました。また、文部科学省 科学研究費新学術領域研究 ゲノム支援(221S0002)、同 先進ゲノム支援(16H06279 PAGS)の支援を受けて実施されました。さらに、国立遺伝学研究所のスーパーコンピューターシステムサービス、基礎生物学研究所の生物情報解析システムを利用して、研究が行われました。
本研究の成果は、英国の科学雑誌「New Phytologist」に12月20日(日本時間)にオンライン掲載されました。
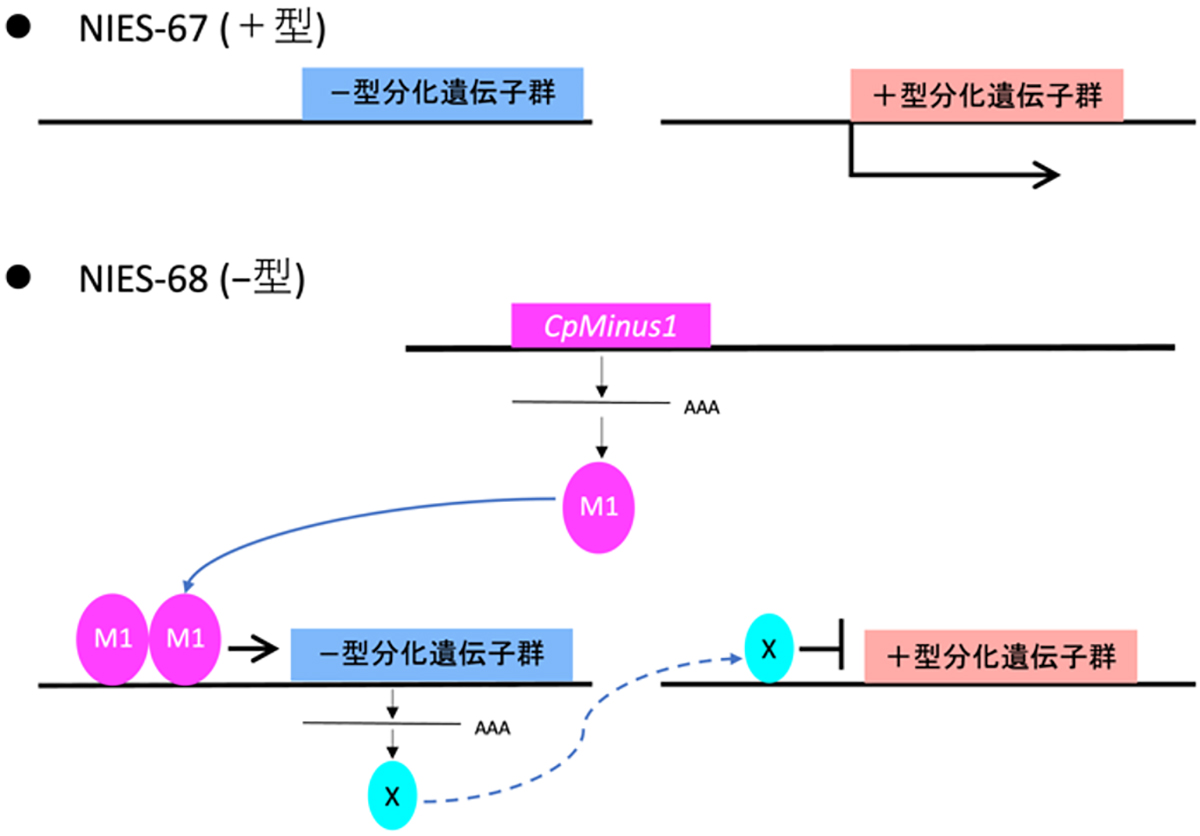
−型細胞では、−型特異的遺伝子であるCpMinus1の発現により、CpMinus1タンパク質(図中のM1)が産生されると、−型への分化に必要な遺伝子群(図中の−型分化遺伝子群)の発現が誘導される。誘導される遺伝子の中には、+型への分化に必要な遺伝子群(図中の+型分化遺伝子群)を抑制するタンパク質(図中のX)をコードしているものが含まれると考えられ、Xの影響により、+型への分化が抑制される。+型細胞では、CpMinus1が存在しないため、−型分化遺伝子群が発現せず、 +型分化遺伝子群の発現も抑制されない。AAAで示したものは、遺伝子から転写されたmRNAを示す。
生薬「甘草」の染色体スケールのゲノム解読に成功
ー薬効成分を作る遺伝子クラスターを解明ー
プレスリリース

Chromosome-scale genome assembly of Glycyrrhiza uralensis revealed metabolic gene cluster centered specialized metabolites biosynthesis
Amit Rai, Hideki Hirakawa, Megha Rai, Yohei Shimizu, Kenta Shirasawa, Shinji Kikuchi, Hikaru Seki, Mami Yamazaki, Atsushi Toyoda, Sachiko Isobe, Toshiya Muranaka, Kazuki Saito
DNA Research DNA Research (2022) 29, 1–14 DOI:10.1093/dnares/dsac043
理化学研究所(理研)環境資源科学研究センター統合メタボロミクス研究グループのアミット・ライ研究員、斉藤和季グループディレクター、かずさDNA研究所の平川英樹主任研究員、千葉大学大学院薬学研究院の山崎真巳教授、大阪大学大学院工学研究科の村中俊哉教授、国立遺伝学研究所の豊田敦特任教授らの共同研究グループは、漢方薬や天然甘味料の原料として使われる重要生薬の甘草(カンゾウ)の染色体スケールの高品質ゲノム配列を解読しました。
本研究成果は、今後、バイオテクノロジーを用いた甘草の品種改良や薬効成分の生産向上に役立つと期待できます。
甘草は、さまざまな漢方薬に最も頻繁に配合されるマメ科植物を基原とする生薬です。甘草には、抗炎症作用や痛み、咳を鎮める効果をはじめ、多数の薬効があります。また、根に含まれる主要成分のグリチルリチンは医薬品、天然甘味料などの原料として世界的に需要が高まっています。質の高い甘草のゲノム配列を解読できれば、ゲノム情報に基づいた効率的な育種などが可能になると期待されていました。
今回、共同研究グループは最先端のシーケンス技術を駆使して、ウラル甘草[2]の染色体スケールの高品質ゲノム解読に成功しました。そして、グリチルリチンなどの生合成に関わる重要な遺伝子が染色体の狭い領域に局在することを確認しました。
本研究は、日本学術振興会(JSPS)科学研究費助成事業基盤研究(S)「薬用資源植物の化学的多様性のゲノム起源(研究代表者:齊藤和季)」による助成を受けて行われました。
本研究は、科学雑誌『DNA Research』のオンライン版(12月20日付)に掲載されました。
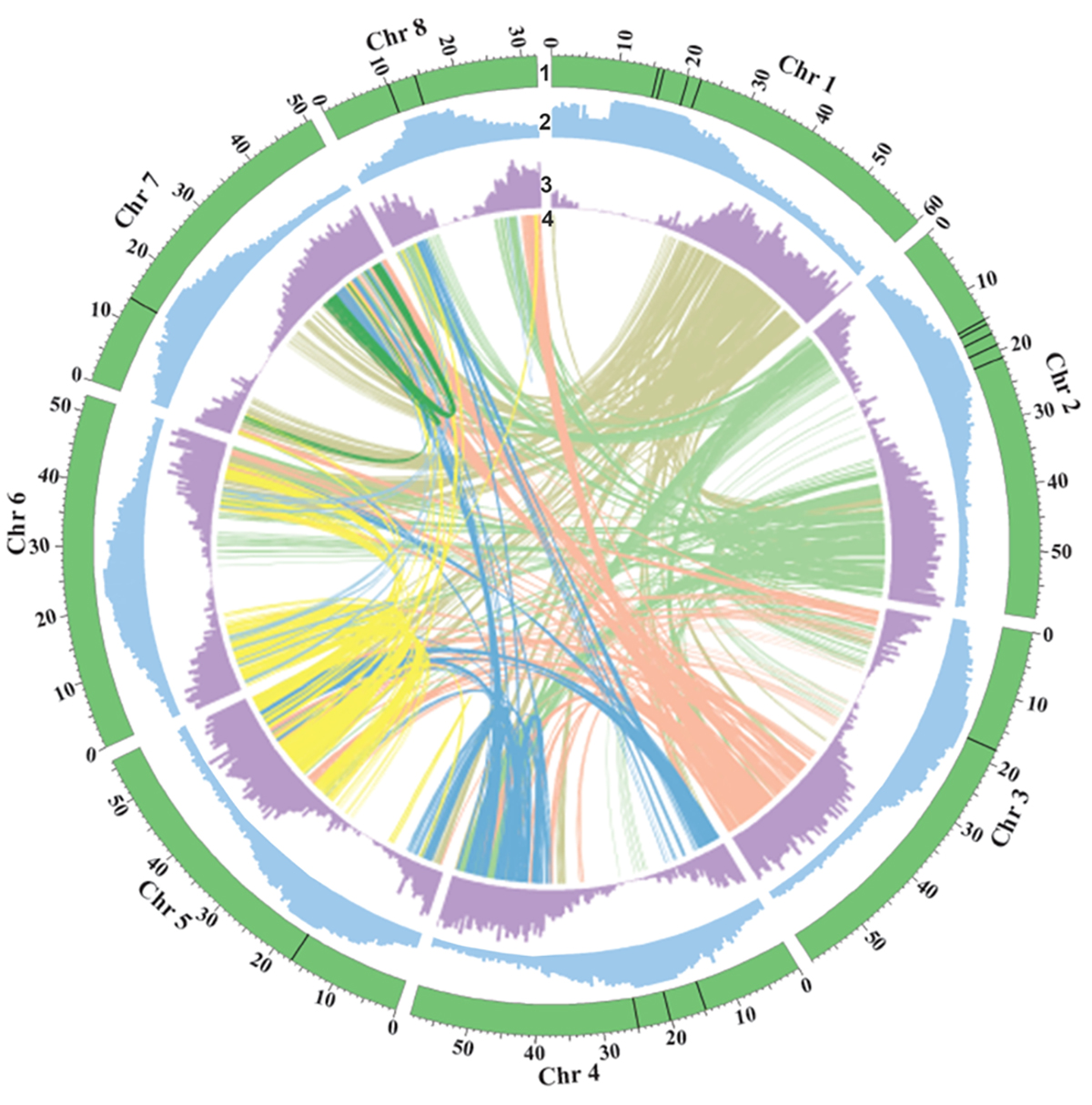
1(緑)は8本の染色体に割り当てた塩基配列。単位はメガ塩基対で、黒線はギャップの位置を示す。2(青)は繰り返し配列の頻度、3(紫)は信頼性の高い予測遺伝子の分布、4はゲノム内のシンテニーを表す。
▶ 理化学研究所が公開する動画はこちら
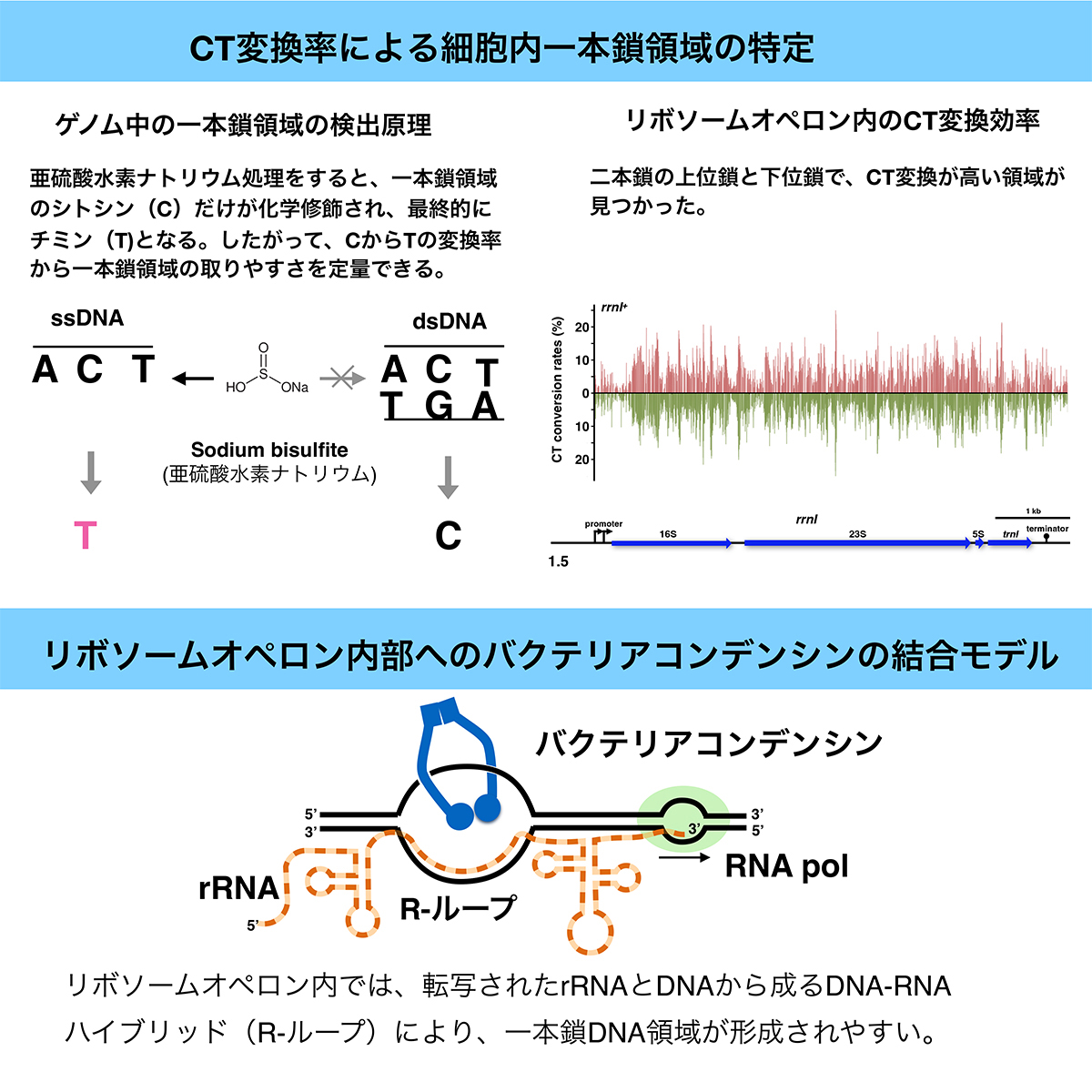
リボソームオペロン内のバクテリア染色体の凝縮に必要な一本鎖領域
Profiling a single-stranded DNA region within an rDNA segment that affects the loading of bacterial condensin
Koichi Yano, Hideki Noguchi, Hironori Niki
iScience (2022) 25, 105504 DOI:10.1016/j.isci.2022.105504
バクテリアの染色体は、DNA複製と連動して新生DNA鎖から順番に折りたたまれ、仕分けられることで娘細胞に分配される。このDNA鎖の折れたたみ、すなわちDNA凝縮を担うのが、バクテリアコンデンシンである。バクテリアコンデンシンはDNA複製開始点の近傍に散在するリボソームDNAオペロン内に集まることで、新生DNA鎖からの凝縮が正しく行われていると私たちは考えている。そして、バクテリアコンデンシンのリボソームDNAオペロン内に集積には、一本鎖DNA結合という特性が関係すると考えてきた。今回、細胞内で一本鎖DNA領域を検出する方法を使い、リボソームRNAオペロン内に一本鎖DNA領域が形成されること、特にプロモーターの下流の領域での一本鎖DNA領域の形成がバクテリアコンデンシンのリボソームRNAオペロン内の集積に重要であることを明らかにした。
多くのバクテリアではリボソームRNAオペロンがDNA複製開始点の近傍に散在する。これはリボソームRNAの増産のためと考えられてきたが、そればかりではなく染色体の分配にも役立っている。
本研究は、国立遺伝学研究所 微生物機能研究室 矢野晃一研究員(現、立教大学理学部、特任助教)、仁木宏典教授、及びデータサイエンス共同利用基盤施設ゲータ解析支援センター 野口英樹センター長らによる共同研究の成果によるものです。
ゲノムダイナミクス研究室 総研大生・南さんが第45回日本分子生物学会年会で「MBSJ2022 サイエンスピッチアワード」を受賞
ゲノムダイナミクス研究室の南克彦さん(総研大遺伝学専攻D5・SOKENDAI特別研究員)が、2022年11月30日~12月2日に千葉県・幕張で開催された、第45回日本分子生物学会年会でサイエンスピッチ(3分間の口頭プレゼンテーション)をおこない、優秀な演題に授与される「MBSJ2022サイエンスピッチアワード」を受賞しました。
▶ 受賞発表タイトル:
複製依存的ヒストン標識によるユークロマチン/ヘテロクロマチン特異的なクロマチン動態解析
▶ 学会HP:第45回日本分子生物学会年会

赤シソの高精度なゲノム配列情報を決定
〜デジタル育種に向けた基盤情報を取得〜
プレスリリース
A highly contiguous genome assembly of red perilla (Perilla frutescens) domesticated in Japan
Keita Tamura, Mika Sakamoto, Yasuhiro Tanizawa, Takako Mochizuki, Shuji Matsushita, Yoshihiro Kato, Takeshi Ishikawa, Keisuke Okuhara, Yasukazu Nakamura, Hidemasa Bono
DNA Research 2022 Nov 16 DOI:10.1093/dnares/dsac044
広島大学大学院統合生命科学研究科の田村啓太研究員、坊農秀雅特任教授、国立遺伝学研究所の坂本美佳特任研究員、谷澤靖洋助教、望月孝子特任研究員、中村保一教授、広島県立総合技術研究所農業技術センターの松下修司主任研究員、三島食品株式会社、プラチナバイオ株式会社からなる研究グループは、最新のDNA配列解析技術を用いて、国内で栽培されてきた赤シソの高精度なゲノム配列情報を決定しました。
生物の基本的な設計図ともいえるゲノム配列情報は、生物の仕組みを明らかにする基礎研究のみならず、より優れた特性をもつ作物などを作り出す品種改良(育種)技術など、産業利用においても重要な基盤となるものです。
研究グループは、三島食品株式会社が育種を進めてきた赤シソ品種「豊香3号」からゲノムDNAを抽出し、これまでドレードオフの関係にあった配列の「長さ」と「精度」の両立を実現した最新のDNA配列解析技術を用いて、赤シソゲノムの配列断片を取得しました。この配列断片を、ソフトウェアを使って連続した領域を連結し、20本の染色体に対応する巨大な配列にまとめました。この巨大配列は、ギャップと呼ばれる未決定領域が1本あたり4か所以内と極めて少なく、うち7本はギャップのない完全な配列として得られました。
本研究により、赤シソについてゲノム情報を利用した「データ駆動型ゲノム育種(デジタル育種)」を実施するための基盤情報が得られたことから、今後赤シソのさらなる高機能化や栽培特性の改良に向けた研究開発の加速が期待されます。
本研究は、広島県ものづくり価値創出支援補助金、科学技術振興機構(JST)共創の場形成支援プログラム(COI-NEXT)「Bio-Digital Transformation(バイオDX)産学共創拠点」(JPMJPF2010)、および日本学術振興会(JSPS)科学研究費助成事業挑戦的研究(萌芽)(21K19118)によって実施されました。
本研究成果は、2022年11月16日にDNA Research誌に掲載されました。
遺伝研の貢献
大量遺伝情報研究室が有するこれまでの植物ゲノム解析の経験を活かして、塩基配列のアセンブルと遺伝子アノテーションを広島大学と共同で実施し、ゲノムデータベースの構築を行っています(近日公開予定)。ゲノム解析には遺伝研スーパーコンピュータを活用しました。

ヒトゲノム複製におけるDNAポリメラーゼ間の分業と複製開始領域の同定
-ゲノム安定性とDNA複製機構の関わり合い-
プレスリリース
Global landscape of replicative DNA polymerase usage in the human genome
Eri Koyanagi#, Yoko Kakimoto#, Tamiko Minamisawa#, Fumiya Yoshifuji#, Toyoaki Natsume†, Atsushi Higashitani, Tomoo Ogi, Antony M. Carr, Masato T. Kanemaki, Yasukazu Daigaku*
#同等貢献,*責任著者
Nature Communications (2022) 13, 7221 DOI:10.1038/s41467-022-34929-8
ヒトではDNA合成反応に働く酵素(DNAポリメラーゼ)が17種類存在し,それらのDNA合成反応の効率・正確性はそれぞれ異なり,ゲノム複製におけるポリメラーゼ間の分業は,ゲノム情報の安定性を決める主な要因です。特に,がん細胞においては,多くのDNAポリメラーゼ遺伝子に変異が生じ, DNAポリメラーゼの使われ方が大きく変化することが報告されていますが,その実態は明らかになっていません。我々は,ヒト培養細胞を使用して,全ゲノムにわたりDNAポリメーラの機能を解析する方法Polymerase usage sequencing(Pu-seq)法を開発し,主要なDNAポリメラーゼと言われていたPolε(イプシロン)とPolα(アルファ)それぞれが主にリーディング鎖・ラギング鎖合成に関与することを明らかにしました。また,これらのポリメラーゼのプロファイルを組み合わせた解析から,ゲノム上に多数存在する複製開始領域を今までにない精度で予測することにも成功しました。今後は,本研究で開発されたPu-seq実験によって,がん化などによる細胞の状態変化によって生じるゲノム複製機構の変遷を明らかにし,ヒトなどの大きなゲノムを持つ生物が潜在的に有するDNA複製の柔軟性(flexibility),また,それに伴う脆弱性(fragility)の全容解明に向け,研究を大きく進めていく予定です。
本研究は、以下の支援を受けて実施されました。
・国立研究開発法人科学技術振興機構(JST)戦略的創造研究推進事業 さきがけ 研究領域:ゲノムスケールのDNA設計・合成による細胞制御技術の創出「レプリケーター領域の構成的理解を介したゲノム複製の制御技術の確立(研究代表者:大学保一)」
・国立研究開発法人科学技術振興機構(JST) 創発的研究支援事業 塩見パネル「ゲノム複製におけるDNAポリメラーゼ間の協調的機能(研究代表者:大学保一)」
・日本学術振興会(JSPS)科学研究費補助金 JP16H06151,JP20H03233,JP21K19203(研究代表:大学保一)
・日本学術振興会(JSPS)科学研究費補助金 (JP20H05396JP21H04719 and JP22H04703(研究代表:鐘巻将人)
・内藤記念科学振興財団 研究助成(研究代表者:大学保一)
・武田科学振興財団 ライフサイエンス研究助成(研究代表者:大学保一)
・国立遺伝学研究所 共同研究「NIG-JOINT」(研究代表者:大学保一)
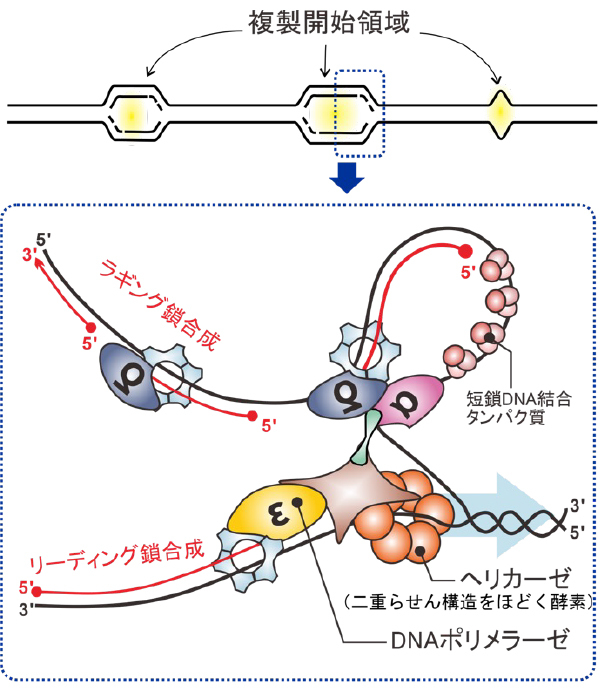
ヘリカーゼがほどいた一本鎖DNA上でPol εがリーディング鎖,PolαとPolδラギング鎖合成を担う。
新分野創造センターのテニュアトラック教員がテニュア獲得
2022年12月1日付けで新分野創造センターの久保 郁 准教授がテニュアを獲得しました。
遺伝研の新分野創造センター(Center for Frontier Research)は,「あたらしい人材」と「あたらしい分野」を同時に育成するためのインキュベーションセンターです。遺伝研の卓越した研究環境や様々なサポートを活用して若手の優れた研究者がテニュアトラック独立准教授として研究室を運営し、遺伝学とその周辺領域に新しい分野を開拓する研究を行っています。テニュアを獲得した教員は遺伝研に新しい研究部門を創り、自らが創成に貢献している新分野を牽引していきます。

- 久保 郁 准教授















