国立遺伝学研究所が取り組む新型コロナウイルス・全ゲノム解析の紹介
国立遺伝学研究所は新型コロナウイルス感染症(COVID-19)の拡大に際して、新型コロナウイルス(SARS-CoV-2)の全ゲノム解析による分子疫学調査(SARS-CoV-2 RNA全ゲノム解析)を静岡県と連携・協働して進めています。感染拡大にともないウイルスゲノムには様々な変異が入ります。その変異パターンを解析することで、感染ルートの解明など感染症対策に大きく貢献できると考えています。
![]() 「静岡県新型コロナウイルスのゲノム解析の結果」はこちらから(情報更新日2022年10月24日)
「静岡県新型コロナウイルスのゲノム解析の結果」はこちらから(情報更新日2022年10月24日)
ゲノム解析を行う意義
一般的に、ウイルスや細胞がゲノムを複製する際には、わずかな割合で複製ミスが生じ、ゲノムのA, G, C, T(U)の塩基の「ならび」が変化します。この変化を「変異」と呼びます。塩基のならびが変化したウイルスや生物の個体を「変異型」と呼びます。変異の中には遺伝子から作られるタンパク質の働きに影響を及ぼすものがあります。新型コロナウイルスの場合、感染時に重要な働きをするスパイクタンパク質によって感染性が変わることが多いため、スパイクタンパク質の変異が注目されています。
ウイルスの塩基配列に生じた全ての変異を特定できる方法が「ゲノム解析」です。つまり、配列情報から、これまで明らかにされてきた新型コロナウイルスの配列とどの部分が異なるのか1塩基レベルで判明します。これによって陽性となった被検者がどのような変異型のウイルスに感染しているのかがわかります。世界中で公開されている変異型ウイルスの配列情報と比較することによって個々の変異型ウイルスがどのように伝搬して来たか推測することも可能です。スパイクタンパク質の領域に未知のアミノ酸変異があった場合には、それらアミノ酸変異による感染性への影響を研究する契機にもなり得ます。
新型コロナウイルスの検査方法 ~PCR検査、抗原検査、抗体検査、ゲノム解析~
新型コロナウイルスを検査する方法には、現在、PCR検査、抗原検査、抗体検査、ゲノム解析があり、それぞれの方法に一長一短があります。
「PCR検査」は新型コロナウイルスゲノムの特定の領域をもとに同じ配列をPolymerase Chain Reaction (ポリメラーゼ連鎖反応、PCR反応)によって増幅し、その量を定量します。つまり、PCR検査でわかるのは検体中の「ウイルス塩基配列の存在」です。欠点として、感染しているにもかかわらずゲノムが採取できず検出できなかったり、逆に、感染性のないウイルスの残骸を検出してしまうという限界もあります。一方で、増幅の元となる配列の選び方によって、既知の変異型を検出する方法にも用いられます。
「抗原検査」は新型コロナウイルスのタンパク質に反応する「抗体」を用いて、検体中の「抗原」、すなわちウイルスの構成タンパク質を検出する方法です。概して抗原検査はPCR検査に比べて検出感度が落ちるとされていますが、PCRのような特殊な装置を必要とせず比較的迅速に結果が得られるという利点があります。抗原検査は季節性インフルエンザでは確立された診断方法です。
「抗体検査」は新型コロナウイルスに対する「抗体」の有無を調べる検査です。抗体検査でわかるのは、過去に「新型コロナウイルスに感染した」という「経験」です。現在のところ、抗体検査に医薬品としての精度が確立していないことから、診断薬としての使用は推奨されていません。
「ゲノム解析」では、被検者の新型コロナウイルスの塩基配列情報を全て明らかにすることができます。しかし解析には、シーケンサーと呼ばれる高額な大型機器が必要になるばかりでなく、得られたデータを情報解析するための高性能なコンピュータや解析技術も必要となります。一連の解析には時間もかかるため、他の方法のように多くの被検者を検査することができません。しかしながら、得られた配列データはワクチンや抗ウイルス薬の開発、PCR反応時のプライマー設計、変異速度の見積もり、変異型の予測、ウイルス遺伝子の機能解明、感染症疫学調査、ヒトゲノムとの関連性、など、多様な分野において欠くことのできない基盤情報となり、新型コロナウイルス感染症の克服だけでなく、新興再興感染症の研究などにも大いに役立ちます。
ゲノム解析の実際
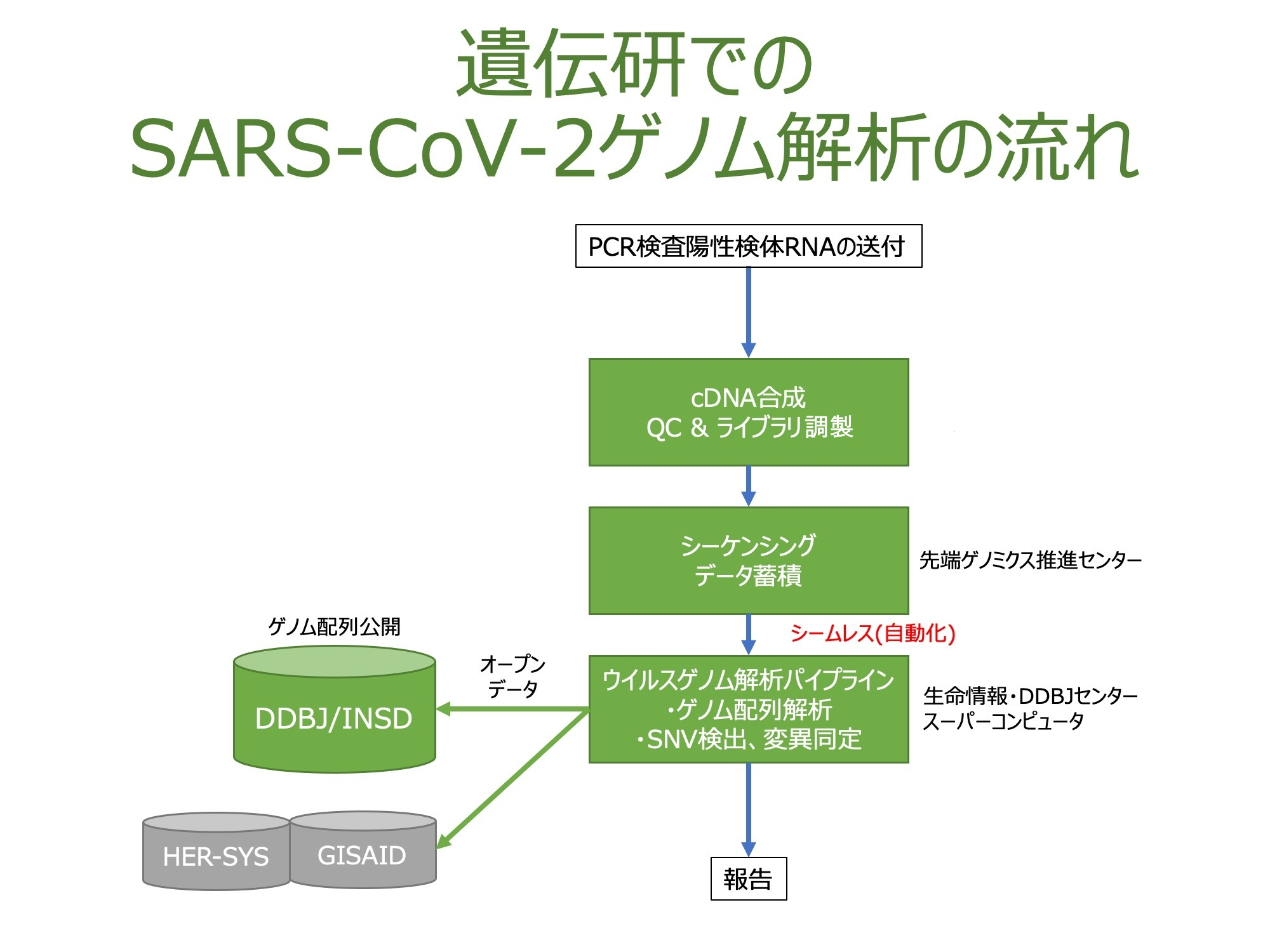
新型コロナウイルスのゲノムを解析するには、PCR検査で陽性になった被検者の生体試料から抽出された新型コロナウイルスのRNAを利用します。まず、RNAの並びをDNAにコピー(逆転写)し、そのDNAを鋳型にして3万塩基あるウイルスゲノム全体を100ほどの短い領域にわけてPCR増幅します。増幅した短い領域を、リードと呼びます。作成したリードを次世代シーケンサーを使用して塩基配列を読み取ってから、コンピュータによってリードをつなぎ合わせて、ウイルスゲノム全体を再構築します。構築したゲノム配列を、公開されている他のゲノム配列と比較することで、ゲノム上に生じた変異を全て明らかにする事ができます。
遺伝研では、次世代シーケンサーが産出した配列データは即座に遺伝研スーパーコンピュータに送られ、新たに開発した解析パイプラインにより自動的に解析されます。最後は研究者が解析結果を目視で確認します。遺伝研で採用している実験プロトコルおよび解析プロトコルは近日中に公開予定です。また、解析パイプラインはGitHub上で公開を予定しており、どなたでも利用して頂く事が可能です。
データ公開の意義
解析で明らかになったゲノム配列は広く一般公開することが重要です。その配列を利用して、世界中の研究者が新たな変異型の発見や感染経路の特定に利用できるからです。私達が実施する解析も、世界中の研究者によって公開された情報をもとに実施しています。国立遺伝学研究所は、ウイルスに限らない塩基配列情報を世界中で共有するためのデータベース(DNA Data Bank of Japan)を30年以上運営しています。ウイルスゲノムの情報はこのDDBJを通じて公開するだけでなく、コロナウイルス専用のデータベース(GISAID)からも公開します。
▶ Japan Covid-19 Open Data ConsortiumによるSARS-CoV-2ゲノム配列登録情報 (DDBJ)
静岡県新型コロナウイルスのゲノム解析の結果
遺伝研では、静岡県からの要請を受け、県内で発生したSARS-CoV-2のゲノム解析を進めています。ゲノム解析によりSARS-CoV-2の配列情報を明らかにすることでウイルスの系統が判明します。
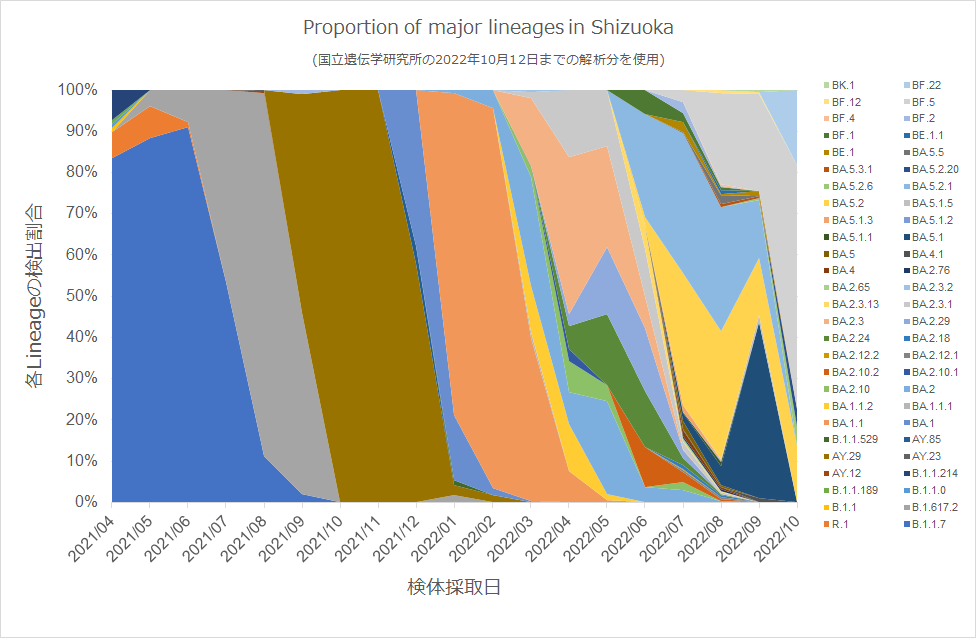
以下のグラフは、静岡県全体で発生したSARS-CoV-2の各系統(Lineage)について、検出頻度と時間推移を示しています。 (縦軸は「割合」を、横軸は「検体採取日」を示す。) ※注意:本グラフは、遺伝研がゲノム解析した検体のみの結果を示す。
以下の表は、遺伝研がゲノム解析を実施した、地域ごと月ごとの検体数を示しています。
国立遺伝学研究所による静岡県のSARS-CoV-2検体のゲノム解析結果(2022年10月12日までのデータ) : 東部月ごと
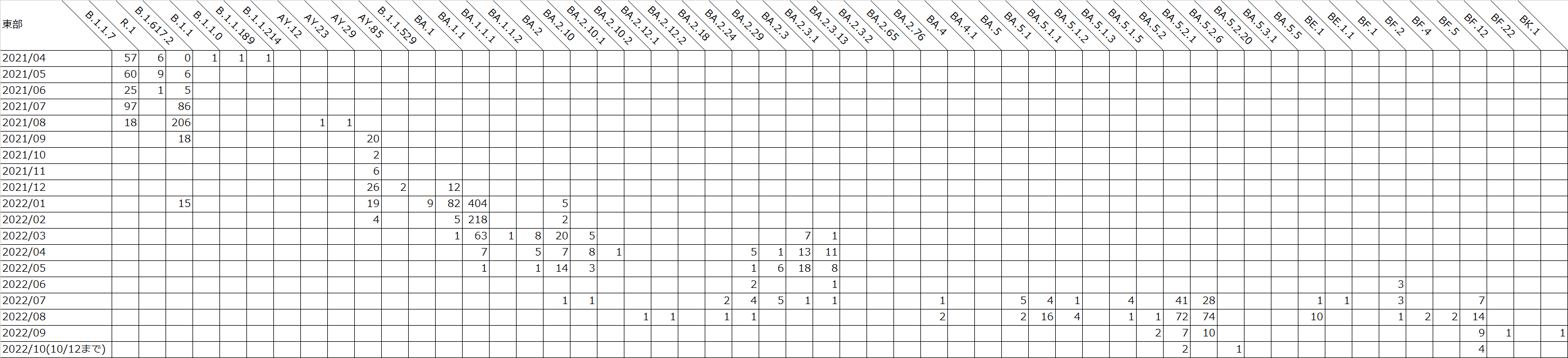
表はクリックで拡大します。
国立遺伝学研究所による静岡県のSARS-CoV-2検体のゲノム解析結果(2022年10月12日までのデータ) : 中部月ごと
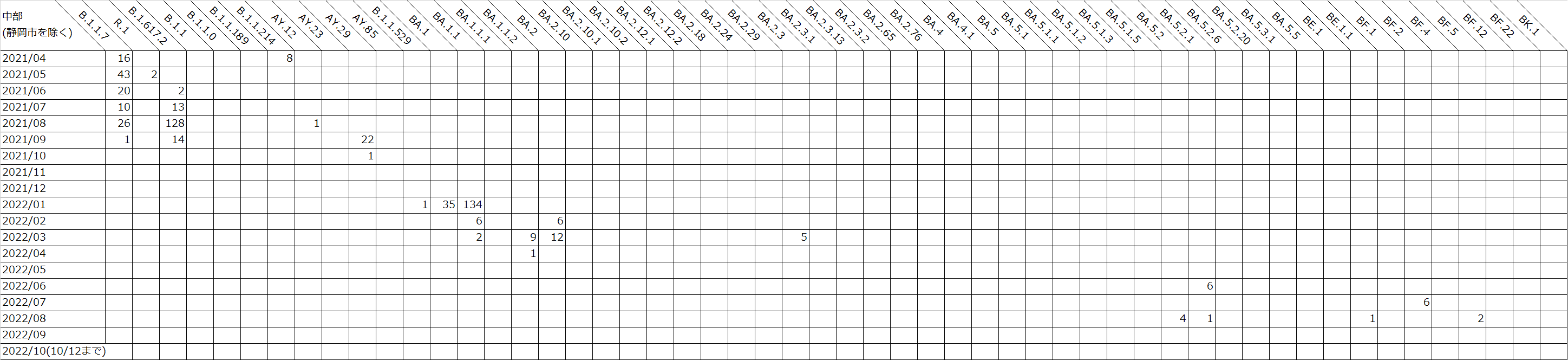
表はクリックで拡大します。
国立遺伝学研究所による静岡県のSARS-CoV-2検体のゲノム解析結果(2022年10月12日までのデータ) : 西部月ごと
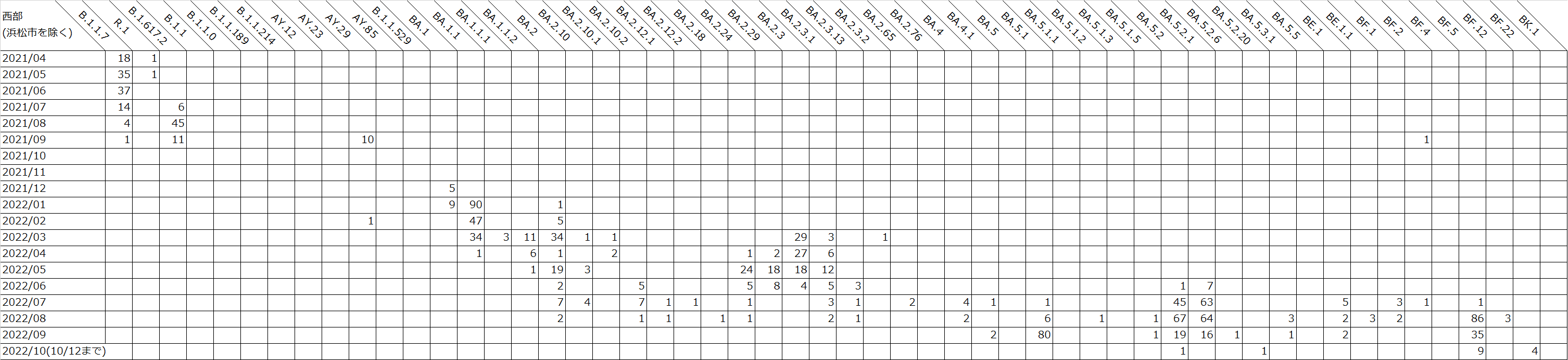
表はクリックで拡大します。
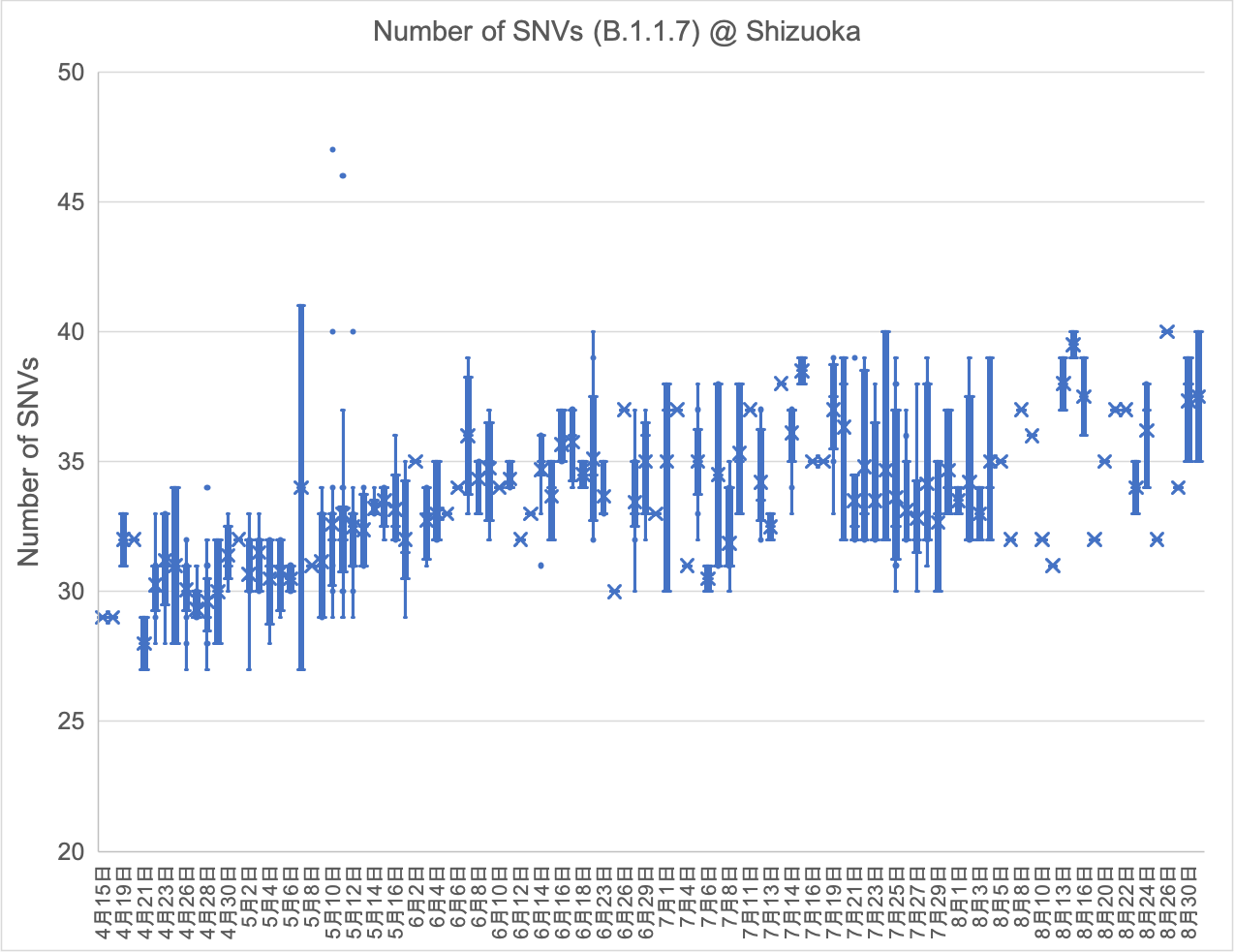
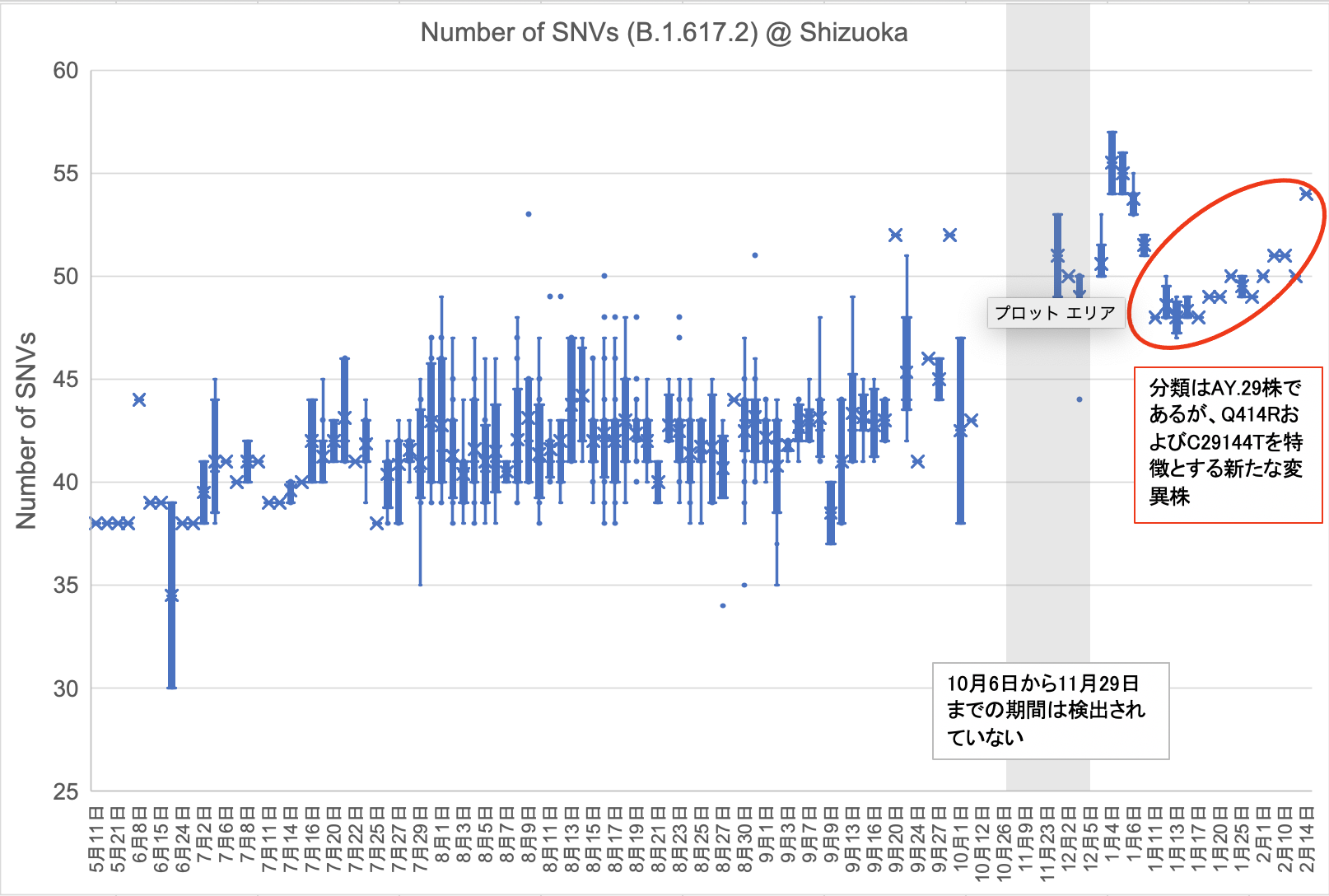
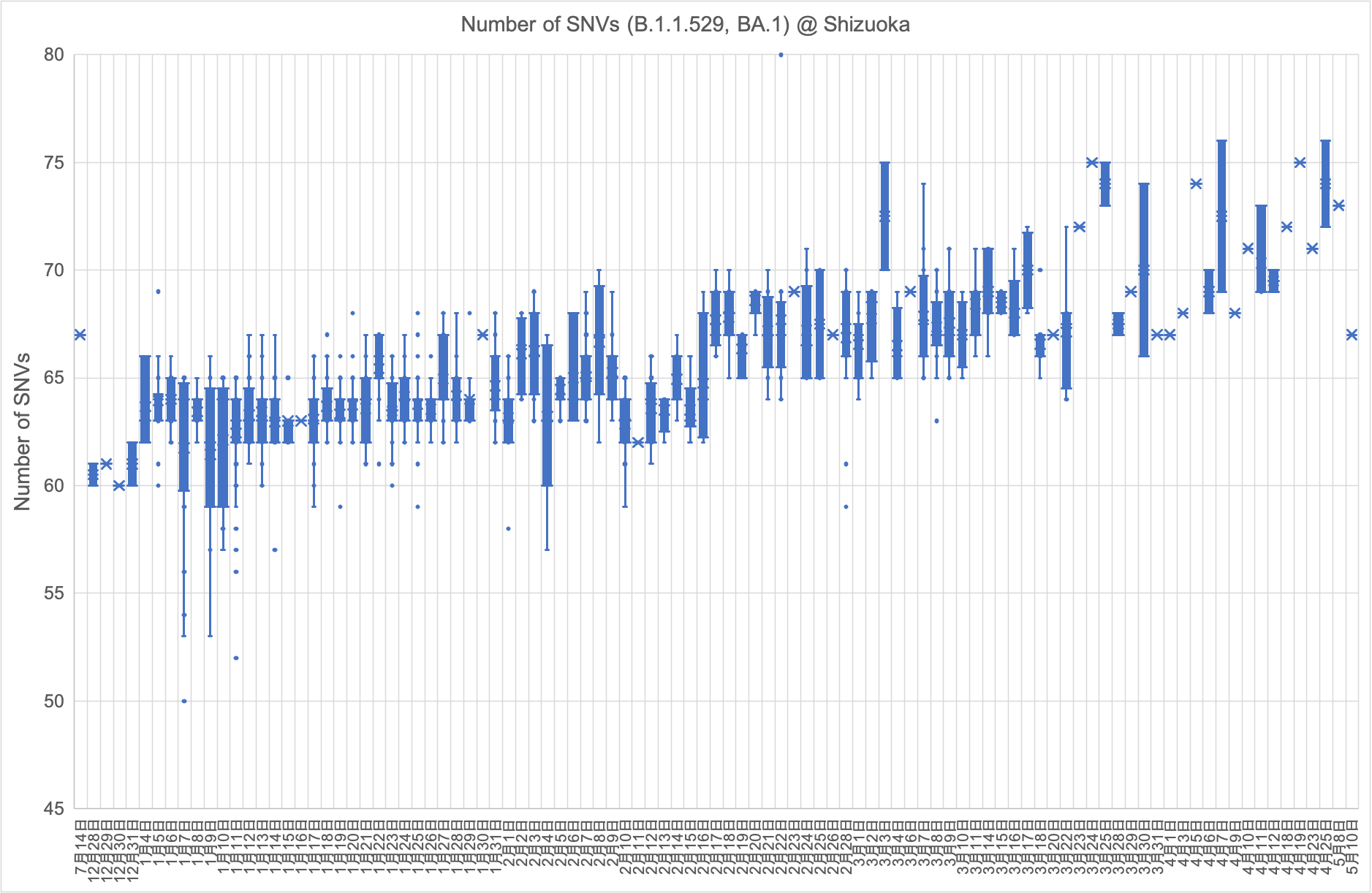
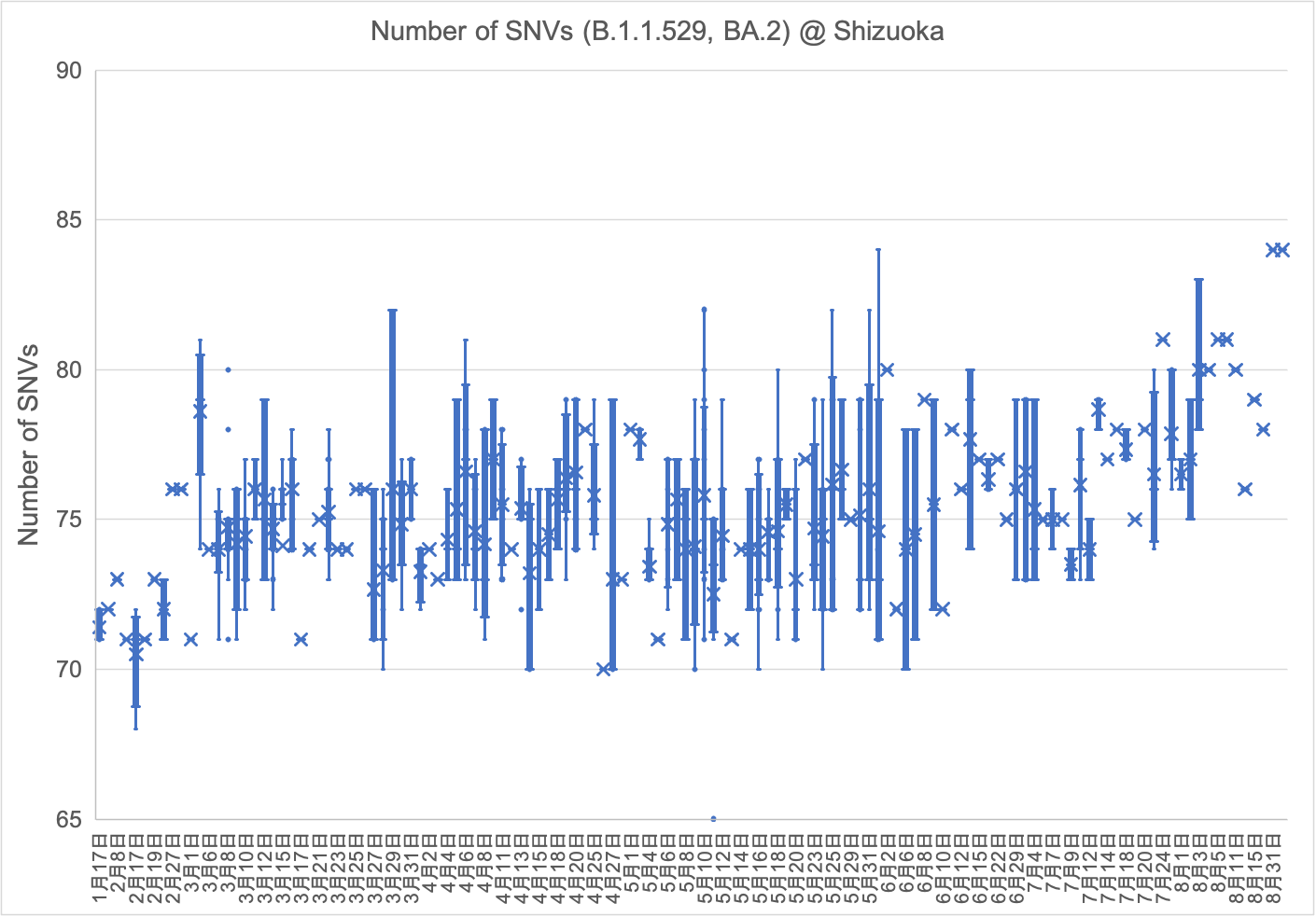
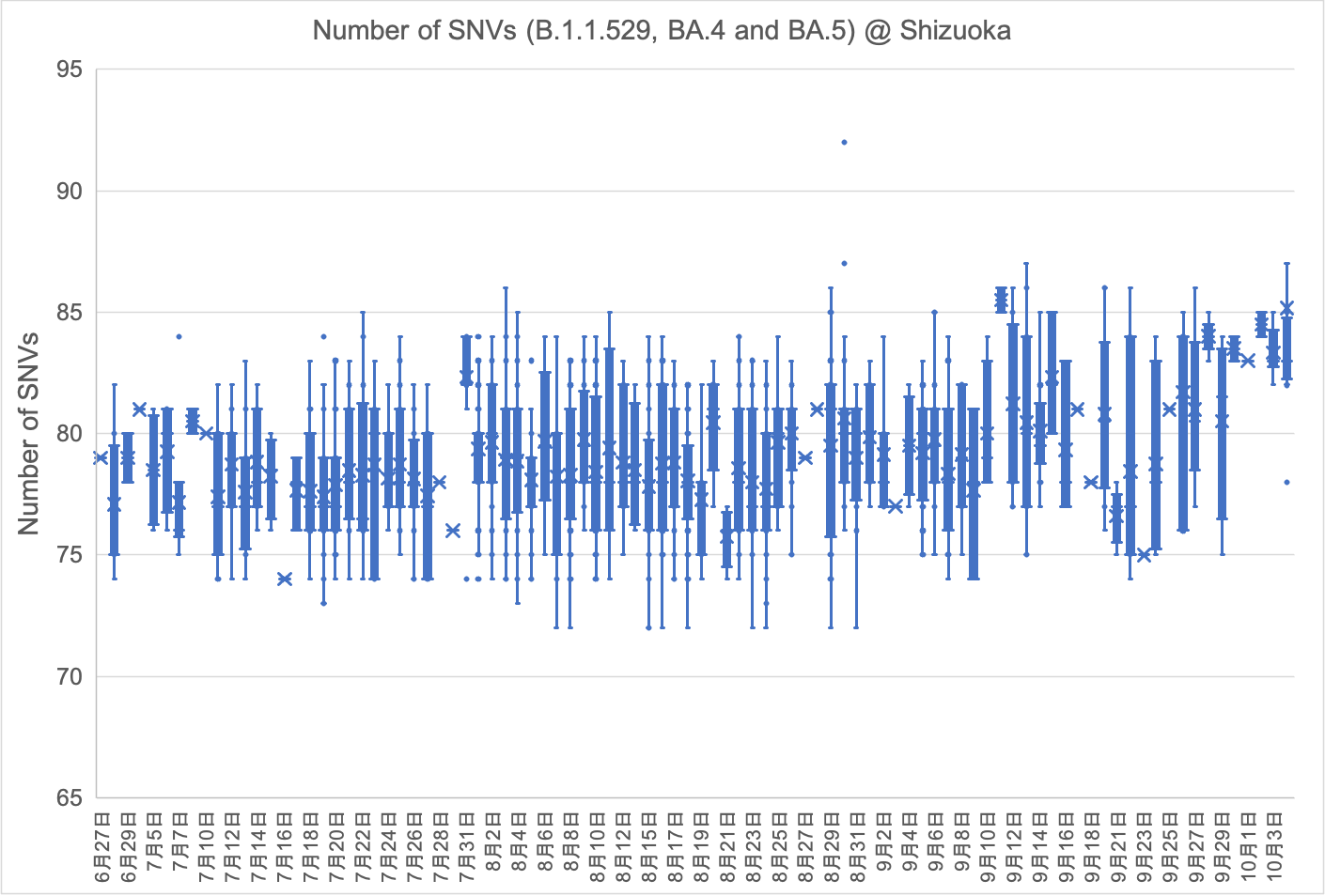
「B.1.1.7株(アルファ型)、B.1.617.2株(デルタ型)およびB.1.1.529株(オミクロン型)のSNV(1塩基変異)数の推移」 (2022年10月20日)
以下の図は、静岡県内で検出されたB.1.1.7株(アルファ型)およびB.1.617.2株(デルタ型)のSNV(1塩基変異)数の推移を示しています。新型コロナウイルスはヒトからヒトに感染を繰り返すことでゲノムに変異が入ります。アルファ型は2021年のゴールデンウィーク頃に急速に変異が蓄積しており、6月以降は一定数を維持しています。デルタ型は2021年7月中旬からSNV数が上昇し、その後増加と減少を繰り返していることから、「県外から新たなデルタ株が流入→それらが県内で感染拡大」、ということを繰り返している結果だと考えられます。(2021年9月15日)
これまで一定数を維持していたアルファ型のSNV数が8月下旬から微増傾向にあります。デルタ型の流行に隠れて感染を繰り返し、徐々にゲノムに変異が蓄積していることから、今後もアルファ型の動向も注視する必要があります。(2021年9月27日)
これまでSNV数の増加と減少を繰り返していたデルタ株ですが、9月中旬以降にSNV数が急増しました。感染者数の減少によりゲノム解析した検体数も少ないため、この傾向が一時的なものなのか継続するのかを判断するためには、引き続き観察を続ける必要があります。(2021年10月19日)
2021年12月28日に静岡県内で採取した検体からB.1.1.529株(オミクロン型)が検出されました。その後の急速な感染拡大に伴い変異数も微増しています。(2022年2月14日)
オミクロン型と比較して圧倒的に数が少ないものの、AY.29株(デルタ型)が検出され続けています。静岡県では2021年10月6日から11月29日までAY.29株は検出されていませんでしたが、11月30日に検出されたAY.29株ではゲノム全域の変異が約5箇所増えていました。一方、今年の1月11日以降に検出されているAY.29株は、ゲノム全域の総変異数が少なく、Q414RおよびC29144Tの変異を同時にもつ、これまでのAY.29株とは異なるタイプのデルタ型です。オミクロン型の流行に隠れて観察が難しいものの、この新しいデルタ型も今後注視する必要があります。(2022年2月14日)
県内で検出されたオミクロン型は、2021年12月からBA.1株、次いで2月からBA.2株で構成されています。BA.1株のSNV数は微増を続け、3月初旬から急速に増大した後、5月中旬以降は検出されなくなりました。BA.2株のSNV数も微増を続け、7月初旬から急速に増大した後、9月以降は検出されなくなりました。6月27日には新たにBA.4株が検出されました。SNV数は初検出以降、ほとんど変化がありませんでしたが、9月初旬から微増しはじめ、10月初旬から急速に増大しています。(2022年10月20日)
ゲノム多様性から予測した「デルタ株」の地理的推移(時系列) 2021年8月20日
静岡県内で検出された「デルタ株」の全ゲノム情報と日本全国で検出された「デルタ株」の全ゲノム情報を統合し、ウイルス株の親子関係を推定するハプロタイプ解析を実施しています。検出地域や検出日と併せて解析する事で、県内で検出されたウイルス株がどの地域からいつ頃流入したかを推測する事ができます。以下の動画は、auspice.us および nextstrain.org を利用して作成しています。 (データ解析:阿部貴志(新潟大学教授)、有田正規(遺伝研教授)、ほか)
ゲノム多様性から予測した「オミクロン株」の地理的推移(時系列) (2022年10月18日)
静岡県内で検出された「オミクロン株」の全ゲノム情報と日本全国で検出された「オミクロン株」の全ゲノム情報を統合し、ウイルス株の親子関係を推定するハプロタイプ解析を実施しています。検出地域や検出日と併せて解析する事で、県内で検出されたウイルス株がどの地域からいつ頃流入したかを推測する事ができます。以下の動画は、auspice.us および nextstrain.org を利用して作成しています。 (データ解析:阿部貴志(新潟大学教授)、有田正規(遺伝研教授)、ほか)
静岡県との連携・協働に関する過去のアナウンス
▶ 新型コロナウイルスの全ゲノム解析実施に関する覚書を静岡県と締結 2021年4月30日
▶ 静岡県の協力要請に基づく新型コロナウイルスの全ゲノム解析の開始 2021年4月26日
補足説明
▶ 新型コロナウイルスとは
コロナウイルスは一般的に風邪症状を誘発するRNAウイルスとして知られます。なかでも新型コロナウイルス(SARS-CoV-2)は肺炎症状を含む新型コロナウイルス感染症(Covid-19)を引き起こします。
▶ 新型コロナウイルスのゲノムとタンパク質のはたらき
新型コロナウイルスのゲノムは一本のRNAからできています(例えばインフルエンザウイルスのゲノムは8本のRNAから成ります)。この一本鎖RNAは約3万文字の塩基のならびで構成されていて、mRNAとして機能します。このRNAには「ウイルスゲノムを複製するためのRNA重合酵素」や「ウイルスが作ったタンパク質を切断して活性化させるタンパク質切断酵素」、「ウイルスの殻に存在してウイルスの感染性を決めるスパイクタンパク質」、「殻をつくるタンパク質」などが記載されています。















