Archive
- Home
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
ヒトテロメア配列を認識するポリアミド化合物の特異性の向上
(i) Structural Evaluation of Tandem Hairpin Pyrrole–Imidazole Polyam-ides Recognizing Human Telomeres
Hirata, A., Nokihara, K., Kawamoto, Y., Bando, T., Sasaki, A., Ide, S., Maeshima, K., Kasama, T., and Sugiyama, H. Journal of the American Chemical Society (JACS), July 18, 2014. DOI: 10.1021/ja506058e
(ii) Tandem Trimer Pyrrole-Imidazole Polyamide Probes Targeting 18 Base Pairs in Human Telomere Sequences
Kawamoto, Y., Sasaki, A., Hashiya, K., Ide, S., Bando, T. *, Maeshima, K. *, and Sugiyama, H.**co-corresponding authors
Chemical Science, January 20, 2015. DOI: 10.1039/C4SC03755C
ピロール・イミダゾール(PI)ポリアミド化合物は、DNAの二重らせんの副溝を通して、塩基配列を特異的に認識することができます。この方法は、これまで行われていた、FISHなどの塩基対を認識させる方法に比べ、迅速、簡便、そしてクロマチン構造が維持できるような温和な条件で塩基対認識を行える利点を持ちます。これまでに私たちは、哺乳類のテロメアDNA [(TTAGGG)n]を認識するPIポリアミド(TH59)の合成(Maeshima et al., EMBO J. 2001)および、その大量合成法を開発してきました (Kawamoto et al., JACS. 2013)。
今回、従来のTH59を改良し、テロメア配列への結合特異性を上げることに成功しました。TH59はヘアピン構造が二つヒンジを介して連結し、テロメアリピートに結合します。本論文では、ヒンジの長さを至適化したポリアミド (HPTH59-b, 図A)、ヘアピン構造を三つ連結させ、より長い18bpテロメアリピートを認識するポリアミド (TT59, 図B)を合成しました。これらの改良により、ポリアミドが非特異的配列に結合する頻度を減少させ、テロメア領域以外からのシグナルを飛躍的に減少させることができました。今後、簡便かつ高精度なテロメア長の定量法として、基礎研究のみならず臨床分野において広く用いられることが期待されます。
本研究は、京都大・理学研究科・杉山教授グループおよび株式会社ハイペップ研究所 (京都府)との共同研究です。遺伝研・共同研究Bのサポートを得ておこなわれました。
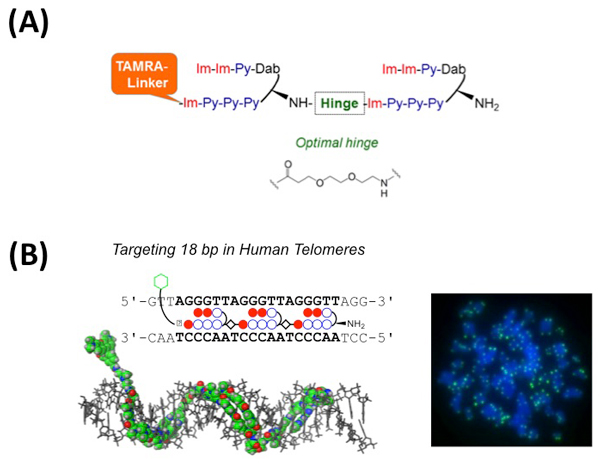
(A)ヒンジが改良されたHPTH59-bの模式図。改良型ヒンジ領域の化学構造が示されている。
(B)左、ヘアピン構造を三つ連結させたTT59ポリアミドの模式図、および結合様式。右、染色体スプレッドをTT59で標識した画像。青色 (DNA染色)の染色体末端が緑色のドット状に標識されている。
太田朋子名誉教授がクラフォード賞を受賞
スウェーデン王立科学アカデミーは、2015年のクラフォード賞を太田朋子名誉教授とリチャード・レウォティン米ハーバード大学名誉教授へ贈呈することを発表しました。今回の賞は、両教授による遺伝的変異および進化への理解を促す先駆的研究に対して贈られます。授賞式は5月5〜7日にスウェーデンのストックホルムで行われます。 クラフォード賞は、天文学、数学、生物科学、関節炎の各分野の研究に対して年ごとに順次与えられるものであり、ノーベル賞が扱わない専門分野を補完しています。
今回の受賞は、太田先生が遺伝研で行った遺伝的多型の理解に関する基礎的貢献が認められたものです。太田先生は1973年、集団内における変異および同種間における違いを理解する重要な要素としての「ほぼ中立説」をNature誌に発表しました。現在では弱い淘汰はゲノム進化を理解する上での中核となる考えであるとされており、太田先生の考え方は生体医学、システム生物学、比較ゲノム解析の研究に大きな影響を与えています。
2015年度国立遺伝学研究所「共同研究」「研究会」募集について (募集は締め切りました)
転写因子の物理的サイズはクロマチンドメイン内の転写制御にとって重要である
The physical size of transcription factors is key to transcriptional regulation in chromatin domains
Kazuhiro Maeshima, Kazunari Kaizu, Sachiko Tamura, Tadasu Nozaki, Tetsuro Kokubo, and Koichi TakahashiJournal of Physics: Condensed Matter, 27, 064116 (10 pp), 2015. DOI: 10.1088/0953-8984/27/6/064116
真核生物には、大小様々な大きさの転写因子があります。そして、これらの転写因子は、微小な空間に折り畳まれている長いDNAから、必要な遺伝情報を読み出すのに必要です。一般的に、遺伝子特異的な働きをする転写因子のサイズは小さく (~50 kDa)、これらはゲノムDNA中の標的となる調節領域を探して結合します。そして、この小さい転写因子が、基本転写因子、メディエーター、RNAポリメラーゼ、ヌクレオソームリモデラー、ヒストン修飾因子など、巨大タンパク複合体 (1-3 MDa以上) の標的配列への結合を誘導すると考えられています。最近、細胞核内では、ヌクレオソーム線維が凝集することによってできるクロマチンドメイン(topologically associating domains, 図の青ボール集団) が多数形成されていることが分かってきました。私たちは、この凝集したクロマチンドメインにおける転写制御に、転写因子やその複合体の物理的なサイズが重要であるという新しいモデルを提唱しました。
私たちは、まずモンテカルロ・シミュレーションによって、凝集したクロマチンドメイン内部に入ることができる転写因子の物理的なサイズを決定しました。その結果、遺伝子特異的な転写因子群(小さいサイズ)は、ドメイン内部に入り込むことができる一方(図の黄ボール)、サイズが大きな転写複合体群は、ドメイン内部に入ることはできないことが分かりました(図の緑ボール)。
この結果、サイズの小さな転写因子がクロマチンドメイン内の標的配列を探索して結合し(図a)、その転写因子-標的配列複合体がドメイン表面に出てきた際、この転写因子を「目印」として、サイズの大きい転写因子複合体が標的配列に結合すると考えました (図b)。標的配列に結合した巨大な転写因子複合体は、ドメイン内部へと動くことが難しいため、標的配列をドメインの表面に維持する「ブイ(浮き袋)」のような働きができます(図b)。そして、ヌクレオソームリモデラー、ヒストン修飾因子など、他の巨大複合体と共に安定的な転写を可能にするという新しいモデルを提唱しました (図c)。
これらの転写因子の物理的サイズに依存した、標的探索の方法や、標的配列をドメイン表面につなぎ止める性質は、DNA複製や修復、組換えを含む様々なDNA機能に共通するメカニズムと考えられます。
本研究は理研・生命システム研究センター高橋恒一・海津一成、横浜市立大学・古久保哲朗らとの共同研究としておこなわれ、英国Journal of Physics: Condensed matter誌の「Physics of chromatin」特集号に掲載されました。
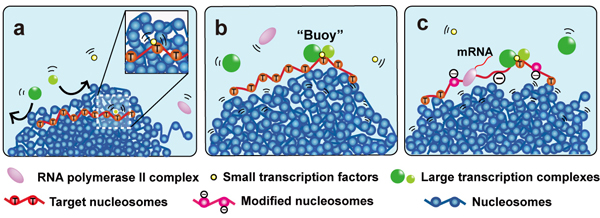
転写制御における「ブイ(浮き袋)」モデル
凝集したクロマチンドメインは青いボール集団で示している。
(a) 黄色い小さな転写因子は凝集したクロマチンドメイン内部を動くことができるが、緑の大きい転写因子複合体はドメインに入ることができない。 (b) 大きな転写因子複合体がブイ(浮き袋)となって、標的配列がドメイン表面で維持される。(c) ヌクレオソームリモデラー、ヒストン修飾因子など、他の巨大複合体と共に、安定的な転写がおこる。
1分子のタンパク質でDNAの2本鎖をほどく新しい仕組み
超分子構造研究室・白木原研究室
Structural basis for replication origin unwinding by an initiator-primase of plasmid ColE2-P9: Duplex DNA unwinding by a single protein
Hiroshi Itou, Masaru Yagura, Yasuo Shirakihara, and Tateo ItohJournal of Biological Chemistry, 2015 Feb 6;290(6):3601-3611. DOI: 10.1074/jbc.M114.595645
DNAを複製する際、2本鎖構造が一時的に解かれます。この2本鎖構造を壊すためには、複数のタンパク質が集まって出来る複合体が必要です。1本鎖が露出した部分に、ヘリカーゼ、プライマー合成酵素、DNA合成酵素等が結合し、DNAの複製が始まります。大腸菌を宿主とするColE2プラスミドの複製開始因子Repは、プラスミド上の複製開始点に特異的に結合し、DNAの2本鎖構造を解き、更にRNAプライマーの合成を行う多機能なタンパク質です。
今回の研究では、Repが持つDNA結合領域と複製開始点との複合体の立体構造を解明しました。RepのDNA結合領域は3つのモジュールで構成され、1分子内の各モジュールが連携して複製開始点へ特異的に結合し、その2本鎖構造を巧みに解く仕組みが明らかになりました。さらに、立体構造および機能が未知であったPriCTと呼ばれる領域が、2本鎖の解裂に重要な役割を果たす事も今回初めて明らかになりました。このドメインは様々なバクテリアを宿主とするプラスミドだけでなく、バクテリアや真核生物のウィルス等でも保存されていることから、今後これらのタンパク質が担う分子機構に対する理解がより深まると期待されます。
本研究は信州大学理学部等との共同研究で行われました。
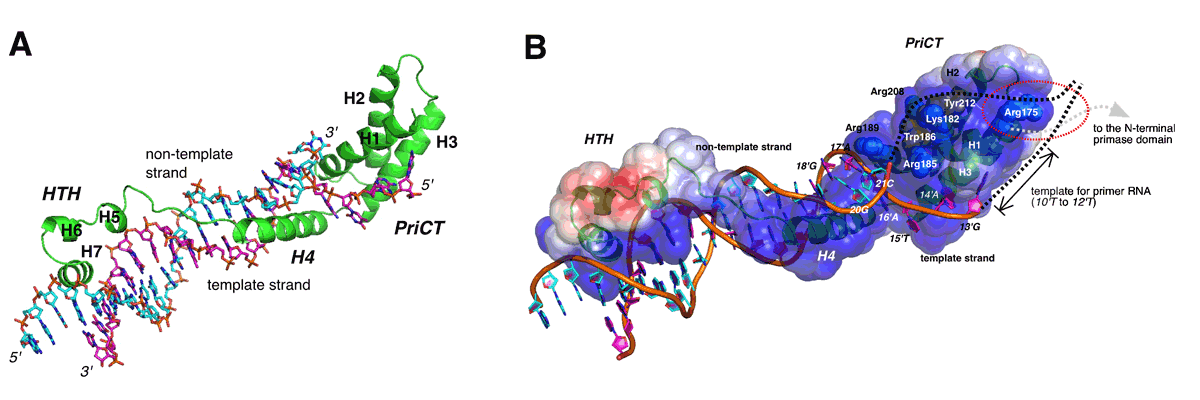
(A) ColE2 RepのDNA結合領域と複製開始点との複合体の立体構造。DNA分子に沿って細長く巻き付くような形を持つ事で、DNAの構造を壊すに足りる結合力を確保している。
(B) 本研究から提案された複製開始点解裂のモデル。 PriCT領域は2本鎖の解裂と解けた構造の安定化に中心的な役割を持つ。















