前島研究室・ゲノムダイナミクス研究室
ヒトゲノムの折り畳み構造とダイナミクス
教員
▶ Lab HP ▶ Lab Pamphlet
Research Summary
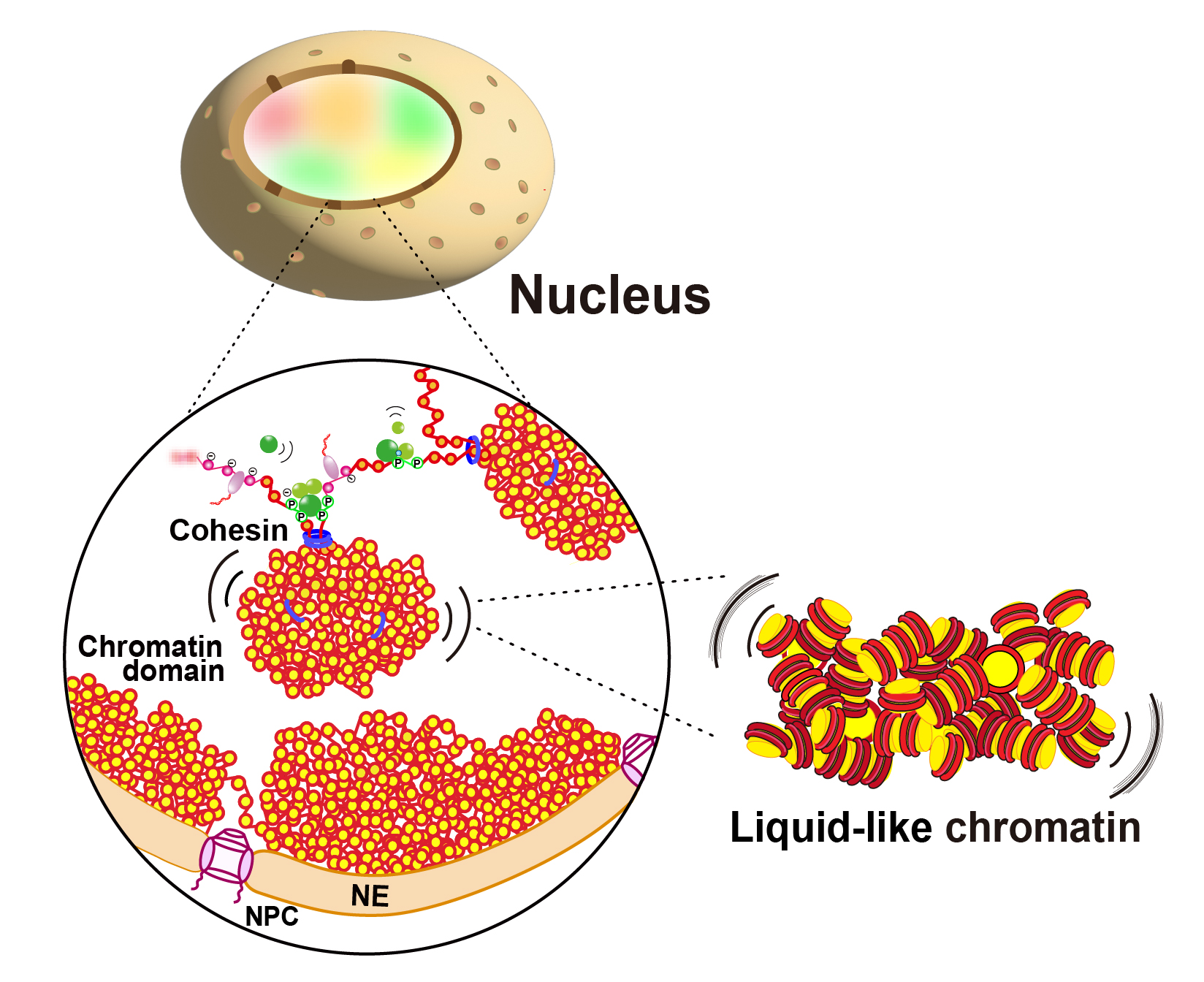
本研究室では、「ヒトゲノムDNAが細胞のなかに、三次元的にどのように折り畳まれ、そしてどのようにヒトゲノムが読み出されるのか?」を研究しています。私たちは、ヒト細胞内のクロマチンがとても不規則な形で柔軟に折り畳まれていることを発見しました。この知見を、遺伝子発現、発生分化、エピジェネティックスなど、幅広い研究につなげていきます。ライブセル1分子イメージング、超解像顕微鏡イメージング、ゲノミックス、シミュレーションなどを組み合わせて、ユニークな研究を目指しています。
研究活動の紹介動画
Selected Publications
Hibino K, Sakai Y, Tamura S, Takagi M, Minami K, Natsume T, Shimazoe MA, Kanemaki MT, Imamoto N, Maeshima K. Single-nucleosome imaging unveils that condensins and nucleosome-nucleosome interactions differentially constrain chromatin to organize mitotic chromosomes. Nat Commun. 2024 Aug 21;15(1):7152.
Iida S, Ide S, Tamura S, Sasai M, Tani T, Goto T, Shribak M, Maeshima K. Orientation-independent-DIC imaging reveals that a transient rise in depletion attraction contributes to mitotic chromosome condensation. Proc Natl Acad Sci U S A. 2024 Sep 3;121(36):e2403153121.
Maeshima K, Iida S, Shimazoe MA, Tamura S, Ide S. Is euchromatin really open in the cell? Trends Cell Biol. 2024 Jan;34(1):7-17.
Nozaki T, Shinkai S, Ide S, Higashi K, Tamura S, Shimazoe MA, Nakagawa M, Suzuki Y, Okada Y, Sasai M, Onami S, Kurokawa K, Iida S, Maeshima K. Condensed but liquid-like domain organization of active chromatin regions in living human cells. Sci Adv. 2023 Apr 5;9(14):eadf1488.
















