Archive
- Home
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
日本列島で生じたトゲウオの種分化の様子を全ゲノム配列から解明
The genomic landscape at a late stage of stickleback speciation: High genomic divergence interspersed by small localized regions of introgression
Ravinet, M., Yoshida, K., Shigenobu, S., Toyoda, A., Fujiyama, A., and Kitano, J.
Plos Genetics Published: May 23, 2018 DOI:10.1371/journal.pgen.1007358
種とは、お互いに交配して一つに混合してしまわないような集合体と定義されます(生物学的種概念による)。もともとは連続的であった集団から、どのようにして異なる種が生じるのか(種分化)は、進化生物学における重要な課題です。
遺伝子流動(2集団の間で遺伝子が交流すること)がある場合、種分化の初期の段階では、ゲノム(個体に存在するDNAの集合体)全体ではなく、限られた一部の配列のみが分化していくことが、近年のゲノム解析によって多くの分類群で示されてきました。その一方で、種分化の後期にどのようにして種分化が完成していくかについては多くが不明でした。特に、地理的に完全に分断されるなどの大きな出来事がない場合にどのように種形成が完成に向かうのかは大きな謎でした。
本研究では、日本に生息するトゲウオ2種(図参照)、太平洋岸を中心に生息するイトヨ(Gasterosteus aculeatus)と日本海を中心に生息するニホンイトヨ(G. nipponicus)について全ゲノム配列を利用してこの課題に取り組みました。この2種は、北海道の道東地域などでは、繁殖地を共有しており、頻度は低いものの交雑を続けていることから、種分化の後期に位置すると考えれています。我々はニホンイトヨの全ゲノム配列を既に決定の上、報告しています(Yoshida et al. 2014 PLoS Genetics 10: e1004223)。本研究では、まず、この2種が約68万年前に分岐を始めたこと、分岐後も程度は低いものの遺伝子の交流をほぼ連続的に続けながら分化してきたことを明らかにしました。次に、2種間のゲノム配列を比較すると、殆どのゲノム領域では分化が進行しているにも関わらず、一部のゲノム領域では遺伝子配列が似通っており(図参照)、このような分化の低い領域は遺伝子流動によることが明らかになりました。遺伝子流動は、組換え率の低い領域、特にネオX染色体(ニホンイトヨのみに存在するX染色体)で低いことも明らかになりました。遺伝子流動は、免疫関連遺伝子に多く、何らかの機能的な意義があるのではないかと推測されましたが、機能的意義に関してはさらに詳しい研究が必要です。
遺伝子流動を保持しながら種分化が後半まで到達した事例において、種間でのゲノム分化のパターンが解析された研究例が少ないことから、貴重な成果と言えます。今後、類似の研究が他の分類群で実施されることによって、本成果の普遍性が検証されることが望まれます。
本研究は、学術振興会の助成を得て生態学研究部門・北野研究室(https://www.nig.ac.jp/labs/EcoGene/)に滞在中のMark Ravinet博士(現・オスロ大学)が中心となって、国立遺伝学研究所・比較ゲノム解析研究室、基礎生物学研究所・生物機能情報分析室との共同研究として実施したものです。科研費(23113007, 23113001, 221S0002, 15H02418)及び、国立遺伝学研究所共同利用-I (98I2017)などの支援を受けました。
本成果は、PLoS Geneticsに掲載されました。
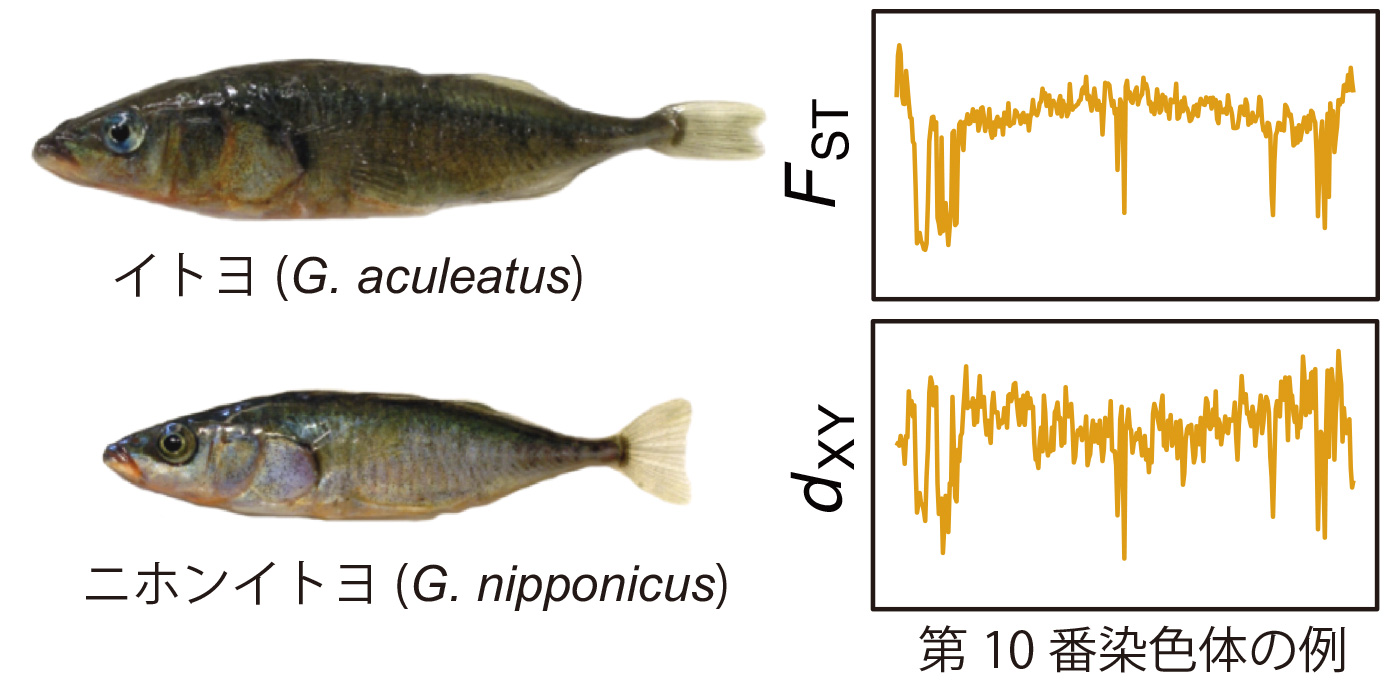
図:左は、イトヨとニホンイトヨのオス。右は、ある染色体(ここでは第10番染色体)に観察されたゲノムの分化のパターン。
第9回国際トゲウオ会議[Stickleback2018]を開催:7月3日-7日
2018年7月3日から7月7日にかけて,京都大学吉田キャンパスにて第9回・国際トゲウオ会議 Stickleback2018 (https://sites.google.com/view/stickleback2018/)を開催します。
本国際会議は、国際ネットワーク形成・MOU推進プロジェクトの一環として、情報・システム研究機構(ROIS)の支援を受けて開催します。他にアメリカ遺伝学会、福井県大野市、京都大学教育研究振興財団の支援を受けています。
プレナリー講演会を公開と致しますので、興味のある方は是非お越しください。いずれの講演者も、進化生態学、進化遺伝学、進化発生学の分野で大きな成果を挙げられている方々です。
会場: 京都大学吉田キャンパス 北部構内 北部総合教育研究棟 益川ホール
http://www.kyoto-u.ac.jp/ja/access/campus/yoshida/map6r_n.html
7/4(水)9:30-10:15
David Kingsley (HHMI and Stanford University School of Medicine)
The arrival of the fittest: sources of molecular variation underlying repeated evolution in vertebrates
7/5(木)9:30-10:15
Dolph Schluter (University of British Columbia)
Fitness maps to a large-effect locus in an experimental stickleback population
7/6(金)9:30-10:15
Andrew Hendry (McGill University)
Eco-evolutionary dynamics
7/7(土)9:30-10:15
Ole Seehausen (Eawag and University of Bern)
The genomic basis of quantum speciation in Lake Victoria cichlids
「夏休み子ども遺伝学講座」を開催
スマホ顕微鏡で見る、遺伝学で活躍する小さな生き物
対象:
三島市内の小学校4年生~6年生 30名
(申込者多数の場合は抽選)
日時:
平成30年7月31日(火)
午前9時30分~午前12時00分(予定)
場所:
国立遺伝学研究所(三島市谷田1111)本館3FA316
持ち物:
筆記用具(※使用する端末、顕微鏡は主催で用意)
申込:
三島市政策企画課まで電話か電子メールでお申込みください。
申込時に ①氏名 ②学年 ③住所 ④電話番号 をお伝えください。
申込み締切は平成30年 7月17日(火)です。
E-mail: seisaku@city.mishima.shizuoka.jp
環境と微生物をビッグデータでつなぐ ~「環境」から微生物を検索し、微生物から「環境」を予測するウェブツール「LEA」を開発~
![]()
Latent environment allocation of microbial community data
Koichi Higashi, Shinya Suzuki, Shin Kurosawa, Hiroshi Mori and Ken Kurokawa
PLOS Computational Biology Published: June 6, 2018 DOI:10.1371/journal.pcbi.1006143
微生物は様々な環境に存在し、物質循環や宿主の健康などに影響しています。したがって「どのような微生物がどのような環境に生息するのか?」という問いは、環境問題や健康問題の解決の糸口となり得るので世界中で探求されました。その結果、特定の「環境」に生息する微生物の群集についてのデータが大量に蓄積される一方で、その「環境」を弁別する記述が研究者間でまちまちとなっていました。この問題によって、研究者間のデータを横断的に解析することが困難となり、微生物の群集と相互作用する環境因子を包括的に理解することができませんでした。
情報・システム研究機構 国立遺伝学研究所の東光一特任研究員および黒川顕教授らのグループは、微生物群集構造の大規模データから様々な環境と微生物とのつながりを明らかにし、その結果を利用して環境と微生物のつながりを可視化するウェブツール「LEA」(http://leamicrobe.jp)を開発しました。
本研究では、数万サンプルの微生物群集データと各サンプルの由来環境について研究者が記述した文書(自然言語)を確率モデルで表現し、微生物群集の形成に影響を及ぼす環境を機械学習技術によって抽出しました。抽出された「環境」はそれぞれ固有の「微生物」群集構造と結びついています。この結果をもとに、「環境」に対応する「微生物」を検索する、あるいは逆に「微生物」から「環境」を予測するウェブツール「LEA」を開発しました。LEAは、ヒト共生微生物による疾患の診断や、環境汚染の検出などへの応用が期待できます。
本研究成果は、「PLOS Computational Biology」に平成30年6月6日午後2時(米国東部標準時間)に掲載されました。
本研究は、文部科学省科研費「新学術領域研究『学術研究支援基盤形成』」先進ゲノム解析研究推進プラットフォームの助成を受けて行われました。
本研究成果は、国立研究開発法人科学技術振興機構バイオサイエンスデータベースセンター(JST-NBDC)のライフサイエンスデータベース統合推進事業(統合化推進プログラム)における研究開発課題「データサイエンスを加速させる微生物統合データベースの高度実用化開発」の一環として開発されました。
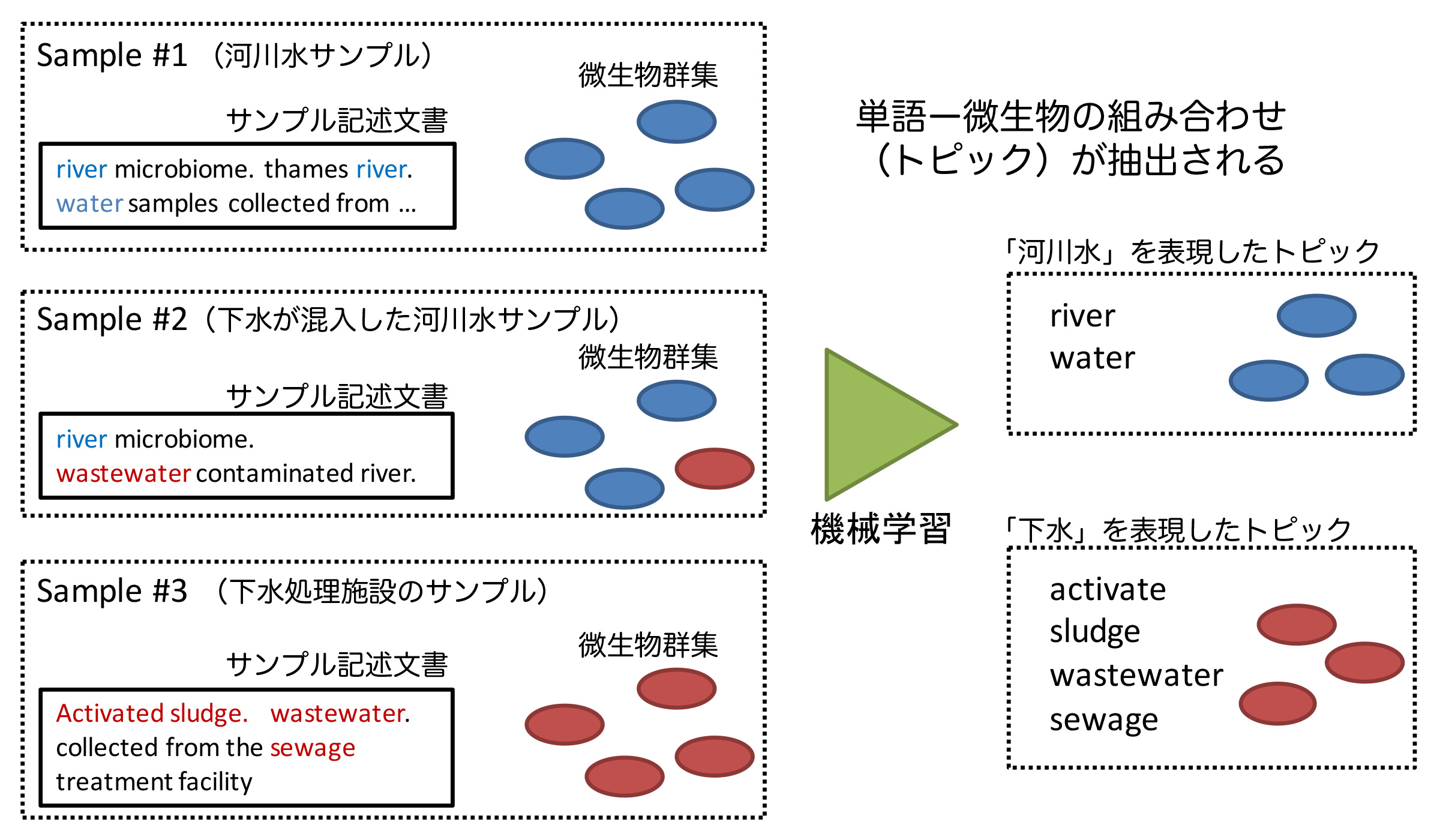
図1: 微生物と自然言語の対応関係を学習
DDBJなどの公共データベース中のすべてのメタゲノムデータには、「サンプルについて研究者が記述した文書(自然言語)」が付随する。
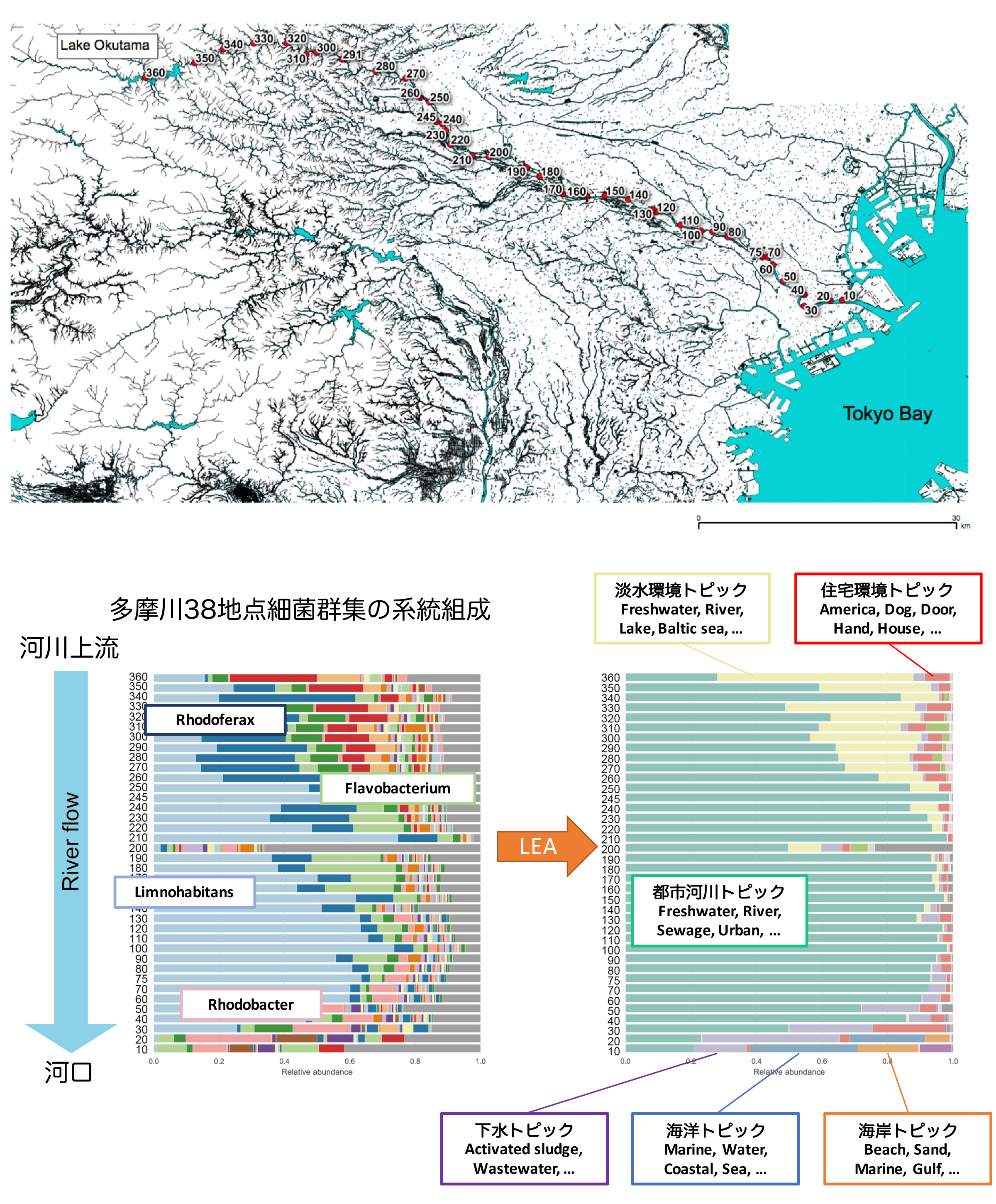
図2:微生物群集構造データからの環境予測
テスト対象:多摩川細菌群集
上流(奥多摩湖)から河口(羽田空港付近)まで38サンプルを解析
国立台湾大学・生命科学院と学術交流協定を締結
今後、共同研究などによって益々の組織間交流が期待されます。
 |
 |
| 国立台湾大学・生命科学院 (JENG, Shih-Tong 学部長とTING, Chau-Ti 助教授) |
国立遺伝学研究所(桂勲 所長と川上浩一 教授) |
卵の品質はDazl遺伝子の適切なONとOFFで制御される
Requirement of the 3′-UTR-dependent suppression of DAZL in oocytes for pre-implantation mouse development
Kurumi Fukuda, Aki Masuda, Takuma Naka, Atsushi Suzuki, Yuzuru Kato, and Yumiko Saga
Plos Genetics Published: June 8, 2018 DOI:10.1371/journal.pgen.1007436
卵子形成は様々な遺伝子の適切な発現によって進行します。マウスの卵子形成に重要な遺伝子の一つ、Dazlはノックアウトすると不妊になり、胎児期も生後もRNAの発現があること等から、卵子形成過程を通じて必要な遺伝子と考えられてきました。
本研究ではこれまでの通説に反し、胎児期の雌性生殖細胞の発生にDazlは必須であるが、生後の卵胞形成においては、むしろDazlは抑制されることが受精後の胚発生の正常な進行に必要であることを明らかにしました。Dazlの発現抑制が破綻すると一部の受精卵は胚発生を停止し、その結果、産子数が減少すること、また、この抑制は3’UTRを介した翻訳抑制によって行われることが新たに分かりました。以上の結果は、卵母細胞におけるDazlの必要性について再考を迫るものです。
本研究は情報・システム研究機構国立遺伝学研究所の福田胡桃(学振特別研究員)、加藤譲(助教)、相賀裕美子(教授)と、横浜国立大学の鈴木敦(准教授)らにより、以下のサポートを受けて行われました。
日本学術振興会(16J11687(福田胡桃)、25840091(加藤譲)、26251025(相賀裕美子))
文部科学省新学術領域(16H01252(鈴木敦)、16H01259(加藤譲)、25112002(相賀裕美子))
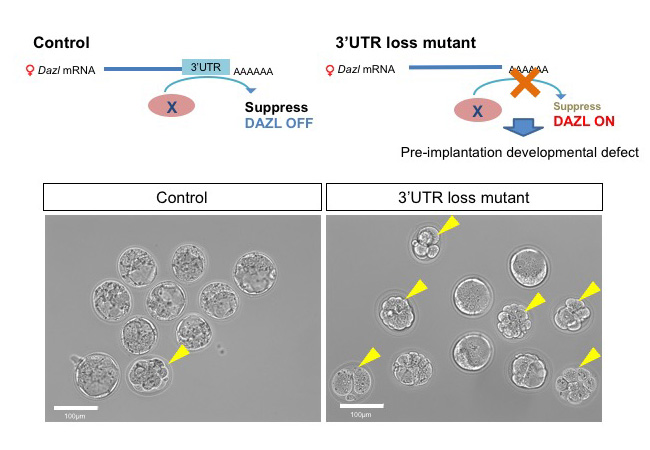
図:通常、生後の卵子形成過程においてDAZL蛋白質の発現は3’UTRを介して翻訳抑制される(図左上)。一方、Dazlの3’UTRが欠損した卵子では生後もDAZLが発現し続け、一部の胚発生が停止する(図右上)。コントロールでは着床後3.5日目で殆どの胚がブラストシストになっているが(図左下)、変異体では約半数が途中で発生を停止している(図右下)。黄色の矢印は発生が停止した異常胚。
高速かつ高精度に細菌群集の系統組成を推定するツールVITCOMIC2を開発
VITCOMIC2: visualization tool for the phylogenetic composition of microbial communities based on 16S rRNA gene amplicons and metagenomic shotgun sequencing.
Mori H, Maruyama T, Yano M, Yamada T, Kurokawa K. BMC Syst Biol
BMC Systems Biology 12 (Suppl 2), 30, 2018. DOI:10.1186/s12918-018-0545-2
情報・システム研究機構国立遺伝学研究所 生命情報研究センター ゲノム進化研究室 森宙史助教および黒川顕教授らは、細菌群集由来のDNAを丸ごとシーケンスすることで、個々の細菌を培養することなく解析可能な手法であるメタゲノム解析、および16S rRNA遺伝子のみをPCR増幅してシーケンスするメタ16S解析により得られる膨大なDNA配列データから、高速かつ高精度に細菌群集の系統組成を推定するウェブアプリケーション、VITCOMIC2を開発し公開しました。VITCOMIC2では、DNA配列間の配列類似性をGraphics Processing Unit (GPU)を用いて高速に検索するツールCLASTを用いることで、各配列の高速な系統推定を実現しています。また、VITCOMIC2ではメタ16S解析等で一般的に用いられている配列クラスタリングを行わずに、全配列を配列類似性検索することによって、配列をクラスタリングで生じる系統推定結果の曖昧性を回避でき、高精度な系統推定を実現しています。VITCOMIC2は、http://vitcomic.orgから利用できます。
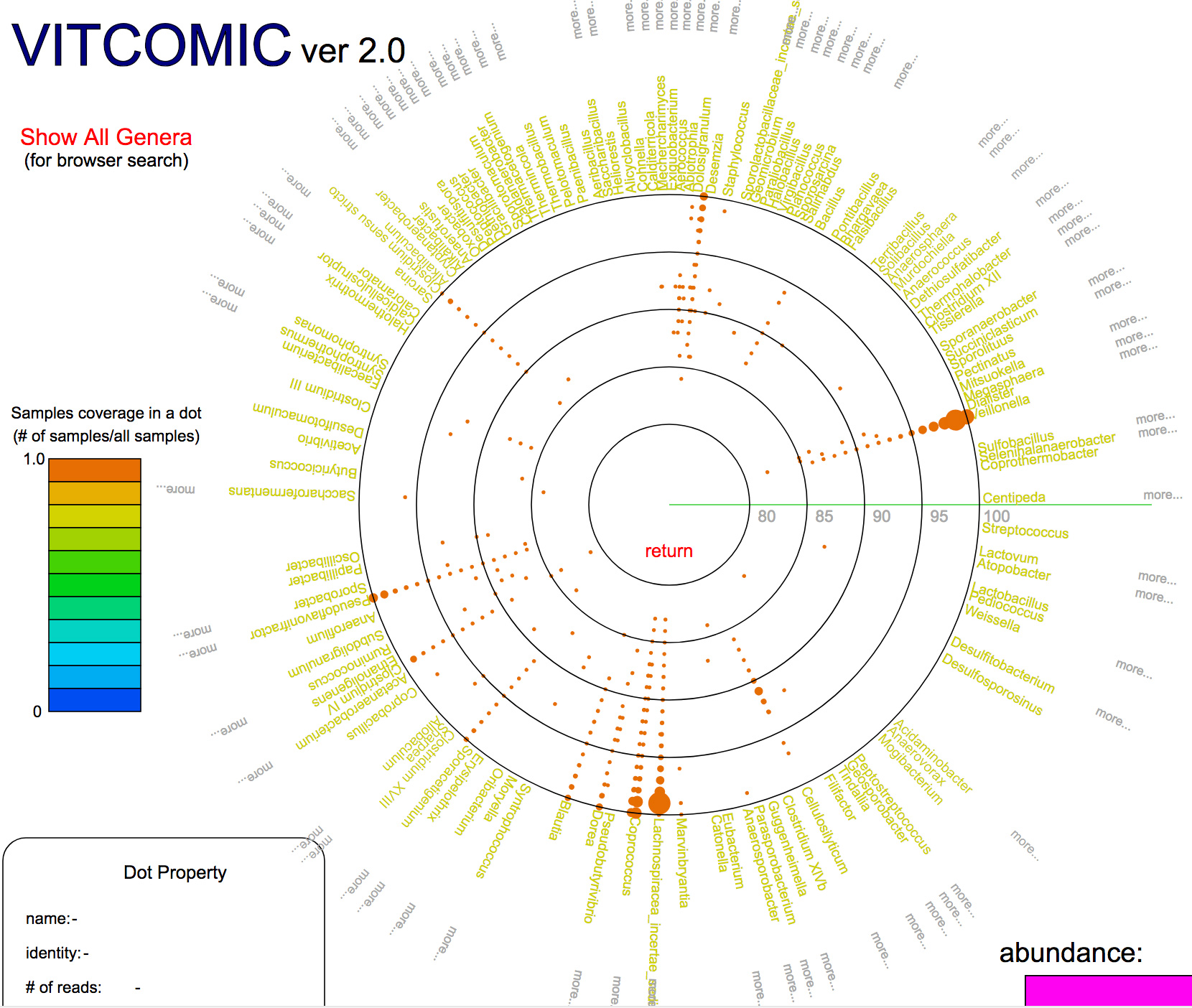
図:VITCOMIC2の結果の表示の例
海水のメタゲノム解析をテーマとする「臨海ハッカソン」を開催:6月10-13日
RinkaiHackathon 2018 受講生募集のお知らせ
臨海実験所を主たる活動の場とし、動植物学と情報学の垣根を超えた研究教育と人材交流を進めることを目的として、数名の有志による研究会”RinkaiHack”を企画しています。
https://sites.google.com/view/rinkaihack/
第2回の公開イベントを、平成30年6月10-13日に広島で開催します。6月10日の午後に福山城内の福寿園にてシンポジウムを、翌11日から3日間を広島大学 向島臨海実験所にて海洋メタゲノムに関するハンズオンセミナーを、それぞれ開講します。
本年は環境DNA/メタゲノムをテーマに、臨海実験所で海水を採集し、DNA抽出から配列解読・解析を行うための講義と実習を計画しています。最近、しばしば話題になる海水のメタゲノム解析の実情を専門家に学び(前日シンポジウム)、3日間の実習でそのエッセンスを実際に一緒に体験しましょう。これらシーケンス解析実習を行うと同時に、実習期間中には、技術の進展によっては変わらない古典的な生物学の知識や思考体系とは何であるかを参加者で議論します。
また、今年から、イベント中の基本言語を英語に変更します。これは、国内の留学生にも積極的に参加してもらいたいと考えたこと、また同時に学外での交流の機会に利用して欲しいと考えたからです。同時に、英語に自信のある日本人学生はもちろん、英語に自信のない学生さんにも参加してほしいと思います。もし英語でのコミュニケーションに自身のない方にも運営スタッフがサポートいたします。
実習の詳細についてはWebsite(https://sites.google.com/view/rinkaihack/registration)を参照してください。また、参加登録、実習に関連する質問等も、Website(Home => Contact タブ)から受け付けています。
実習内容、スケジュール:
ホームページを参照してください。募集締切: 2018年5月11日
* 実習施設・実習内容から定員を20名とし、応募多数の場合は選考を行います。* Hands-onのスケジュールや講演者の日程には変更の可能性があります
招待講師:
川口慎介 かわぐち しんすけ (JAMSTEC) 6月10日(日)ブランマチュー ローマン (京都大学) 6月10日(日)
山本哲史 (京都大学) 6月11日(月)
内山雄介 (神戸大学) 6月12日(火)
ライマー ジェームス (琉球大学) 講演日未定
平岡聡史 ひらおか さとし (JAMSTEC) 6月10日(日)
西村陽介 にしむら ようすけ (東京大学 大気海洋研究所)
Rinkaihackathon2018 運営委員
吉田真明(島根大学隠岐臨海実験所)
川島武士 (国立遺伝学研究所)
濱田麻友子(岡山大学牛窓臨海実験所)
宮本教生 (JAMSTEC)
守野孔明 (筑波大学)
スティアマルガ デフィン (和歌山工専)
大森紹仁 (新潟大学)
田川訓史 (広島大学 向島臨海実験所)
共催
広島大学
DDBJ: DNA Data Bank of Japan
JSBi (日本バイオインフォマティクス学会)
協力
ROIS 未来投資FS
推薦
全国国立大学臨海・臨湖実験所所長会議・議長推薦
















