Archive
- Home
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
【調達情報】入札説明会(官報号外政府調達第73号掲載)について
細胞画像のわずかな特徴の違いの見分け方を教えてくれるAIの開発に成功
Press release
Robust Classification of Cell Cycle Phase and Biological Feature Extraction by Image-Based Deep Learning
Yukiko Nagao, Mika Sakamoto, Takumi Chinen, Yasushi Okada, Daisuke Takao
Molecular Biology of the Cell 22 Apr 2020 DOI:10.1091/mbc.e20-03-0187
東京大学大学院医学系研究科の高尾大輔 助教と東京大学薬学部の長尾幸子 学部生(研究当時)の研究チームは、国立遺伝学研究所の坂本美佳 特任研究員、東京大学大学院薬学系研究科の知念拓実 助教、東京大学国際高等研究所ニューロインテリジェンス国際研究機構(WPI-IRCN)の岡田康志 主任研究者(東京大学大学院医学系研究科・大学院理学系研究科 教授、理化学研究所生命機能科学研究センター(BDR)チームリーダー)らと共同で、ディープラーニングと定量解析により細胞画像から未知の情報を抽出する技術の開発に成功しました。大量の細胞画像から一つ一つの細胞を自動的に切り出し、ディープラーニングにより細胞周期などを判定するAIツールと、その情報をさらに解析することで、細胞周期によって変動する核の形状やゴルジ体の配置パターンなどを抽出する技術を確立しました。これは、人間の目ではとらえにくいわずかな細胞内の変動をAIが発見し、研究者に教えてくれる技術です。
細胞周期によって細胞内の組成や構造は大きく変わることが予想されますが、実際に何がどのように変動するのかを顕微鏡画像から網羅的かつ定量的に解析するためのアプローチは限られていました。具体的に細胞内の何に着目すればいいのかを知るために、今回、共同研究チームはディープラーニングを使って、細胞周期によって変動するパラメータを画像の中から見つける技術を開発しました。これまで研究者が「なんとなく」「経験的に」とらえていた現象や、そもそも見過ごされていた情報を、AIの手助けにより発掘・定量化しようという試みです。その結果、本研究で開発したAIは、DNAやゴルジ体の染色画像の中から、細胞周期によってわずかに変動する特徴を発見しました。この情報を使って共同研究チームが画像を詳しく解析したところ、核やゴルジ体の面積などの特定のパラメータを測定することで画像から細胞周期を分類できることが分かりました。すなわち「画像のどこに着目すれば目的の情報が得られるのかAIが教えてくれる」という研究サポートツールの開発に成功しました。
本研究で開発したツールは汎用性が高く、他の多くの研究への応用を想定しています。細胞周期以外の判定はもちろん、細胞画像以外の画像データへの応用も可能なため、細胞・発生生物学から医療画像の解析など、基礎から応用まで幅広い分野の研究をサポートする強力なツールとなることが期待されます。
本研究は、科学技術振興機構(JST)戦略的創造研究推進事業 チーム型研究(CREST)「統合1細胞解析のための革新的技術基盤」(研究総括:菅野 純夫 東京医科歯科大学 非常勤講師)における「超解像3次元ライブイメージングによるゲノムDNAの構造、エピゲノム状態、転写因子動態の経時的計測と操作」(研究代表者:岡田 康志)および文部科学省世界トップレベル研究拠点プログラム 東京大学国際高等研究所ニューロインテリジェンス国際研究機構(WPI-IRCN)の支援のもとで行われたものです。
また、本研究の一部は以下の事業による支援を受けて行われました。
・文部科学省 科学研究費 新学術領域研究「学術研究支援基盤形成」先端バイオイメージング支援プラットフォーム(ABiS)
・文部科学省 科学研究費 新学術領域研究「情報物理学でひもとく生命の秩序と設計原理」
・日本学術振興会科学研究費基盤研究(C)「分子ダイナミクスの定量・操作と数理モデリングによる中心体複製メカニズムの理解」
・武田科学振興財団
・島津科学振興財団
・コニカミノルタ科学技術振興財団
本研究の成果は、2020年4月22日に米国細胞生物学会誌 Molecular Biology of the Cell に掲載されました。
遺伝研の貢献
AI(人工知能)技術を含む細胞画像解析のための情報と技術サポートを提供しました。
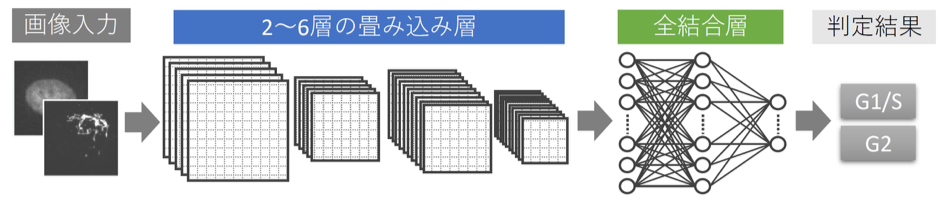
図: AIモデルの模式図
畳み込みニューラルネットワークを使ったディープラーニングモデルを開発した。
新型コロナウイルス感染症に関する遺伝研の対応について【2020年4月21日版】
液体のように振る舞うクロマチン
Fluid-like chromatin: Toward understanding the real chromatin organization present in the cell
Kazuhiro Maeshima, Sachiko Tamura, Jeffrey C. Hansen and Yuji Itoh
Current Opinion in Cell Biology (2020) 64, 77-89. DOI:10.1016/j.ceb.2020.02.016
真核生物のクロマチンはゲノムDNA、ヒストンや他の様々なタンパク質からなる、負に帯電したポリマーです。その電荷に富んだ性質から、クロマチンの構造は、伸びた10-nm線維や折り畳まれた30-nm線維、凝集体/液滴など、周囲の環境に応じて変化します。この10年新たに開発された技術によって、クロマチンは規則正しい構造ではなく、局所的に液体のようにダイナミックで、不規則な構造を持つことがわかってきました。しかし、一つのイメージング (またはゲノミックス) 手法ではクロマチン構造の一側面しか知ることはできません。また私たちは美しいイメージやモデルに感銘を受けがちですが、それらが実際のクロマチンの姿を示していない可能性があります。したがって、細胞内のクロマチンの真の姿を捉えるためには、多くの技術的アプローチに基づいた、幅広い解析を行うことが重要です。この総説論文では、クロマチンに関する現在の知見と、その知見を得るための方法についてクリティカルに論じました。
本研究は、JST CREST (JPMJCR15G2)、JSPS科研費(19K23735, 16H04746, 16H06279 (PAGS), 19H05273)、武田科学振興財団、アメリカ国立科学財団(1814012)、国立遺伝学研究所博士研究員、JSPS特別研究員(PD)の支援を受けました。
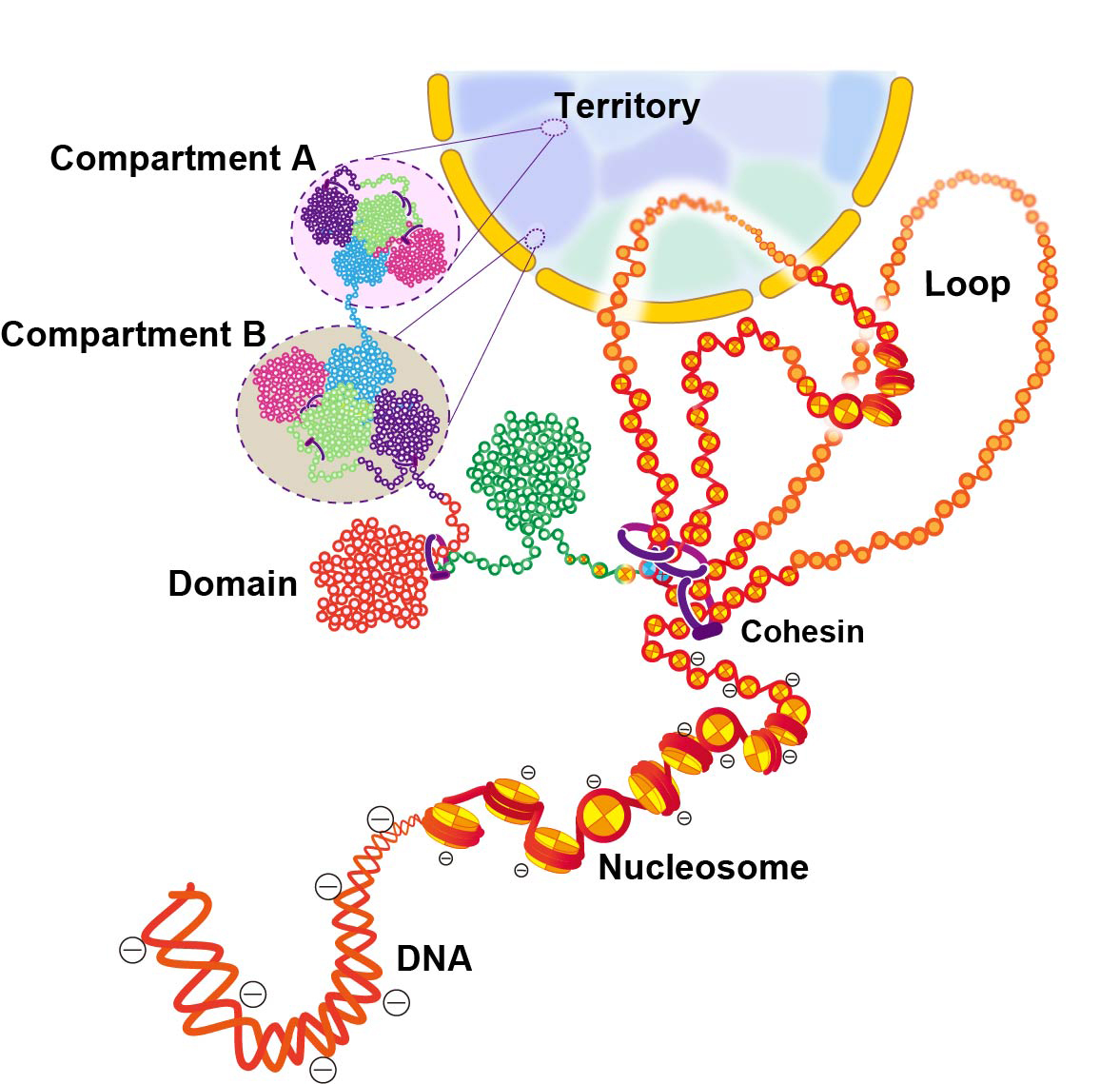
図:細胞核内のクロマチン階層構造の模式図
DNAはヒストン八量体に巻き付き、ヌクレオソームを形成している。10-nmクロマチン線維は、コヒーシンや他のタンパク質によって、ループ構造を形成する。ヌクレオソームの鎖はコンパクトなクロマチンの塊 (ドメイン) を形成し、ドメイン同士が相互作用することでコンパートメントができる。コンパートメントAとBはそれぞれ、転写がアクティブなクロマチンと、転写が不活性なクロマチンを表すと考えられている。間期の染色体は、染色体領域を形成する複数のコンパートメントからなる。この模式図は、現在考えられているモデルを単純化して示したものであり、実際はもっと複雑な階層構造を形成しているとも考えられる。
国立遺伝学研究所の入館制限について
全ての来客を禁止します。来所業者の方には、マスク及び手指のアルコール消毒の徹底と所定の場所以外(各研究棟)への出入りは控えるようにご協力をお願いします。
国立遺伝学研究所長 花岡文雄
お問い合わせ
国立遺伝学研究所 総務・企画係
E-mail: webmaster@nig.ac.jp
絶滅危惧集団に蓄積した有害変異を解明
Accumulation of deleterious mutations in landlocked threespine stickleback populations.
Yoshida, K., Ravinet, M., Makino, T., Toyoda, A., Kokita, T., Mori, S., and Kitano, J. (2020)
Genome Biology and Evolution 31 March 2020 DOI:10.1093/gbe/evaa065
生物の集団サイズが低下すると、有害な変異が集団内に蓄積しやすいことが理論的に予測されています。有害変異がある程度蓄積すると、集団サイズがさらに小さくなって、絶滅への負のスパイラルが生じうると予測されます。
国立遺伝学研究所の太田朋子名誉教授らによって確立された「ほぼ中立説」によると、集団サイズが小さくなると、自然選択の効果(有利な変異が増えたり、不利な変異が減ったりする効果)が弱くなり、弱有害な変異が中立な変異(有利でも不利でもない変異)とほぼ同じ確率で固定することが予測されています。この知見は、絶滅危惧種の保全を考える上できわめて重要な視点です。
現在のゲノム解読技術の進展によって、全ゲノム配列情報から有害変異の存在を間接的に推定することが可能になりつつあります。例えば、多くの分類群で共通している(進化的に保存されている)アミノ酸に生じた変異の有害性を推定する統計的手法が確立されています。実際に、栽培植物や家畜など人為的に作られた生物の小集団では、有害変異が蓄積していることが全ゲノム解析によって既に確認されています。しかし、野外の絶滅危惧集団での検証は殆ど行われていません。
そこで、生態遺伝学研究室の吉田恒太研究員(現・マックスプランク研究所)と北野潤教授を中心とする研究チームは、集団サイズが著しく低下している本州のトゲウオ集団について、全ゲノム配列情報から有害変異の推定に挑みました。トゲウオ科の魚は冷水性で、本州では湧水域などの限られた水域のみで生息が可能です。しかしながら、わずか数十年で、湧水の枯渇や生息域の埋め立てなどによって、本州の陸封トゲウオ集団は絶滅の危機に瀕しています。
本研究の結果、実際に、本州の陸封トゲウオ集団の多くは、海に生息するトゲウオ集団よりも明らかに有害変異が蓄積していることが確認されました(図)。これらの集団については、集団サイズの増加などの保全策を講じる必要性が示唆されました。このように、全ゲノム解析が絶滅危機種や集団のリスク推定といった保全生物学に貢献できることが示されました。本成果は、Genome Biology and Evolutionにオンライン公開されました。
本研究は、国立遺伝学研究所の比較ゲノム解析研究室、東北大学生命科学研究科、岐阜協立大学、福井県立大学との共同研究として、科研費(JP23113007, JP23113001, 221S0002, 15H02418, 19H01003)などの支援を得て行われました。
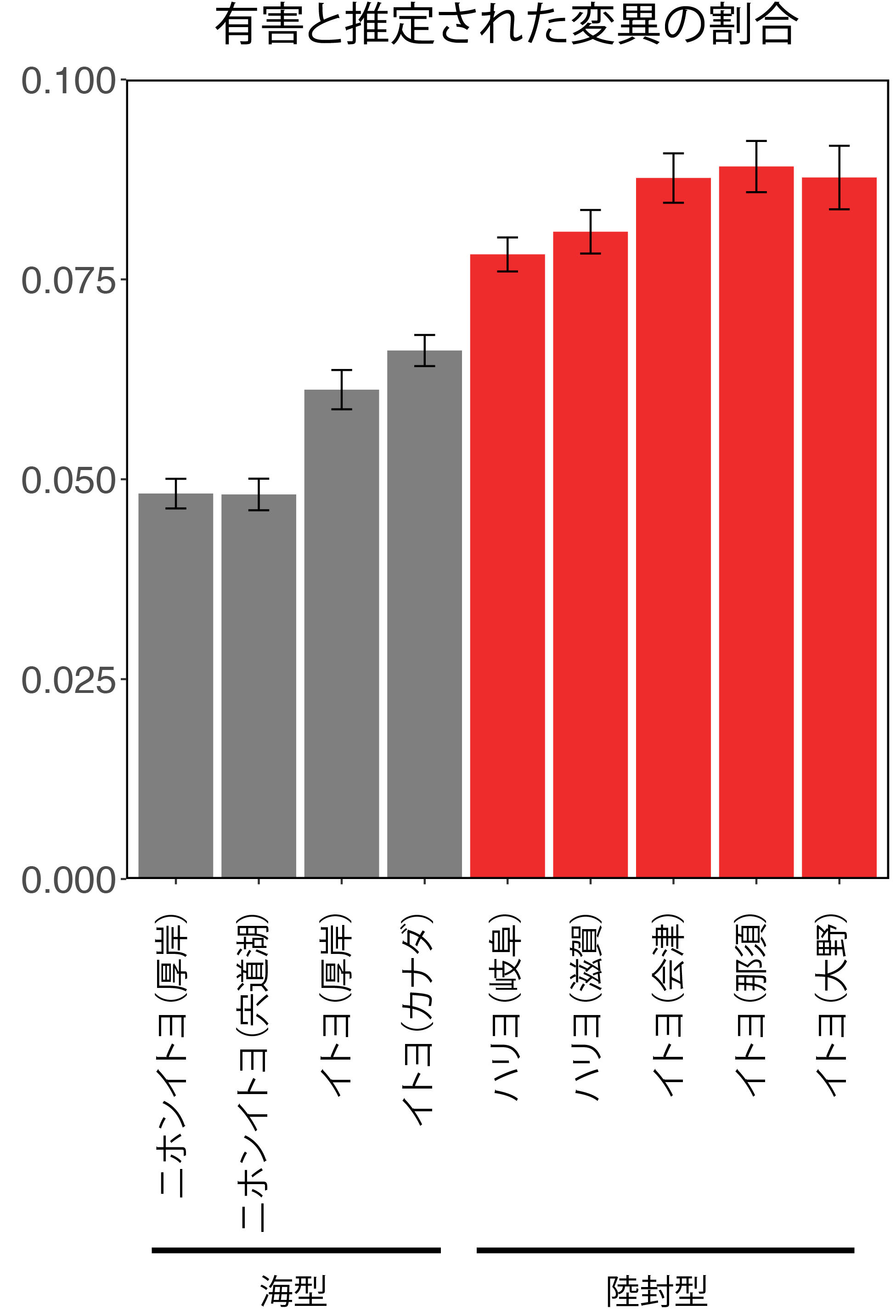
図:陸封集団では、海の集団に比して、有害変異と推定される変異が多く見つかりました。

写真:岐阜県に生息するイトヨ属の陸封化した集団(撮影:秦康之)
生態遺伝学研究室 北野潤教授が第28回木原記念財団学術賞を受賞

生態遺伝学研究室 北野潤教授の第28回(令和元年度)木原記念財団学術賞の受賞が決定しました。
木原記念財団学術賞は、50歳以下の研究者を対象とし、生命科学の分野で優れた独創的研究を行なっている研究者を励まし、顕彰し、今後の研究発展の一助とするために、木原記念横浜生命科学振興財団より贈呈される賞です。
北野教授の「野生動物の表現型多様化の原因遺伝子の解明」の研究が評価されました。















