Archive
- Home
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
日本メダカ雌雄差の地理的変異の遺伝基盤
Ontogenetic stage-specific quantitative trait loci contribute to divergence in developmental trajectories of sexually dimorphic fins between medaka populations
Kawajiri, M., Yoshida, K., Fujimoto, S., Mokodongan, D., Ravinet, M., Kirkpatrick, M., Yamahira, K., and *Kitano, J. Molecular Ecology in press DOI:10.1111/mec.12933雄雌で繁殖に有利な表現型が異なる場合、性差が進化することがあります。雌雄差は集団間によってその程度は大きく異なっていますが、その遺伝基盤はよく分かっていません。このたび、国立遺伝学研究室の北野潤研究室と琉球大学の山平寿智研究室の共同研究により、日本の野生メダカを用いてその遺伝基盤に迫る研究がMolecular Ecology誌にオンライン掲載されました(http://onlinelibrary.wiley.com/doi/10.1111/mec.12933/abstract)。
メダカは、オスの尻ビレがメスよりも発達していることはよく知られており、交配時にオスがメスをつかむために進化したと考えられています。山平研究室では沖縄のメダカの方が、青森のメダカよりも尻ビレの性差が顕著であることを見いだしていました。そこで北野研究室では、性差の度合いの違いを生み出す原因遺伝子座をマッピングし、染色体14番に強い効果のある遺伝子座を同定しました。この際に、テキサス大学のカークパトリック博士と共同で直交ルジャンドル多項式を用いた成長曲線の遺伝解析方法を確立しました。今後は、この遺伝子の実体に迫ることで、野外の生物集団における雌雄差の変異の進化遺伝機構に迫ります。
この研究の一部は、国立遺伝学研究所と琉球大学熱帯生物圏研究センターの共同利用研究の支援のもと行われました。

オス(上)の方が、メス(下)よりも長い尻ビレを持つ
オーキシンデグロン法を利用した簡便な出芽酵母コンディショナル変異体作製法
Rapid Depletion of Budding Yeast Proteins via the Fusion of an Auxin-Inducible Degron (AID)
Kohei Nishimura and Masato T. Kanemaki Current Protocols in Cell Biology, 64, 20.9.1-20.9.16, 2014; DOI:10.1002/0471143030.cb2009s64様々な遺伝学的解析法が確立している出芽酵母は真核生物のモデル生物として長い間重要な役割を果たしてきました。出芽酵母研究においては、コンディショナル変異株として、一般的に温度感受性変異株が作製されてきました。しかしながら、温度変化により標的タンパク質を不活化する特性上、細胞に熱ショック応答が起きること、時としてタンパク質を不活化に時間がかかるなどの問題点がありました。 そこで私たちの研究室では、植物がもつオーキシン分解系を出芽酵母に移植することで、オーキシン添加により標的タンパク質を短時間に分解除去できるオーキシンデグロン法を確立しました(Nishimura et al. Nature Methods, 2009)。その後、改良を加えより効率よく分解誘導するデグロンも報告しました(Kubota et al. Molecular Cell, 2013)。現在、オーキシンデグロン法は出芽酵母コンディショナル変異株作製のための重要な研究ツールになりつつあります。そこで、私たちはオーキシンデグロン変異株を1回のトランスフォーメーションで作製する詳細なプロトコールを出版しました。実験に必要なプラスミドや酵母株は、全てナショナルバイオリソース(http://yeast.lab.nig.ac.jp/nig/)より入手可能です。本プロトコールが出芽酵母研究コミュニティーの発展に役立つことを願っています。
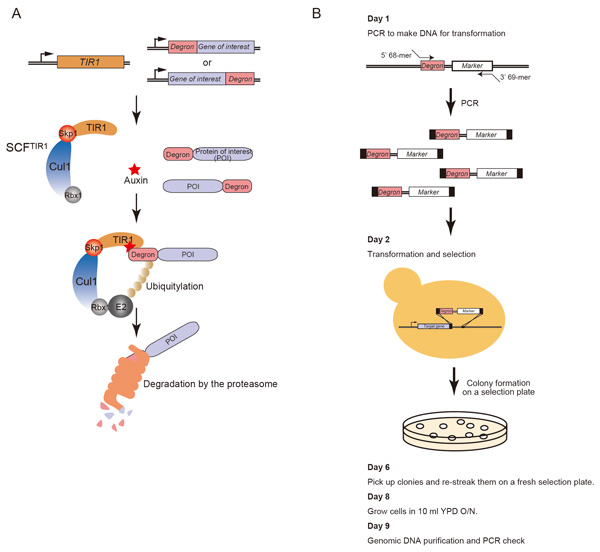
(A) オーキシンデグロン法において、どのようにデグロン融合タンパク質がオーキシン存在下で分解されるかを示した図。 (B) オーキシンデグロン変異株作製のプロトコールを図示した。
2014年度国立遺伝学研究所「共同研究」追加募集について(募集は締め切りました)
セントロメアを構成するクロマチン構造
分子遺伝研究部門・深川研究室
The centromere: chromatin foundation for the kinetochore machinery
Tatsuo Fukagawa, and William C. Earnshaw Dev. Cell 30,496-508,2014; DOI:10.1016/j.devcel.2014.08.016細胞分裂の過程では、紡錘体微小管と染色体上のキネトコア (動原体) 構造が結合することで、正確な染色体分配が遂行される。キネトコアが形成される染色体領域であるセントロメアは、DNAの塩基配列ではなくエピジェネティックな要因で規定される。そして、セントロメアに特異的なヒストンであるCENP-Aが、セントロメアの規定に関わるエピジェネティックな目印として考えられている。分子遺伝研究部門の深川と前遺伝研客員教授の Bill Earnshaw (Edinburgh大学)は、この総説論文においてセントロメアクロマチン研究においての歴史的な論文から最新のセントロメア研究のトピックまでを幅広く解説している。何故セントロメアに反復配列が多いのか、クロマチン因子や修飾はセントロメア形成にどのように関わっているのか、CENP-Aクロマチンの形成機構はどうなっているのか等の疑問をはじめセントロメアのクロマチン構造の理解のためにどのように人工セントロメアや人工染色体が活用されているのかについても詳述している。
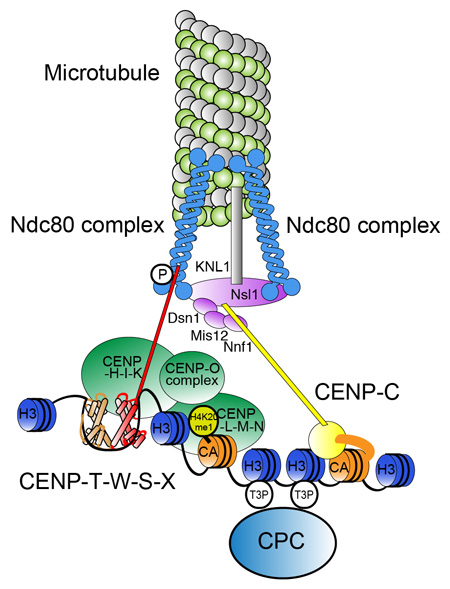
セントロメアの分子構造。CENP-Aから成るヌクレオソームとH3から成るヌクレオソームに加えてCENP-T-W-S-Xがヌクレオソソーム様の構造を形成し、それらが協調してセントロメアに特異的なクロマチン構造が構築される。そのクロマチンを各種タンパク質が認識、集合して機能的な動原体が形成される。















