Archive
- Home
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
元国立遺伝学研究所長 富澤純一氏の逝去について
元国立遺伝学研究所長(第6代)富澤 純一 氏(享年92歳)におかれましては、2017年1月26日(木)に逝去されました。故人のご功績を偲びここに謹んでお悔やみ申し上げるとともに、心からご冥福をお祈りします。

【富澤純一氏について】
富澤純一氏は、1947年に東京帝国大学を卒業後、国立予防衛生研究所化学部長、大阪大学教授、米国国立保健衛生研究所分子遺伝学部門部長を歴任した後、1989年に国立遺伝学研究所長に就任し、8年間に渡り、当研究所の発展に貢献されました。
研究面においては、細菌に感染するウイルス「T4ファージ」を用い遺伝的組換えがDNAの切断と再結合によって起こることの立証、プラスミド(環状2重鎖DNA)の試験管内での完全複製に初めて成功、DNAの複製開始がRNAによって制御されていることを示すなど、日本の分子生物学の草分けとして数々の業績を挙げられました。 これらの功績が認められ、日本学士院会員や全米科学アカデミー外国人会員に選出され、2000年には文化功労者に顕彰されました。日本分子生物学会の学会誌(英文学術誌)「Genes to Cells」では、初代編集長を1996年から2005年まで務めるとともに、若手研究者助成のための基金を寄付し、2011年から「富澤純一・桂子基金」として運用されています。
遺伝子内部におけるクロマチン修飾の役割と制御機構
Gene-body chromatin modification dynamics mediate epigenome differentiation in Arabidopsis
Soichi Inagaki, Mayumi Takahashi, Aoi Hosaka, Tasuku Ito, Atsushi Toyoda, Asao Fujiyama, Yoshiaki Tarutani, Tetsuji Kakutani
The EMBO Journal. Published online 18.01.2017 DOI:10.15252/embj.201694983
ヒストンH3の9番目のリジンのメチル化(H3K9me)やDNAのシトシン残基のメチル化は、トランスポゾンの転写抑制を介してゲノムの安定性に寄与しています。不思議なことに、これらの抑制目印は転写制御領域として知られるプロモーターだけではなく、遺伝子の内部(body)にも見つかります。今回我々は、活性な遺伝子の内部に抑制目印が蓄積するシロイヌナズナの変異体を用いて、遺伝子内部におけるクロマチン修飾の制御と意義にアプローチしました。シロイヌナズナの変異体ibm1では、多数の活性遺伝子の内部にH3K9meやDNAメチル化が蓄積し、結果的に花や葉などの発生異常が引き起こされます(図のA)。今回の研究では、ibm1変異体背景であるにも関わらず発生異常が生じない新たな変異体を同定し(図のA)、これを用いることで下流経路の理解を目指しました。同定された変異体の原因遺伝子であるLDL2はヒストン脱メチル化酵素をコードしていました。ヒストン修飾解析などから、LDL2が遺伝子内部の修飾による転写抑制を仲介することがわかりました。興味深いことにLDL2は、これまでに機能がはっきりしていなかったH3リジン4のモノメチル化(H3K4me1)を遺伝子内部において減少させることで転写抑制を引き起こしていました。同様に、遺伝子内部の修飾による転写抑制経路が多くのトランスポゾンでも働いていることがわかりました。本研究の結果は、これまで注目されてこなかった遺伝子内部に局在するH3K4me1の重要性を示すとともに、生物がトランスポゾンを特異的に抑制する機構の理解につながるものです。
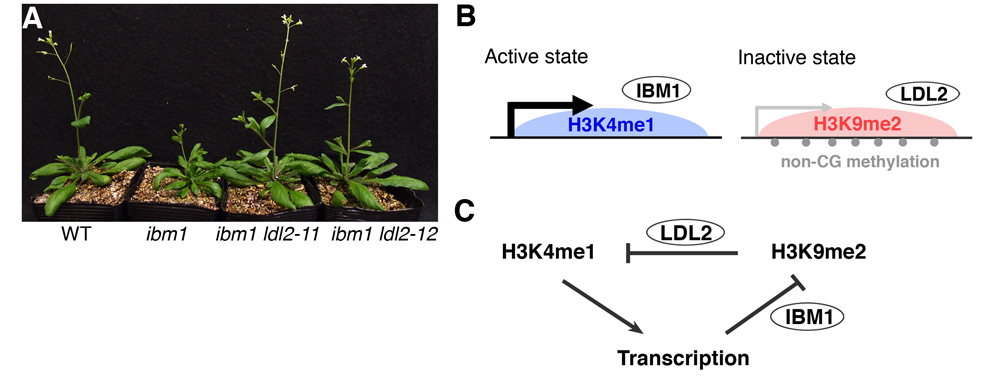
(A) ibm1変異体は小さく異常な形態の葉などの発生異常を示すが、ibm1 ldl2二重変異体は発生異常が回復する。(B)活性な状態と不活性な状態のクロマチンにおける修飾状態はIBM1、LDL2の2つのヒストン脱メチル化酵素によって制御される。(C)フィードバックモデル。転写している領域ではIBM1が働き、H3K9me2を抑える。逆に、H3K9me2が蓄積するとLDL2が働き、H3K4me1を減少させることで転写を抑える。
ウェット&ドライによる機能するカイコガエノラーゼ遺伝子群の同定
DBCLS
比較ゲノム解析研究室
先端ゲノミクス推進センター
Identification of functional enolase genes of the silkworm Bombyx mori from public databases with a combination of dry and wet bench processes
Akira Kikuchi, Takeru Nakazato, Katsuhiko Ito, Yosui Nojima, Takeshi Yokoyama, Kikuo Iwabuchi, Hidemasa Bono, Atsushi Toyoda, Asao Fujiyama, Ryoichi Sato and Hiroko Tabunoki
BMC Genomics, 18:83, 2017 DOI:10.1186/s12864-016-3455-y
現在、ネットでゲノム配列情報を簡単に取得できますが、それらの中には間違ったアノテーションが含まれており、その後の機能解析において正確な解釈の妨げになっています。そこで、東京農工大学農学部 天竺桂弘子講師を中心とするグループは、ドライとウェットベンチの連携手法を開発し、公共データベースから生体内で機能しているカイコガ(Bombyx mori)のエノラーゼ遺伝子群を同定しました。
遺伝学研究所 比較ゲノム解析研究室 豊田敦特任教授、同研究所先端ゲノミクス推進センター 藤山秋佐夫特任教授はトランスクリプトーム配列解読をおこない、ライフサイエンス統合データベースセンター(DBCLS) 仲里猛留特任助教、坊農秀雅特任准教授は公共データベース中からの候補遺伝子探索の技術開発をおこなうことで本研究に貢献しました。
またDBCLSが提供する画像・イラスト集Togo Picture Galleryの生物種のイラストが複数のFigure(fig1, fig3)に利用されております。
本研究は、国立遺伝学研究所が有する遺伝研スーパーコンピュータシステムを利用し、また国立遺伝学研究所公募型共同研究 NIG-JOINT (2014-A171 & 2015-A155)の支援を受けました。
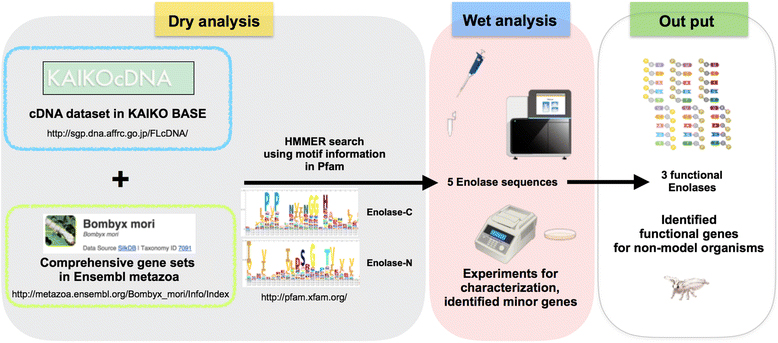
ドライとウェットベンチの連携手法を開発、公共データベースから生体内で機能しているカイコガ(Bombyx mori)のエノラーゼ遺伝子群を同定しました。
ミカンの親はどの品種?〜遺伝解析により60種以上のカンキツ類の親子関係が明らかに〜
![]()
Hybrid Origins of Citrus Varieties Inferred from DNA Marker Analysis of Nuclear and Organelle Genomes
Tokurou Shimizu , Akira Kitajima, Keisuke Nonaka, Terutaka Yoshioka, Satoshi Ohta, Shingo Goto, Atsushi Toyoda, Asao Fujiyama, Takako Mochizuki, Hideki Nagasaki, Eli Kaminuma, Yasukazu Nakamura
PLoS One. 11, e0166969. DOI:10.1371/journal.pone.0166969
情報・システム研究機構 国立遺伝学研究所 大量遺伝情報研究室 神沼英里助教、中村保一教授、比較ゲノム解析研究室 豊田敦特任教授、同研究所先端ゲノミクス推進センター 藤山秋佐夫特任教授、農研機構 果樹茶業研究部門カンキツ研究領域 清水徳朗上級研究員、京都大学農学研究科 北島宣教授らは共同で、15種のカンキツ類の全ゲノム配列を解読し、品種・系統269点について高度な遺伝解析をおこなった結果、60品種以上のカンキツ類の親子関係を明らかにしました。
日本人にとって身近な食材であるカンキツ類には、温州ミカン、レモン、ユズなど多種多様な品種があります。それらの品種は少数の祖先品種の掛け合わせにより栽培品種として選抜されてきたと考えられています。しかしながらカンキツ類は遺伝的多様性に富んでいるため、これらの品種の親子関係はほとんどわかっていませんでした。
研究グループは、品種を特徴づけるDNAマーカーを開発し、カンキツ類の遺伝解析をおこないました。その結果、温州ミカンをはじめとする22品種で両親となる品種を特定し、さらに、種子親と花粉親の組合せを明らかにしました。また、45の在来品種(5)において片親や起源、親子関係を明らかにしました。
この研究により、インド東北部からアジアにわたる地域が起源と考えられている祖先品種が世界各地へどのように伝来して現在のような多様な品種になったのか明らかになると期待されます。さらに、未利用の品種との交配組合せの可能性を示すことで、優れた性質をもつ新しい品種の開発へ応用されることが期待できます。
遺伝学研究所 比較ゲノム解析研究室 豊田敦特任教授、同研究所先端ゲノミクス推進センター 藤山秋佐夫特任教授は主要品種のゲノム配列決定をおこない、大量遺伝情報研究室 神沼英里助教、中村保一教授はカンキツ類SNP(一塩基多型)解析の技術開発をおこなうことで本研究に貢献しました。
本研究は、新領域融合プロジェクト「生命システム」サブテーマ1超大量ゲノム情報(代表:藤山秋佐夫)、農林水産省委託プロジェクト研究「ゲノム情報を活用した農畜産物の次世代生産基盤技術の開発プロジェクト」(代表:清水徳朗)、科学研究費補助金 基盤研究B「海のカンキツロードの解明」(代表:北島 宣)の支援を受けて実施されました。
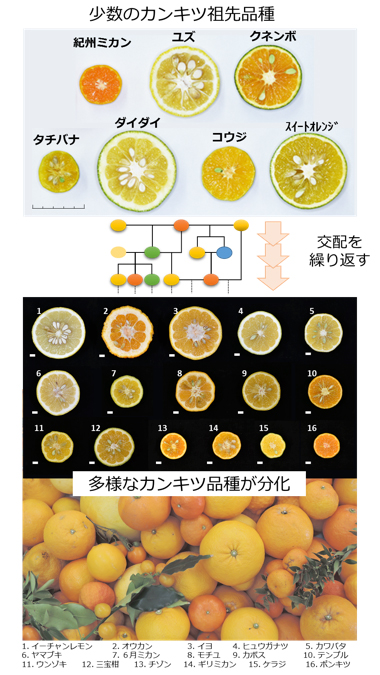
少数のカンキツ類祖先品種から多様なカンキツ類品種が分化した















