Archive
- Home
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
細胞内共生による葉緑体誕生に必須であった細胞と葉緑体の分裂同調化機構の解明
Chloroplast division checkpoint in eukaryotic algae.
Nobuko Sumiya, Takayuki Fujiwara, Atsuko Era, and Shin-ya Miyagishima
Proc. Natl. Acad. Sci. USA. (2016). Published online, DOI:10.1073/pnas.1612872113
真核細胞内で呼吸の場となるミトコンドリアや光合成の場である葉緑体は、バクテリアが真核細胞内に共生することによって誕生しました。
バクテリア細胞が宿主である真核細胞内に永続的に維持され、細胞内小器官へと変遷していくためには、バクテリア細胞の分裂が宿主細胞の分裂に同期される必要があります。この分裂を同調化させるメカニズムの解明は、真核細胞の誕生を理解する上でも重要ですが、その知見は長い間得られていませんでした。
これまでに私たちは、葉緑体は、葉緑体中央に形成される細胞核コードのタンパク質群からなるリングが収縮することによって分裂することを明らかにしてきました。多くの藻類細胞は1ないし数個しか葉緑体をもたず、葉緑体は宿主細胞周期の決まった時期に一度だけ分裂することが知られています。私たちは、このような藻類細胞では、葉緑体分裂リングを構成するタンパク質群が宿主細胞周期のS期のみに発現することにより、葉緑体分裂の開始がS期に限定されることも示してきました。しかし一回の細胞周期に葉緑体分裂が一回に限定される仕組みは不明でした。
今回私たちは、単細胞紅藻シアニディオシゾンにおいて開発した導入遺伝子誘導発現系を用いて葉緑体分裂を停止させたときの宿主細胞周期への影響を調べました。その結果、葉緑体分裂位置の収縮前に分裂を停止させた場合には宿主細胞周期がM期前期で停止すること、収縮開始後に分裂を停止させた場合には宿主細胞周期は停止しないことがわかりました。これらの結果は、葉緑体分裂がひとたび開始すると、細胞周期が葉緑体分裂リング構成タンパク質群の発現を終えるM期中期まで進行し、その結果葉緑体分裂が1回に限定されることを示しています。また、葉緑体分裂と宿主細胞分裂の同調化は、宿主細胞による葉緑体分裂リング形成時期の限定と、葉緑体分裂リングの収縮開始による宿主細胞周期のM期中期以降への進行許可という、宿主細胞と葉緑体双方による制約の掛け合いによって成立していることもわかりました。さらに同様の実験結果が、葉緑体誕生後初期に分岐した灰色藻でも認められたことから、このような葉緑体と細胞の分裂同調化メカニズムは藻類の共通祖先で葉緑体誕生時に獲得されたものであることも示唆されます。
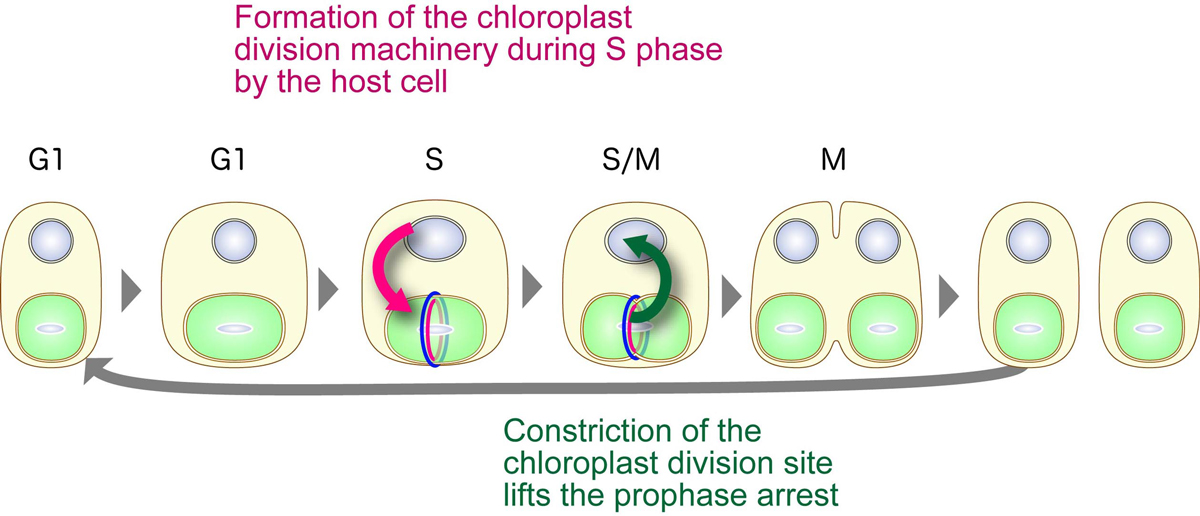
宿主真核細胞と葉緑体の相互作用による分裂同調化機構。葉緑体分裂はその中央に形成されるリング状装置の収縮によっておこる。この装置はそれぞれ核コードの、シアノバクテリア由来のFtsZ(赤)、宿主由来のダイナミン(青)等で構成される。宿主細胞は、葉緑体分裂リング構成タンパク質の発現、葉緑体分裂開始を自身のS期に限定する。一方で葉緑体分裂リング収縮は宿主細胞のM期前期停止を解除する。
アサガオの全ゲノム解読 ― アサガオの学術研究100年目のイノベーション
![]()
Genome sequence and analysis of the Japanese morning glory Ipomoea nil
Atsushi Hoshino, Vasanthan Jayakumar, Eiji Nitasaka, Atsushi Toyoda, Hideki Noguchi, Takehiko Itoh, Tadasu Shin-I, Yohei Minakuchi, Yuki Koda, Atsushi Nagano, Masaki Yasugi, Mie Honjo, Hiroshi Kudoh, Motoaki Seki, Asako Kamiya, Toshiyuki Shiraki, Piero Carninci, Erika Asamizu, Hiroyo Nishide, Sachiko Tanaka, Kyeung-Il Park, Yasumasa Morita, Kohei Yokoyama, Ikuo Uchiyama, Yoshikazu Tanaka, Satoshi Tabata, Kazuo Shinozaki, Yoshihide Hayashizaki, Yuji Kohara, Yutaka Suzuki, Sumio Sugano, Asao Fujiyama, Shigeru Iida, and Yasubumi Sakakibara
Nature Communications. 7, Article number: 13295 (2016) DOI:10.1038/ncomms13295
基礎生物学研究所の星野敦助教、慶應義塾大学理工学部の榊原康文教授、九州大学大学院理学研究院の仁田坂英二講師らは、日本独自の研究資源であるアサガオの全ゲノム配列をほぼ完全に解読することに成功しました。アサガオが約43,000個の遺伝子をもっていることや、その多彩な品種を生み出すもとになった動く遺伝子(トランスポゾン)のゲノム上の分布状況、「渦」と呼ばれる変異の原因遺伝子なども新たに判明しました。アサガオは日本伝統の園芸植物であり、花色や形態形成などの分子遺伝学的な解析材料としての重要性から、活発に研究されています。今回の成果によりゲノム情報基盤が整備されたことで、アサガオがモデル植物として世界中のより多くの研究者に活用されることが期待されます。本研究成果は2016年11月8日に国際学術誌 “Nature Communications”(ネイチャー・コミュニケーションズ)に掲載されます。本研究は文部科学省科学研究費新学術領域研究「ゲノム支援」(代表:小原雄治 国立遺伝学研究所)の一環として実施されました。
国立遺伝学研究所 生命情報研究センター比較ゲノム解析研究室 豊田敦特任教授、先端ゲノミクス推進センター 藤山秋佐夫特任教授、野口英樹特任准教授らのグループは第2世代、第3世代シーケンサーを駆使して全ゲノムデータを提供するとともに、断片化したDNA配列の再構築に関する技術サポート、BACライブラリの両端ならびに全長配列の決定、遺伝子多型解析などに用いられるRad-seqサンプルの配列決定などで本研究に貢献しました。

図1 さまざまな色とかたちのアサガオ

図2 左:全ゲノム解読を行ったアサガオの実験系統、東京古型標準型。 右:変化アサガオ「渦小人」















