Archive
- Home
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
遺伝研 一般公開2018 : 4月7日(土)開催 – 講演の動画を配信中
大量遺伝情報研究室 藤澤貴智 特任研究員がLODチャレンジ2017 アイデア賞優秀賞を受賞

(JODチャレンジ2017審査委員長)、
DBCLS 山本泰智 特任准教授、
大量遺伝情報研究室 藤澤貴智 特任研究員
大量遺伝情報研究室 藤澤貴智 特任研究員は、DBCLS 山本泰智 特任准教授と連名で、LODチャレンジ2017 において、アイデア賞優秀賞を受賞しました。LOD (Linked Open Data)は、世界中のだれもが意味情報を付与したデータを公開することで、インターネット上を一つの知識ベースとして扱うことを可能にすることのできる仕組みです。LODチャレンジは、そのデータ公開や技術開発に関するコンテストであり、受賞した構想は、データ公開を促進するためのアイデアです。ROIS URAステーションにおいて共同研究打合せ中に生まれ、現在、研究開発の実施を計画しております。
▶ Linked Open Data チャレンジ Japan 2017 受賞作品発表
LODチャレンジ2017は、さまざまな分野でデータ作りやデータの活用にチャレンジされている方々による活動の発表の場を提供するために、新たなデータづくり、データ公開、データ共有の仕掛けやオープンデータ活用のアイディア、アプリケーションなどを「作品」として募集し表彰するものです。
▶ 大量遺伝情報研究室
ウンシュウミカンの全ゲノムを解読 -カンキツ品種改良の効率化に期待-
![]()
Draft sequencing of the heterozygous diploid genome of Satsuma (Citrus unshiu Marc.) using a hybrid assembly approach
Tokurou Shimizu, Yasuhiro Tanizawa, Takako Mochizuki, Hideki Nagasaki, Terutaka Yoshioka, Atsushi Toyoda, Asao Fujiyama, Eli Kaminuma, Yasukazu Nakamura
Frontiers in Genetics 05 December 2017 DOI:10.3389/fgene.2017.00180
1.農研機構は情報・システム研究機構国立遺伝学研究所と共同で、ウンシュウミカン「宮川(みやがわ)早生(わせ)」の全ゲノム配列を解読しました。ゲノムの大きさは約3億6000万塩基対でした。
2.ゲノム配列中に約2万9千個の遺伝子が存在すると推定し、その中からカンキツの着色に関わるカロテノイド生合成の遺伝子や、結実性に関わるジベレリンの生合成遺伝子91個を特定しました。
3.ウンシュウミカンの両親であるキシュウミカンとクネンボの塩基配列も解析して比較したところ、クネンボの片親がキシュウミカンであること、つまり、ウンシュウミカンはキシュウミカンの子供にさらにキシュウミカンが交配されて生まれたことがわかりました。
4.本成果で得られた全ゲノム配列を利用すれば、ゲノムワイド関連解析を利用した果実形質や栽培性に関わる重要遺伝子の機能推定が高速化され、カンキツの品種育成を効率化できると期待されます。
5.本成果は国際科学雑誌「Frontiers in Genetics」に12月5日に掲載されました。
遺伝研の貢献
国立遺伝学研究所 比較ゲノム解析研究室 豊田敦特任教授、同研究所先端ゲノミクス推進センター 藤山秋佐夫特任教授は宮川早生品種のDNA解読をおこない、断片状の塩基配列を生成しました。その断片塩基配列データをもちいて、大量遺伝情報研究室 谷澤靖洋特任研究員、望月孝子特任研究員、神沼英里助教、中村保一教授は、染色体配列を再構築するゲノムアセンブリ解析や遺伝子注釈解析を行いました。解析には遺伝研スーパーコンピュータを使用しました。
研究支援
本研究は、農林水産省委託プロジェクトゲノム情報を活用した農産物の次世代生産基盤技術の開発プロジェクト「多数の遺伝子が関与する形質を改良する新しい育種技術の開発」(NGB)、ROIS新領域融合プロジェクト「生命システム」サブテーマ 1 超大量ゲノム情報(代表:藤山秋佐夫)、科学研究費補助金の支援を受けて実施されました。
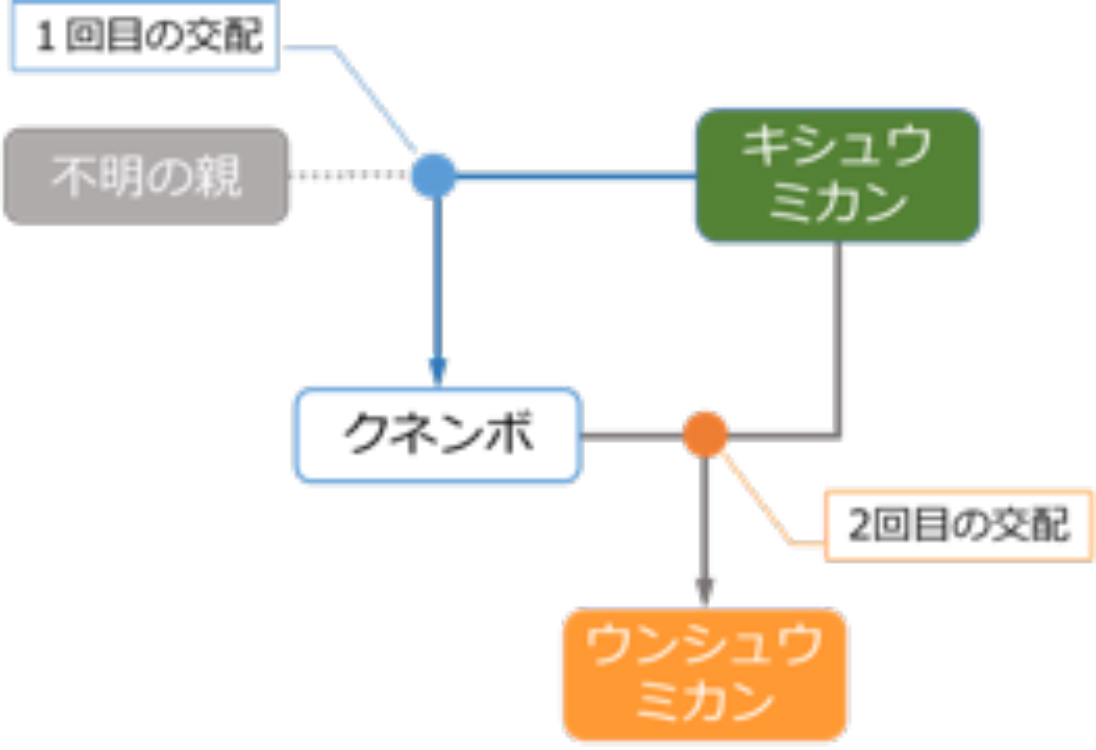
図1.全配列解析から明らかとなったウンシュウミカンの成立経過
ウンシュウミカンの片親であるクネンボはキシュウミカンの子であることを全配列解析から確認しました。すなわち、ウンシュウミカンはキシュウミカンの子供に、さらにキシュウミカンが交配された結果生まれた品種であることがわかりました(クネンボのもう一方の片親は不明です)。

図2:ウンシュウミカン、キシュウミカン、クネンボの果実
プレスリリース資料中でご紹介した成果「DNAの違いから、芽生え段階でカンキツの様々な果実特性を高精度に予測」についてこちらでお読みいただけます
イネの葯組織特異的な小分子RNA生合成を誘導する転写因子を特定
EAT1 transcription factor, a non-cell-autonomous regulator of pollen production, activates meiotic small RNA biogenesis in rice anther tapetum.
Seijiro Ono, Hua Liu, Katsutoshi Tsuda, Eigo Fukai, Keisuke Tanaka, Takuji Sasaki, Ken-Ichi Nonomura.
PLOS Genetics, 14 (2), e1007238, (2018) DOI:10.1371/journal.pgen.1007238
小分子RNAは、遺伝子の転写や翻訳の抑制を介して、個体発生やウィルス防御などで重要な働きをします。私たちは今回、葯を構成するタペート組織で発現するイネEAT1転写因子が、減数分裂期特異的に24塩基長の小分子RNA生産を誘導することを発見しました(図1A, B)。EAT1は、小分子RNAの素材となる前駆体RNA、および前駆体を24塩基長に切断するDCL5酵素をコードする遺伝子の転写を直接活性化していたのです(図1C)。
また、小分子RNAの一部はタペート細胞に隣接する減数分裂細胞へと移動する可能性が、減数分裂細胞で発現するMEL1タンパク質の解析から明らかになりました(図1C)。小分子RNAが、植物の減数分裂細胞と周辺の体細胞の協調的な発生制御を取り持つ伝達役として機能する可能性を示唆する成果です。
本研究成果は、米国オンラインジャーナル「PLOS Genetics」に平成30年2月12日に掲載されました。
本研究は、遺伝研と包括連携協定を結ぶ東京農業大学、および新潟大学との共同研究の成果です。また、文科省科研費・新学術領域「植物新種誕生原理」(17H05849)、科研費・基盤研究(A) (25252004)、東京農業大学生物資源ゲノム解析センター生物資源ゲノム解析拠点事業、遺伝研共同研究 (NIG-JOINT 2015-61、2016-65、87A2017)、および遺伝研博士研究員制度による支援を受けました。
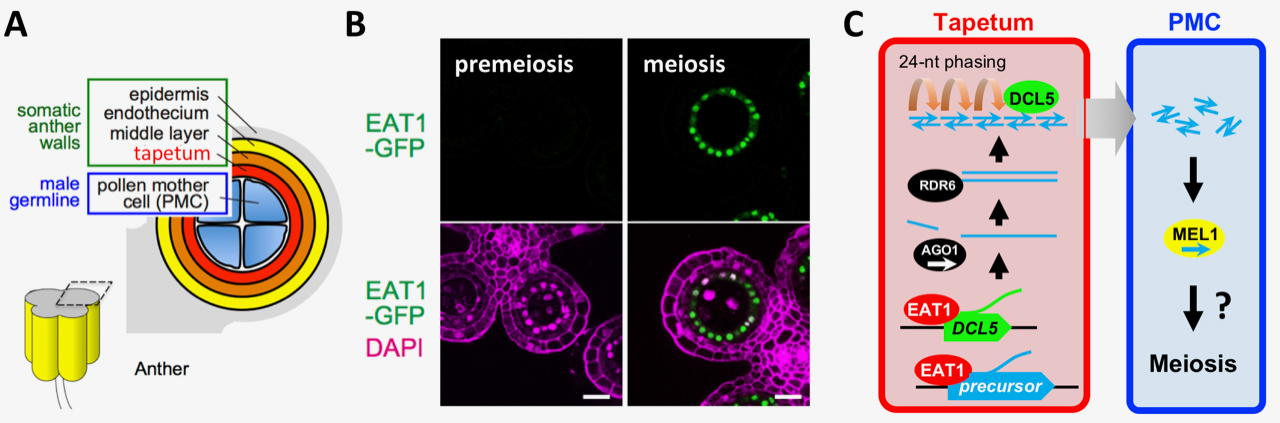
図1. EAT1転写因子は葯タペート組織で24塩基長の減数分裂small RNA (sRNA) の生合成を促進
(A) 葯 (anther) の断面図。タペート組織 (tapetum) は減数分裂細胞(PMC)と接する。(B) EAT1転写因子(緑)は減数分裂期 (meiosis) タペート細胞の核に蓄積するが、前減数分裂期 (premeiosis) では発現しない。スケールバー; 20µm。(C) EAT1はタペートでsRNA前駆体とDCL5遺伝子の転写を活性化する。前駆体RNAは切断・二本鎖化を経てDCL5により24塩基に切断される。PMCで発現するMEL1と24塩基長sRNAの結合から、sRNAの細胞間移動が示唆される。
「先進ゲノム支援」情報解析講習会のご案内
斎藤成也教授の論文引用が5万件を超えました
斎藤成也教授が1987年にMolecular Biology and Evolution に発表された下記論文の引用が5万件(Google Scholar Citations)を超えました。(2018年2月6日現在)
▶ 論文:The neighbor-joining method: a new method for reconstructing phylogenetic trees.
N Saitou, M Nei
Molecular biology and evolution 4 (4), 406-425, 1987
論文引用とは:優れた論文は、ほかの論文に引用される回数が多くなることから、論文が引用される回数は、その論文の質を表す指標のひとつと考えることができます。このため、被引用回数が多い論文は知的貢献度が高いと見なされています。
Google Scholar Citations では、著者ごとの論文とその引用回数を明示的に確認することができます。















