Archive
- Home
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
イネ栽培化の初期過程に重要な役割を果たした3つの遺伝子
プレスリリース
A stepwise route to domesticate rice by controlling seed shattering and panicle shape
Ryo Ishikawa, Cristina Cobo Castillo, Than Myint Htun, Koji Numaguchi, Kazuya Inoue, Yumi Oka, Miki Ogasawara, Shohei Sugiyama, Natsumi Takama, Chhourn Orn, Chizuru Inoue, Ken-Ichi Nonomura, Robin Allaby, Dorian Q Fuller and Takashige Ishii
PNAS (2022) 119, e2121692119 DOI:10.1073/pnas.2121692119
神戸大学大学院農学研究科を中心とした、国立遺伝学研究所、英国・ユニバーシティカレッジロンドン、ウォーリック大学、ミャンマー・イエジン農科大学、カンボジア・農務省からなる国際共同研究チームは、栽培イネ(Oryza sativa L.)が野生イネ(O. rufipogon)から分化する初期過程で、3つの遺伝子の変化がコメの収量を飛躍的に向上させた可能性を明らかにしました。
種子脱粒性(用語説明)の喪失は、収量の増加につながる栽培化の重要なステップです。研究チームは、戻し交雑により野生イネの脱粒性関連遺伝子を栽培イネの遺伝子に置き換え、栽培化の過程を実験的に再現することを試みました。すると、これまで栽培化の主要因と考えられてきた脱粒性遺伝子sh4の栽培イネ型への置換だけでは不十分で、新たに同定したqSH3遺伝子とsh4遺伝子がともに栽培イネ型に置換されることで、野生イネのもつ脱粒性がさらに抑制されることがわかりました。それでも自然環境下ではかなりの種子が脱粒し、収穫効率の向上には更なる因子が必要と思われました。
野生イネは穂が開帳しており、栽培イネの穂とは大きく形が異なります(図1)。研究チームは、qSH3とsh4に加え、穂の開帳性を制御するSPR3遺伝子を栽培イネ型に置換した野生イネ、すなわち脱粒性喪失かつ穂が閉じた野生イネでは、飛躍的に収量が増大することを見出しました(図1)。穂が閉じる性質は、脱粒した種子が互いに絡まり合い、穂に留まり易くなって収穫増加につながることから、栽培化因子のひとつと考えられています。また、構造力学解析の結果から、閉じた穂では重力による種子基部の離層への負荷が開いた穂よりも小さいため、脱粒性の抑制と更なる収量増をもたらした可能性が考えられました(図2)。
今回の成果は、種子脱粒性の人為的な制御による新たな多収イネ品種の開発につながる可能性があります。本研究の一部は遺伝研共同研究事業(NIG-JOINT 83A2016-2018)として実施されました。文部科学省ナショナルバイオリソース(NBRP)イネの支援を受けて遺伝研が保存する野生イネ系統が貢献しています。
(用語説明)
種子脱粒性:野生植物が種子を飛散させて繁殖するための性質。種子基部に離層組織が形成されて引き起こされる。
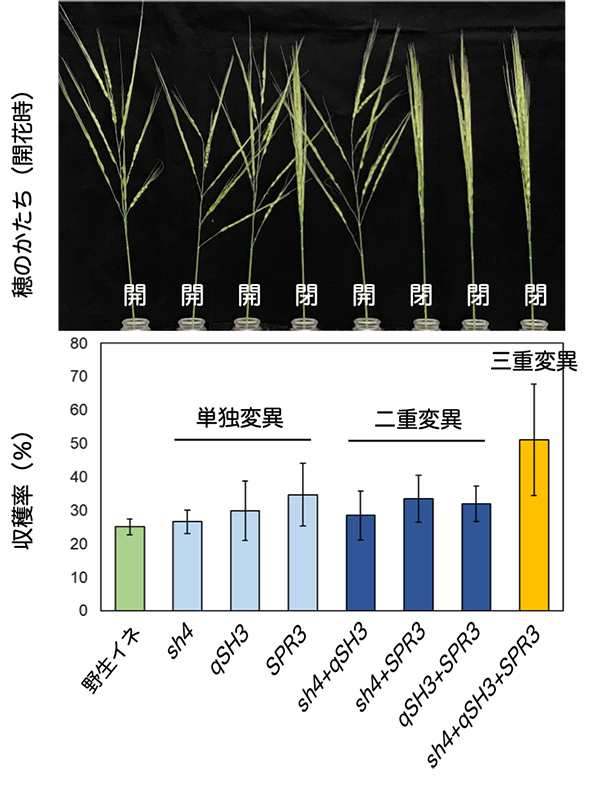
(上段)3つの遺伝子 (sh4, qSH3, SPR3) の栽培イネ型変異を様々な組み合わせで導入した野生イネ (O. rufipogon) 個体の穂の形。それぞれの個体の遺伝子組み合わせは下段の並びに対応。(下段)それぞれの遺伝子組み合わせの野生イネ個体における収穫率。 3つの変異を一緒に導入した個体では収穫率が顕著に上昇した。
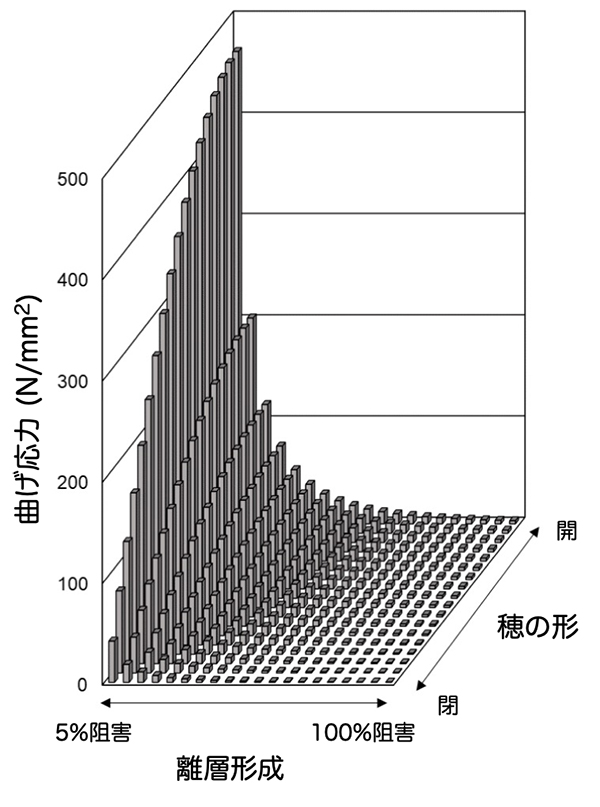
種子基部にかかる負荷、すなわち曲げ応力が高いほど種子は脱粒しやすくなる。sh4やqSH3などの導入で離層形成が阻害されるほど、またSPR3のような遺伝子の導入で穂の開帳度合いが低くなるほど曲げ応力は小さくなる。すなわち、野生イネの栽培化初期過程でsh4とqSH3に変異導入で脱粒性が低下したことに加え、SPR3変異導入により穂が閉じた形になることで、より高い収穫率が実現した可能性が高い。
考古学的遺物から見つかる古代ウイルスとその進化研究
Detection of Ancient Viruses and Long-Term Viral Evolution
Luca Nishimura, Naoko Fujito, Ryota Sugimoto, and Ituro Inoue*
Viruses (2022) 14, 1336 DOI:10.3390/v14061336
古代人の遺骨をはじめとする考古学的遺物には古代DNAや古代RNAが含まれています。これらのDNAやRNAには古代人や古代人に感染していた細菌、ウイルスなどのゲノム情報が含まれています。このゲノム情報を探索することにより、1997年から現在に至るまで遺骨やミイラ化した組織から20種類を超える古代ウイルスが見つかってきました。これらの古代ウイルスの中には100年前にスペイン風邪をもたらしたインフルエンザウイルスや1万年ほど前の遺骨から見つかったB型肝炎ウイルスなどが含まれています。これらの古代ウイルスゲノムを解析することにより、現代のウイルスだけからはわからない複雑かつ長期的なウイルスの進化過程などを明らかにすることができます。本総説では古代ウイルスの探索手法やこれまでに報告のあった古代ウイルスを紹介し、それらのゲノム情報を使ったウイルス進化研究や今後の展望について議論しています。
国立遺伝学研究所・人類遺伝研究室の西村瑠佳 学振特別研究員 (総研大生) と井ノ上逸朗教授らのグループによって執筆され、Viruses誌の特別号Virus Bioinformatics 2022に掲載されました。
謎のグルコース代謝経路「ポリオール経路」の生理機能を解明
~進化的に保存されたポリオール経路によるグルコース感知機構~
プレスリリース
The polyol pathway is an evolutionarily conserved system for sensing glucose uptake.
Hiroko Sano*, Akira Nakamura, Mariko Yamane, Hitoshi Niwa, Takashi Nishimura, Kimi Araki, Kazumasa Takemoto, Kei-ichiro Ishiguro, Hiroki Aoki, Yuzuru Kato, Masayasu Kojima
*責任著者
PLOS Biology 2022 June 10 DOI:10.1371/journal.pbio.3001678
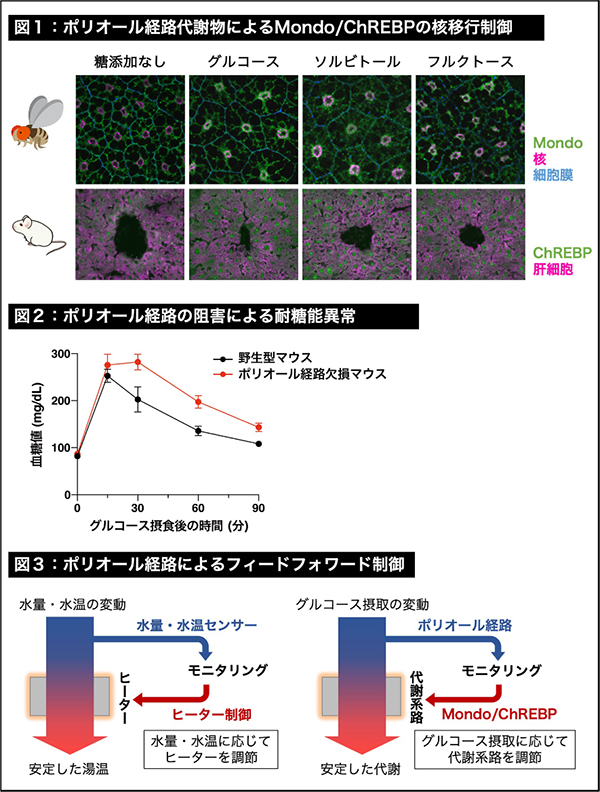
グルコース(ブドウ糖)は動物にとってエネルギー源でありDNAやRNAなどの生体分子の素にもなる重要な栄養素です。我々人間を含む動物は摂食で増える血中のグルコースを感知してグルコースを利用する仕組み(代謝)をうまく調節する必要があります。メタボリック症候群や糖尿病などの代謝疾患は、この調節がうまくいかなくなる病気です。動物がどのように血中のグルコースを感知しているかはまだ良く分かっていません。
今回、久留米大学分子生命科学研究所の佐野浩子講師は、これまで働きが分かっていなかった「ポリオール経路」と呼ばれる代謝経路がグルコース摂取の感知に重要であることを明らかにしました。この発見は久留米大学循環器病研究所、熊本大学発生医学研究所、同大学生命資源研究・支援センター、群馬大学生体調節研究所、国立遺伝学研究所との共同研究の成果です。
本研究は以下の研究支援事業の支援を受けて行われました。 日本学術振興会科学研究費助成事業 [16H06276 (AdAMS), 17H03686, 17K07419, 19H05743, 20H03265, 20K06647, 21H02489]、熊本大学発生医学研究所共同研究、群馬大学生体調節研究所内分泌・代謝学共同研究拠点共同研究、金原一郎記念医学医療振興財団基礎医学医療研究助成、文部科学省高深度オミクス医学研究拠点ネットワーク形成事業研究助成、文部科学省私立大学戦略的研究基盤形成支援事業
研究成果は2022年6月10日、生命科学系学術誌「PLOS Biology」に掲載されました。
遺伝研の貢献
画像解析技術を用いてMondo/ChREBPの核内局在を定量化しました。これにより、ポリオール経路がショウジョウバエとマウスでグルコース摂取依存的にMondo/ChREBPを活性化することが明らかになりました。
生きた細胞でのクロマチンのふるまい:1分子ヌクレオソームイメージングから学ぶ
Chromatin behavior in living cells: lessons from single-nucleosome imaging and tracking
*Satoru Ide, Sachiko Tamura, *Kazuhiro Maeshima *責任著者
BioEssays 2022 June 03 DOI:10.1002/bies.202200043
真核細胞において、長いDNAは糸巻きとして働くヒストンに巻かれ、ヌクレオソーム(直径約11nm)を形成します。このヌクレオソームに様々なタンパク質、RNAが結合したものをクロマチンと呼びます。細胞内でクロマチンはどのように収納されているのでしょうか?近年多くの証拠によって、クロマチンは従来考えられていたような30-nm線維を含む規則正しい階層構造ではなく、不規則かつ流動的な塊として存在することがわかってきました。クロマチンはゲノムDNA上で起きる化学反応 (転写やDNA複製など)の場として機能することから、クロマチンの折りたたみやその動きの観察はゲノムの機能を理解する上で重要です。
この総説論文では、生きた細胞におけるクロマチンのふるまいを観察するために非常に有力な手法であるヌクレオソーム1分子イメージングについて議論しました。この技術によってヌクレオソーム分子の動きを直接見ることで、ヌクレオソームが構成する構造やその物性を知るためのさまざまな情報が得られます。このイメージングはヌクレオソームの代わりに別のタンパク質を同様に蛍光標識すれば、核小体のような核内構造体の解析にも有効です。今後の1分子ヌクレオソームイメージングの汎用化が期待されます。
本研究は、日本学術振興会(JSPS) 科研費(21H02535、20H05936、21H02453)、上原記念生命科学財団の支援を受けました。
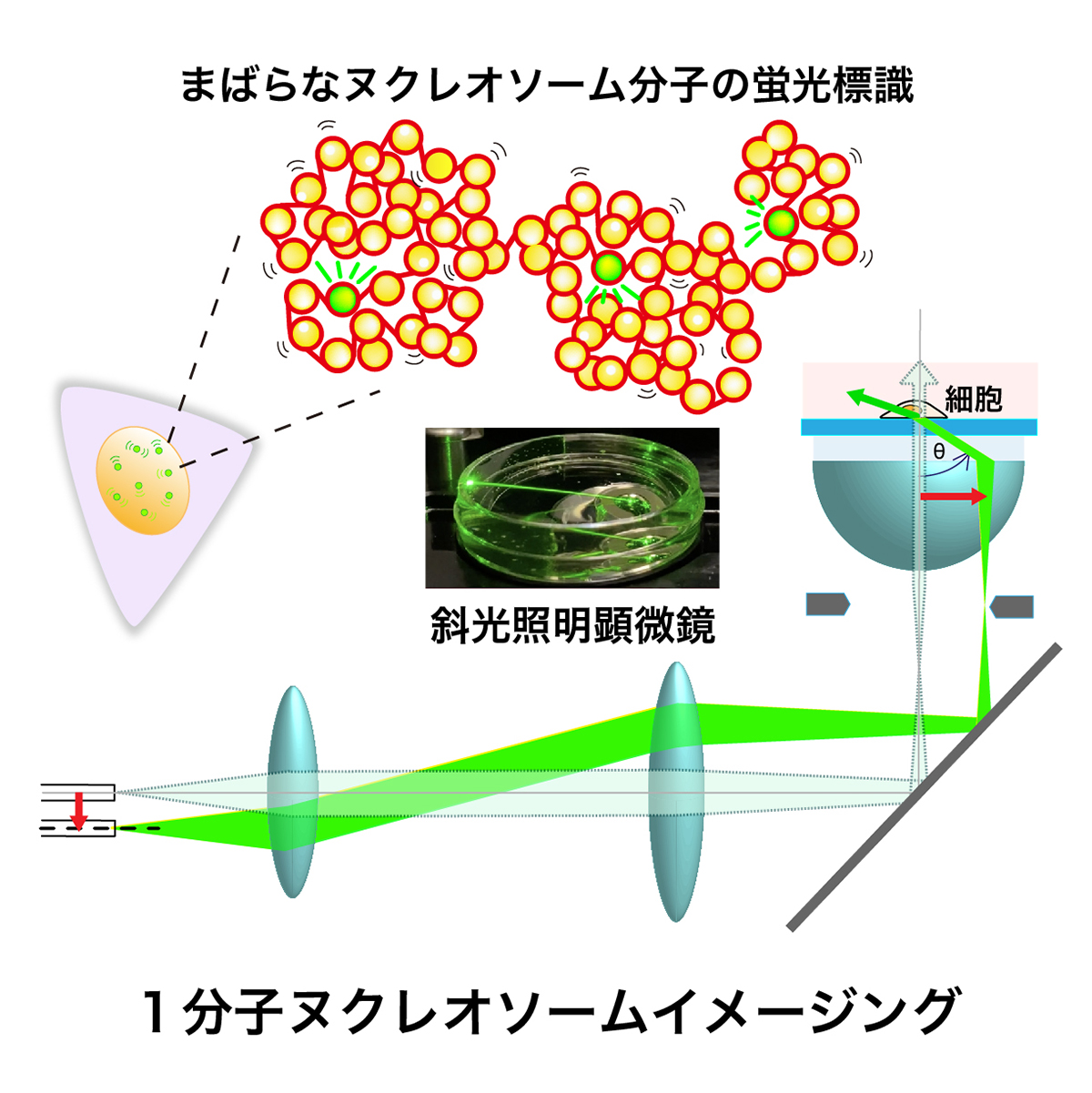
図:1分子ヌクレオソームイメージング。①まばらな分子(ヌクレオソーム)の蛍光標識と②励起光レーザーを斜めに当てること(斜光照明)で、可能な限り焦点面のみを励起し、焦点面以外由来のバックグラウンド蛍光を減少させることによって、生きた細胞で1個1個の分子を観察することができます。われわれが使用しているニコン社製ECLIPSE Ti TIRF蛍光倒立顕微鏡では、レーザー光の入射位置がレンズの中心部の場合、ビームは対物レンズから垂直に出ます(薄緑色の経路)。一方、レーザー光の入射位置をシフトさせると斜めのシート照明が得られます(濃緑色の経路)。
細胞周期の間期におけるクロマチンの定常的な動き
プレスリリース
Single-nucleosome imaging reveals steady-state motion of interphase chromatin in living human cells
Shiori Iida, Soya Shinkai, Yuji Itoh, Sachiko Tamura, Masato T. Kanemaki, Shuichi Onami, Kazuhiro Maeshima
Science Advances (2022) 8, eabn5626 DOI:10.1126/sciadv.abn5626
私たちの体は約40兆個の細胞からできています。それぞれの細胞には全長約2メートルにも及ぶ生命の設計図、ヒトゲノムDNAがクロマチンとして収納されています。クロマチンの収納のされ方については、近年、多くのことが分かってきた一方で、生きた細胞でのクロマチンの局所的なふるまい(ゆらぎ)についてはほとんど分かっていませんでした。
このたび、国立遺伝学研究所 ゲノムダイナミクス研究室 飯田史織 総研大生(SOKENDAI特別研究員)・伊藤優志 学振特別研究員 (現 東北大学助教)・田村佐知子 テクニカルスタッフ・前島一博 教授らのグループは、理化学研究所 新海創也 研究員・大浪修一 チームリーダー、国立遺伝研 分子細胞工学研究室 鐘巻将人 教授と共同で、光学顕微鏡の分解能を超える超解像蛍光顕微鏡を駆使することで、生きた細胞内のクロマチンのゆらぎを観察することに成功しました(動画1)。
一つの細胞が二つの娘細胞に分裂する前、その準備としてDNAの量は二倍になり、DNAを収納する核の大きさも二倍以上になります(細胞周期 間期)。これまで、このような核内の環境の変化は、クロマチンの動きに大きな影響を与えるとされていました。しかし本研究によって、クロマチンのゆらぎの程度は、DNAの量や核の大きさの変化に影響されず、一定であることが示されました(動画2、図)。さらに、DNAの量を変えずに、核だけを大きくしても、クロマチンのゆらぎは変化しませんでした。クロマチンのゆらぎの程度は、DNAへの近づきやすさ(ゲノム情報の読み出しやすさ)に直結します。間期のあいだ、クロマチンのゆらぎの程度が一定に保たれることで、細胞は、DNAに書かれた遺伝情報を、常に同じ環境で読み出し、RNA転写やDNA複製など、必要な仕事を同じように実行できると考えられます。
本研究は、科学技術振興機構(JST) 戦略的創造研究推進事業(CREST) (JPMJCR15G2)、JST 次世代研究者挑戦的研究プログラム(JPMJSP2104)、日本学術振興会(JSPS) 科研費(20H05550、21H05763、19K23735、 20J00572、18H05412、19H05273、20H05936)、武田科学振興財団、上原記念生命科学財団の支援を受けました。
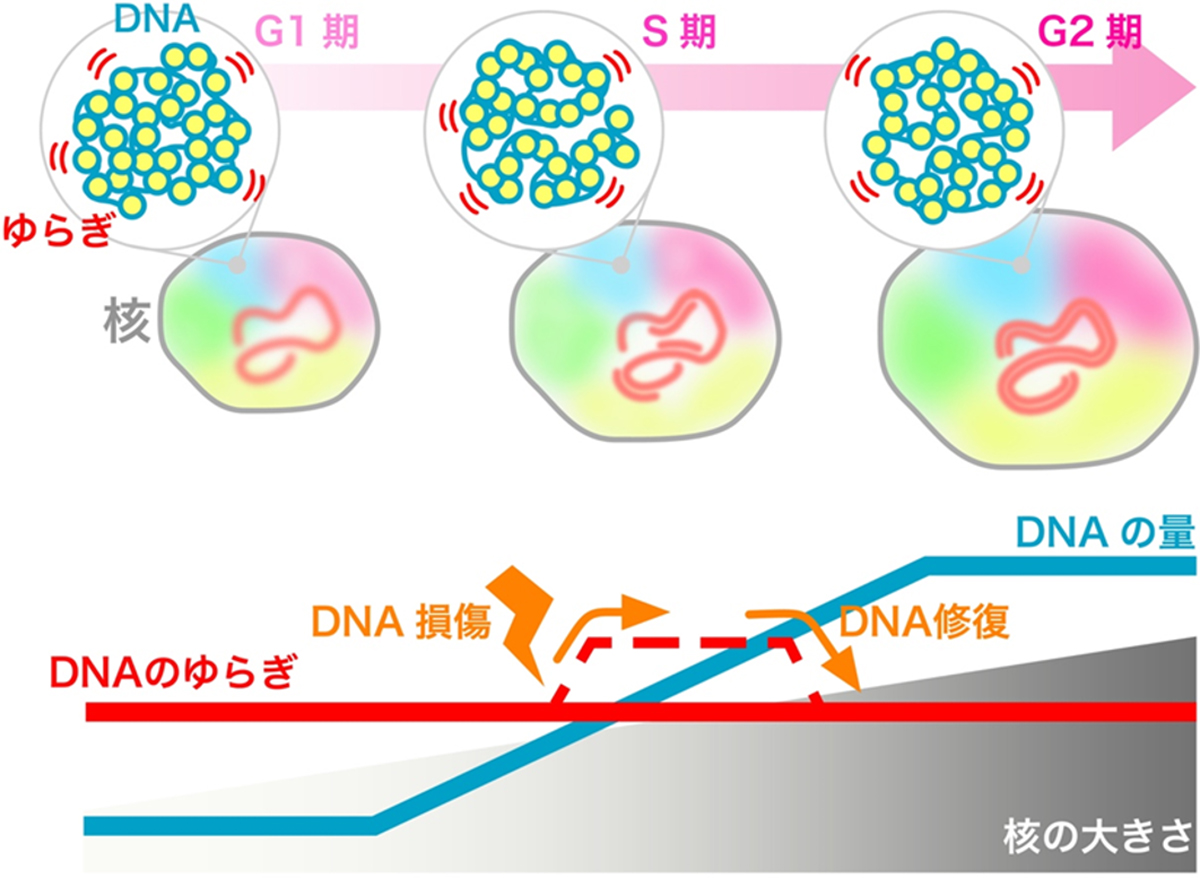
図: 細胞周期の間期(G1期→S期→G2期)において、核の大きさは二倍以上に大きくなり、DNAの量も二倍になる。このような核内の環境の変化にも関わらず、クロマチンのゆらぎは一定であった。間期のあいだ、クロマチンのゆらぎの程度が一定に保たれることで、細胞は、DNAに書かれた遺伝情報を、常に同じ環境で読み出し、RNA転写やDNA複製など、必要な仕事を同じように実行できると考えられる。
動画1: 超解像蛍光顕微鏡により観察された生きた細胞の核内におけるヌクレオソーム(クロマチンの最小ユニット)のゆらぎの動画。1個1個のドットが1個1個のヌクレオソームを示す。 1コマ50ミリ秒。
動画2: G1期(左)、G2期(右)のヌクレオソームのゆらぎの動画。G2期の核(ドットが見える範囲)はG1期の核に比べて大きいが、ヌクレオソームのゆらぎにはあまり違いはない。 1コマ50ミリ秒。
▶ EurekAlert!の記事はこちら
ユーラシアにおけるハツカネズミの遺伝的多様性を解明
~人類の歴史の解明や基礎医学研究への貢献に期待~
プレスリリース
斎藤研究室・集団遺伝研究室
Insights into Mus musculus population structure across Eurasia revealed by whole-genome analysis
Kazumichi Fujiwara, Yosuke Kawai, Toyoyuki Takada, Toshihiko Shiroishi, Naruya Saitou, Hitoshi Suzuki, Naoki Osada
Genome Biology and Evolution (2022) 14, evac068 DOI:10.1093/gbe/evac068
北海道大学大学院情報科学研究院の長田直樹准教授と同大学大学院情報科学院博士後期課程の藤原一道氏らの研究グループは、同大学大学院地球環境科学研究院、国立遺伝学研究所、理化学研究所バイオリソース研究センター、国立国際医療研究センターとの共同研究により、ユーラシア大陸の幅広い地域から集められた野生ハツカネズミの全ゲノム配列を決定し、その遺伝的多様性を明らかにしました。
ハツカネズミは医学研究において用いられる代表的な実験動物ですが、南アジア周辺に生息していた野生の集団が起源となっており、主要な三つの亜種が存在すると考えられています。野生のハツカネズミはその後、アフリカからやってきた人類とともに生息域を広げ、特に1万年ほど前から始まる農耕の発展以降、爆発的に世界中に拡散しました。
本研究では、ハツカネズミの研究に生涯をささげた森脇和郎博士が長年収集してきたサンプルを含む、野生ハツカネズミ98個体の全ゲノム配列を決定し、ユーラシア大陸においてどのように三つの亜種が分布しているのか、また、過去にどのような集団動態を経て野生ハツカネズミ集団が成立してきたのかについて明らかにしました。このような研究は、農耕の発展以降、人類がどのようにユーラシア大陸を移動したのかという疑問に答えるのに役立つだけでなく、医学研究で用いられる実験動物がどのような遺伝的変異をもっているか明らかにすることで、実験結果の解釈や再現性に貢献することが期待されます。
本研究は文部科学省科学研究費補助金新学術領域(複合領域)「ゲノム配列を核としたヤポネシア人の起源と成立の解明」(18H05505)研究費の支援を受けて行われました。
なお、本研究成果は、2022年5月7日公開のGenome Biology and Evolution誌にオンライン掲載されました。
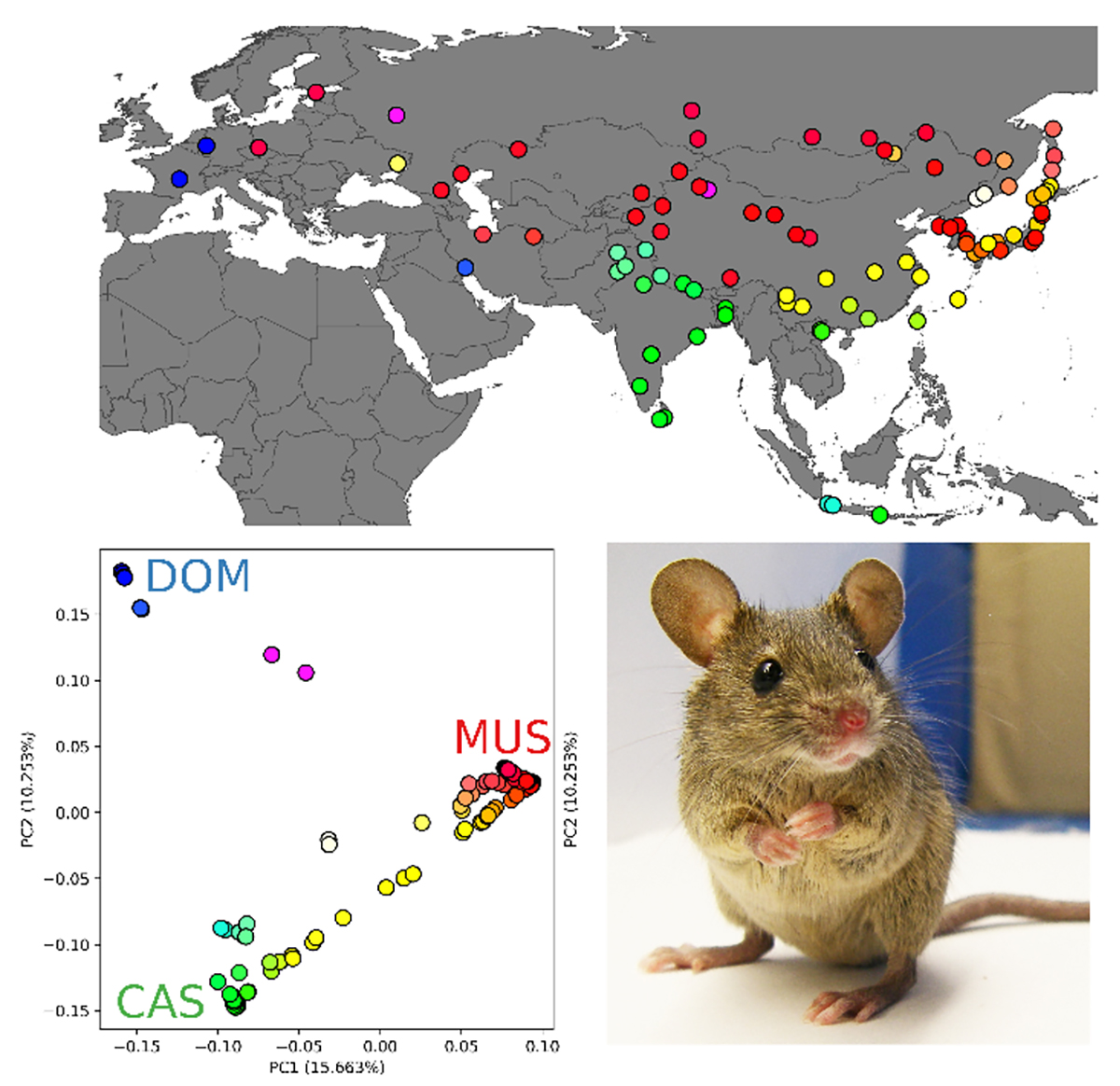
(上)解析に用いられたサンプルの採取地を地図上に示したもの。色は左下のパネルに対応。(左下)主成分分析という手法を用いてゲノムの特徴を可視化したもの。三角形の頂点がそれぞれの亜種がもつ遺伝的特徴に対応。(右下)野生ハツカネズミ由来の実験系統個体(HMI/Ms)















