Archive
- Home
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
ゲノムダイナミクス研究室 総研大生・飯田さんが 米国MBL・Analytical and Quantitative Light Microscopy courseを修了しました
ゲノムダイナミクス研究室 飯田史織さん(総研大遺伝学専攻D4・学振特別研究員DC2)が、米ウッズホール・Marine Biological Laboratory にて開催された、Analytical and Quantitative Light Microscopyコース(4月26日〜5月5日)への参加が認められ、コースを修了しました。このコースはMarine Biological Laboratoryにて毎年開催されている、光学顕微鏡に関する歴史ある教育コースです。
本コース参加にあたって、総研大遺伝学コースの森島奨学寄付金の援助を受けました。

とロブスターが描かれたAnalytical and
Quantitative Light Microscopy 2023
のTシャツを着ています。
擬似的なヒト腸内細菌群集を作製し様々な解析手法を評価
![]()
Assessment of metagenomic workflows using a newly constructed human gut microbiome mock community
Hiroshi Mori*, Tamotsu Kato*, Hiroaki Ozawa, Mitsuo Sakamoto, Takumi Murakami, Todd D. Taylor, Atsushi Toyoda, Moriya Ohkuma, Ken Kurokawa, Hiroshi Ohno
*equal contribution
DNA Research 2023 May 31 DOI:10.1093/dnares/dsad010
腸内細菌群集はヒトの健康に重要なので研究が活発におこなわれています。これらの研究では、DNA抽出や配列データの情報解析などさまざまな解析手法が開発されています。こういった研究の活用で特に問題となるのが「腸内細菌群集の個人差」です。個人差のある研究対象について、個々の研究成果からより一般性の高い結論を得るためには、複数の研究から得られたデータを統合的に解析する「メタ解析」が重要になります。メタ解析をおこなう上で一番の課題は、異なる研究成果間の実験上のバイアスの存在であり、バイアスを定量するためには、群集を構成する細菌のメンバーリストや各メンバーの混合割合があらかじめわかったヒト腸内細菌群集の「模擬群集」が必要不可欠です。
今回、情報・システム研究機構 国立遺伝学研究所の森宙史准教授、理化学研究所の大野博司チームリーダーが率いる研究チームは、ヒト腸内細菌群集の模擬群集を作製し、その模擬群集を用いて様々な解析手法を評価しました。
本研究チームは、ヒト腸内細菌群集中で最も多く存在する細菌の種の中から、代表的な18種18株を選び等量混合して模擬群集を作成しました。この18株のDNAを定量するために、信頼性の高いqPCR法を開発しました。この模擬群集とqPCR法によるDNA定量法を用いて実際の各菌株の混合割合を定量した上で、ヒト腸内細菌群集の研究で広く用いられている10種類のDNA抽出法、7種類の16S rRNA遺伝子のプライマーセット、ショットガンメタゲノムの4種類の情報解析手法のそれぞれについて、菌株組成の推定結果に与える実験上のバイアスを評価しました。
本研究成果は、異なる解析方法で得られたヒト腸内細菌群集の系統組成データを比較解析するための重要な基盤データを提供し、新たな解析手法の開発を促進することが期待できます。
本研究は、日本医療研究開発機構 革新的先端研究開発支援事業(AMED-CREST) (JP19gm1010006)、革新的先端研究開発支援事業(PRIME) (JP19gm6010007)の支援を受けておこなわれました。
本研究成果は、国際科学雑誌「DNA Research」に2023年5月31日(日本時間)に掲載されました。
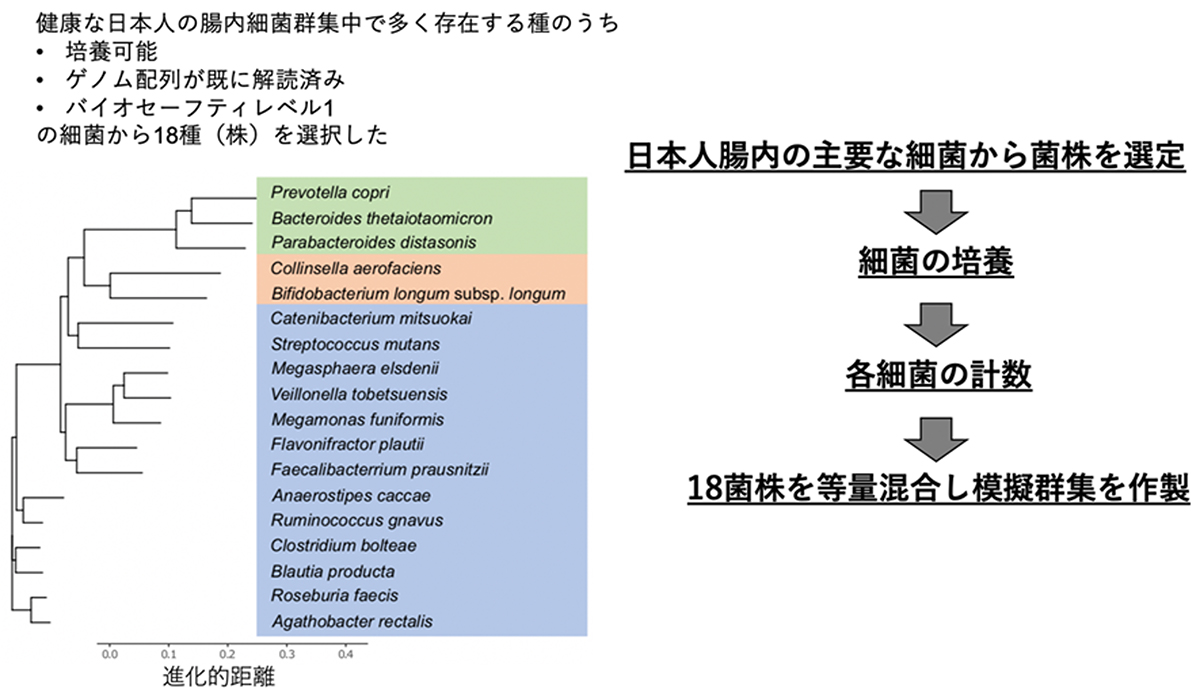
図: ヒト腸内細菌群集の模擬群集の作製方法。
系統樹の色枠は各系統が属する門を表しており、それぞれ緑枠がBacteroidota、橙枠がActinomycetota、青枠がBacillotaになります。
植物の減数分裂運命決定機構に関する総説を発表
Genetic Regulation of Mitosis–Meiosis Fate Decision in Plants: Is Callose an Oversighted Polysaccharide in These Processes?
Harsha Somashekar and Ken-Ichi Nonomura
Plants (2023) 12, 1936 DOI:10.3390/plants12101936
減数分裂は、受精に向けて染色体数を半減させる特殊な細胞分裂です。その過程では、両親のゲノム情報がシャッフルされて新たな遺伝子組み合わせが生み出され(減数分裂組換え)、次世代に伝達されます。減数分裂組換え機構は真核生物で広く保存されており、盛んに研究がされています。一方、生殖細胞が減数分裂に移行するための機構は生物種ごとに多様であり、特に植物では研究が進んでいません。
本総説では、主に顕花植物を対象に、減数分裂細胞を生み出す元となる細胞(胞子原基細胞)がどのように発生・増殖し、減数分裂に至るのかについて、最新の知見を交えて解説しました。植物の細胞壁成分のひとつであるカロース多糖が、雄性減数分裂の適切な開始タイミングと進行の制御に必須の役割を果たすという私たちの研究グループの成果も取り上げています。
なお、本総説の図を用いたイメージ画像は、143論文が掲載されているPlants 第12巻10号の表紙を飾りました。
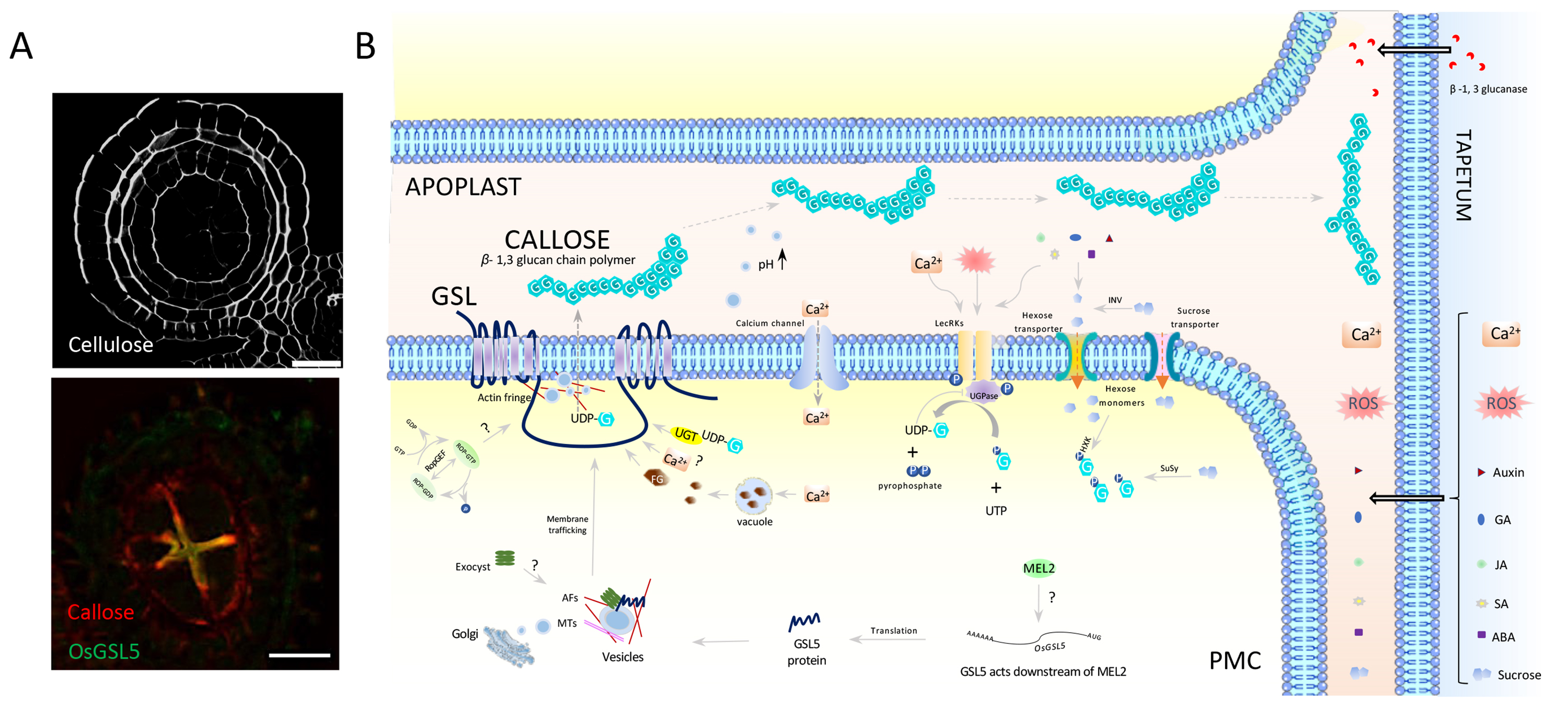
図:葯室におけるカロース合成・輸送モデル
(A) 減数分裂直前の葯の輪切り切片。(上)セルロース染色像。(下)カロース(赤)とGSL5(黄)の同時抗体染色。バーは20µm。(B) 雄性の減数分裂細胞(PMC)の細胞膜にアンカーされるカロース合成酵素(GSL)は、減数分裂移行の直前に、合成したカロースを隣接するPMCの間(APOPLAST)にカロースを放出する。カロースは細胞間隙を移動し、GSLが存在しないPMCとタペート細胞(TAPETUM)の細胞間隙に運ばれ、葯室内の全てのPMCを包み込む。
消えるのか?残るのか?
〜遺伝子の運命は周辺のゲノム環境に左右される~
![]()
The Impact of Local Genomic Properties on the Evolutionary Fate of Genes
Yuichiro Hara, Shigehiro Kuraku
eLife (2023) 12, e82290 DOI:10.7554/eLife.82290
生物の生存に欠かせない機能を果たす遺伝子は多様な生物で共有されています。これは祖先の生物でも生存に重要であった遺伝子が現生の生物に受け継がれたためです。一方で、機能が必要ではなくなると、その遺伝子がなくても個体は生存できるため、遺伝子は進化の過程で失われることがあります。
これまで、進化において遺伝子が失われることは、「必要でなくなれば無くてよい」、すなわち、遺伝子機能の要不要という側面で議論されてきました。一方で、遺伝子はゲノムと呼ばれる遺伝情報の一部であり、ゲノムの情報はDNAという高分子にコードされているため、遺伝子が失われる現象はその遺伝子がコードされる領域のDNAの特徴にも影響をうける可能性があります。しかしながら、遺伝子が失われることに対して遺伝子機能以外の要因はわかっていませんでした。
東京都医学総合研究所の原雄一郎主席研究員(研究当時 国立研究開発法人理化学研究所 生命機能科学研究センター 客員研究員)と情報・システム研究機構 国立遺伝学研究所の工樂樹洋教授(研究当時 国立研究開発法人理化学研究所 生命機能科学研究センター チームリーダー、現客員研究員)による研究チームは、進化の過程で「消えやすい」遺伝子をヒトゲノムに多数同定し、消えやすい遺伝子がもつ機能面以外の特徴を見出すことに挑みました。その結果、「消えやすい」遺伝子は、1)GC含量が高く、2)塩基置換が高頻度であり、3)発現が抑制されている、という特徴を持つことを見つけました。さらに、消えやすい遺伝子の特徴は、脊椎動物の多様な種における相同なゲノム領域で共有されていることがわかりました。
これらの結果から、進化における遺伝子の消えやすさには、遺伝子機能の要不要だけではなく、その遺伝子が存在するゲノム領域の特徴が大きく影響していることが明らかになりました。すなわち、ゲノム領域の特徴は、遺伝子の存亡という進化の出来事に関わっているのです。また、遺伝子の存亡を決めるゲノム領域の特徴が、ヒトとサメの共通祖先の時代から数億年にわたって保持されていて、特定の遺伝子が継続して消えやすい状態にあることもわかりました。進化において同じ遺伝子が色々な系統で失われるという現象は、このようにゲノムの不均一性に由来していると考えられます。
本研究は、以下の支援を受けて行われました。
理化学研究所(工樂)
科研費(20H03269 (工樂), 21K06132 (原))
日本医療研究開発機構(AMED)(JP21wm0325050 (原))
持田記念医学薬学振興財団 2019年度研究助成金 (原)
本研究は、情報・システム研究機構 国立遺伝学研究所が有する遺伝研スーパーコンピュータシステムを利用しました。
本研究成果は、国際科学雑誌「eLife」に 2023年5月25日(日本時間)に掲載されました。

図: 「消えやすい」遺伝子の特徴
イトヨの形態と水質・気温との相関を検証
Plate reduction in southern Japanese freshwater populations of threespine stickleback (Gasterosteus aculeatus)
Kanbe, H., Hosoki, T. K., Kokita, T., Mori, S., and Kitano, J.
Ecology and Evolution (2023) 13, e10077 DOI:10.1002/ece3.10077
系統の離れた分類群で類似の表現型が進化することを収斂進化あるいは平行進化といいます。トゲウオ科のイトヨでは、淡水に進出した集団は体の側面を覆う鱗板という骨化組織が退縮することが知られており、これは、欧米各地で独立に生じていることから平行進化の例として知られています。しかし、日本では、鱗板の退縮した淡水集団は少数あるものの、ほとんどの集団では退縮が生じておらず、その要因は謎でした。
このたび、生態遺伝学研究室の総研大生・神部飛雄さんと北野潤教授らの研究チームは、日本における淡水集団の鱗板変異と相関のある環境要因を調べました。その結果、鱗板退縮は、欧米の集団で報告のあるような低いカルシウム濃度や水の濁りとは相関がなく、温暖な本州南部でのみ生じていることがわかりました。欧米のイトヨ生息地に比して、日本の生息地のカルシウム濃度は全体的に比較的高く、水も比較的透明であることが、日本の淡水集団における鱗板の退縮が生じない1つの原因であると推察されました。
緯度や水温が、どのように鱗板退縮に影響しているのかについて今後も研究を続けます。
本成果はEcology and Evolution誌に発表されました。

図:日本列島では、岐阜県など南方の淡水集団のみ鱗板が退縮している。細木拓也氏が撮影。
六量体複製ヘリカーゼMCM2–7を効率よく組み立てる意義とは?
―複製ライセンシングチェックポイントを説明する新仮説―
The assembly of the MCM2–7 hetero-hexamer and its significance in DNA replication.
Yuki Hatoyama and Masato T. Kanemaki.
Biochemical Society Transactions 2023 May 5 DOI:https://doi.org/10.1042/BST20221465
細胞が増殖をするには、遺伝情報をコードする染色体DNAを複製しなければなりません。染色体DNAを複製する際には、その二重らせんを引き離して一本鎖にする複製ヘリカーゼであるMCM2–7が必要不可欠です。この複製ヘリカーゼMCM2–7はリング状の六量体を形成しています。
英国生化学会が出版する総説雑誌Biochemical Society Transactionsに掲載された本総説では、MCM2–7六量体がどのようなメカニズムにより細胞内で効率よく組み立てられるのか、またその生理学的意義について、我々の論文報告をもとに議論しました。本総説ではMCMBPがシャペロンとして作用することで、六量体MCM2–7の組み立てを促進しており、その結果DNA複製前にMCM2–7が十分量DNA上へ供給されることで、細胞のゲノム完全性につながっているというモデルを提唱しています(図)。
染色体に結合したMCM2–7が不足していると、細胞はそれを感知して細胞周期をG1期に停止させる複製ライセンシングチェックポイントの存在が提唱されています。本総説で細胞はMCM2–7の不足を直接感知しているのではなく、MCM2–7の発現量が低下した細胞ではDNA複製過程でDNA障害が生じており、次のG1期においてそのDNA障害を検知しているのではないかという新仮説を提唱しました。
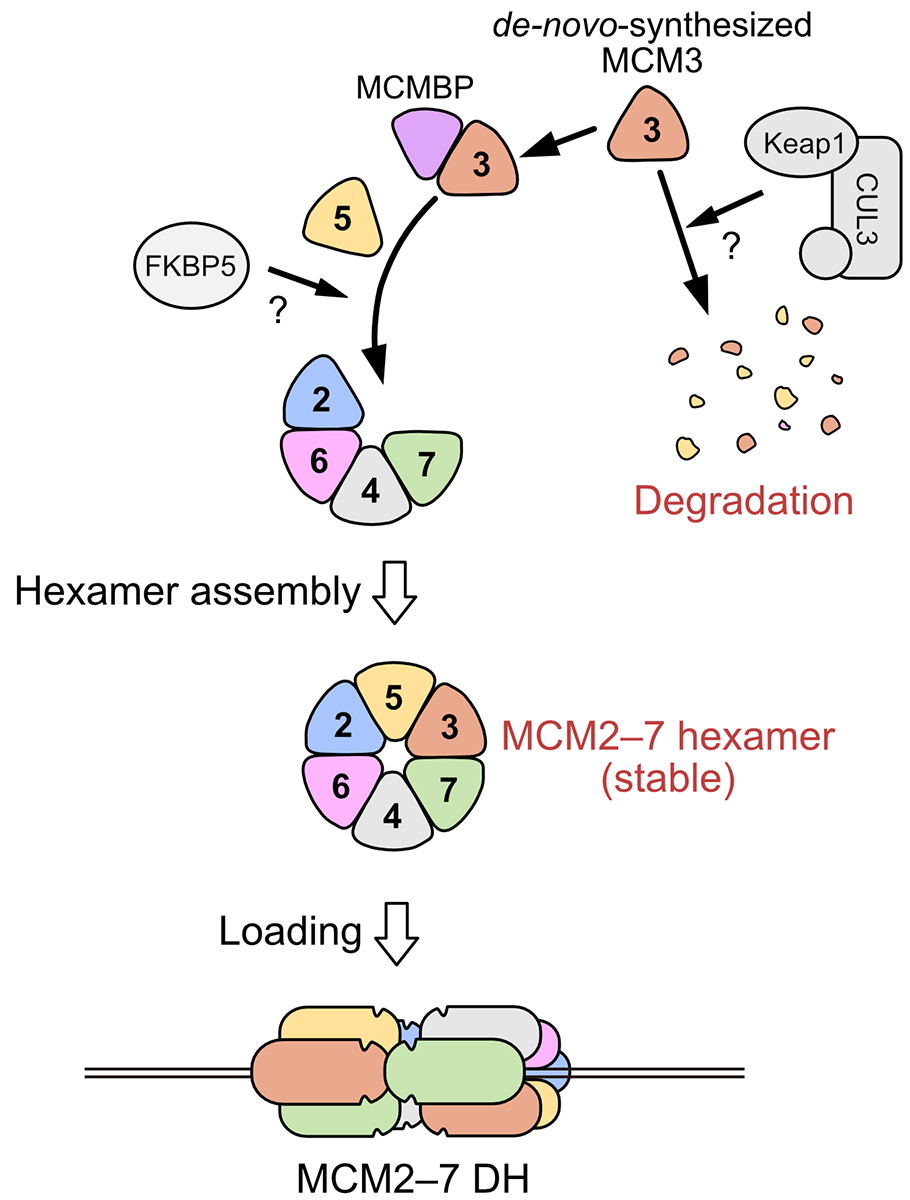
図:MCM2–7複合体の細胞内組み立て機序のモデル図。MCMBPはMCM3と結合しその安定性を保ち、さらにMCM2,4,6,7複合体への組み込みを助けることで、安定的な6量体であるMCM2–7を効率よく組み立てる。
2023年度夏に実施する入学者選抜試験に関する重要なお知らせ
2023年度夏に実施する入学者選抜試験における英語能力評価に関するお知らせ
倉田のり遺伝研名誉教授が「みどりの学術賞」を受賞しました

情報・システム研究機構 国立遺伝学研究所 倉田のり名誉教授が「みどりの学術賞」を受賞することが2023年3月10日に発表され、4月28日に東京で「みどりの式典(授賞式)」がおこなわれました。
「みどりの学術賞」は、国内において植物、森林、緑地、造園、自然保護等に係る研究、技術の開発その他「みどり」に関する学術上の顕著な功績のあった個人に内閣府から授与される賞です。
▶ 功績概要: 「イネのゲノム情報基盤の確立と生殖・多様性研究」に関する功績
▶ 倉田のり名誉教授の経歴と功績概要 (pdf)
▶ みどりの学術賞

内閣府提供
マウス開発研究室 短時間雇用職員(技術補佐員)募集
マウス開発研究室では以下の業務を担当していただける短時間雇用職員(技術補佐員)を募集いたします。
| 【採用時期】 | 2023年6月以降(応相談) |
| 【職務内容】 | ゲノム編集マウス作製とその解析実験の補佐(体外受精・胚操作・DNA解析など分子遺伝学的解析) |
| 【募集人員】 | 1名 |
| 【勤務地】 | 静岡県三島市谷田1111 国立遺伝学研究所 |
| 【応募資格】 | マウスを用いたゲノム編集の経験があるものが望ましいが、未経験でも分子生物学の知識と経験があり、技術習得の意欲があること (防疫の為、げっ歯類・ウサギ類を自宅で飼育されている方はご遠慮下さい) |
| 【給与・待遇】 | 遺伝研の規定に準ずる |
| 【勤務時間】 | 週5日勤務 (月曜日から金曜日)9:00-16:00(昼休憩1時間)応相談 |
| 【休日】 | 土曜、日曜、祝日、12/29~1/3 |
| 【雇用期間】 | 年度契約、評価に基づき更新可。 |
| 【応募】 | 封筒またはメール件名に「マウス開発研究室 技術補佐員応募」と明記の上、履歴書(写真添付・メールアドレス記載)を下記宛先まで郵送またはメールでお送りください。 書類選考通過者のみ面接を行い、採用を決定いたします。 |
| 【応募締切】 | 適任者が見つかり次第、締め切ります。 |
| 【備考】 | 車通勤可。 提出いただいた書類は、情報・システム研究機構個人情報保護規定に則り厳重に管理し、採用審査の用途に限り使用されます。 これらの個人情報は正当な理由なく第三者への開示、譲渡及び貸与することはありません。 お送りいただいた履歴書は返却いたしませんので、ご了承ください。 |
【問合先・提出先】
〒411-8540 三島市谷田1111
大学共同利用機関法人 情報・システム研究機構
国立遺伝学研究所 マウス開発研究室 tel: 055-981-5843
担当:小出
、室伏
組換え因子RAD51はCMGヘリカーゼの後ろでフォーク反転構造形成を促進する
RAD51 bypasses the CMG helicase to promoter fork reversal.
Liu W, Saito Y, Jackson J, Bhowmick R, Kanemaki MT, Vindigni A and *Cortez D.
Science (2023) 380, 382-387 DOI:10.1126/science.add7328
複製フォークはDNA障害などにより停止することが知られており、停止した複製フォークの電子顕微鏡観察からフォーク反転構造(ニワトリ足構造とも呼ばれる)が起きることが観察されています。また、このフォーク反転構造形成にはRAD51組換えタンパク質が必要であることが知られていました。しかしながら、反転フォークにおいて、それまで複製フォークを動かしていたCMGヘリカーゼが残っているのか、そうであればどこに居るのかわかっていませんでした。本研究では、 RAD51変異体の解析から、反転フォークはCMGヘリカーゼの後ろで形成されること、さらにはCMGヘリカーゼがなくなると、反転フォーク形成にRAD51の要求性がなくなることを見出しました。
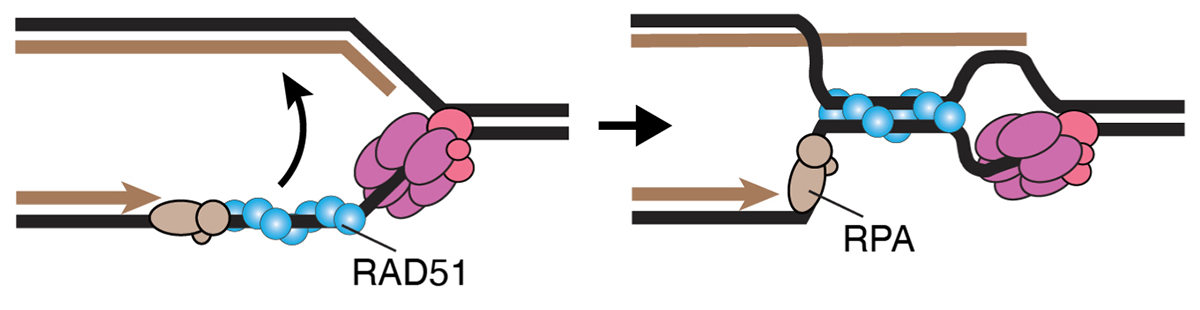
本研究はDavid Cortez教授(Vanderbilt University, USA)と鐘巻研究室の共同研究として行われました。鐘巻研究室はCMGヘリカーゼを構成するMCMタンパク質を分解できるAID2細胞を作成しました。















