野々村研究室・植物細胞遺伝研究室
植物の生殖発生制御メカニズムの分子細胞遺伝学
教員
Research Summary
減数分裂は、受精に向けて染色体数を半減させ、遺伝情報を安定的に子孫に伝達するための特殊な細胞分裂です。また、減数分裂組み換えを通じて遺伝的多様性の創出に貢献しており、その性質は動植物の品種改良にも活用されています。私たちは、減数分裂を含む植物の生殖発生を制御する分子メカニズムの解明を目指しています。
また、遺伝研が保有する植物栽培施設を利用し、野生イネ系統や栽培イネ実験系統を主体としたイネ科植物リソースの保存・提供・栽培受託事業を通じて、植物分野の共同利用研究をサポートしています。
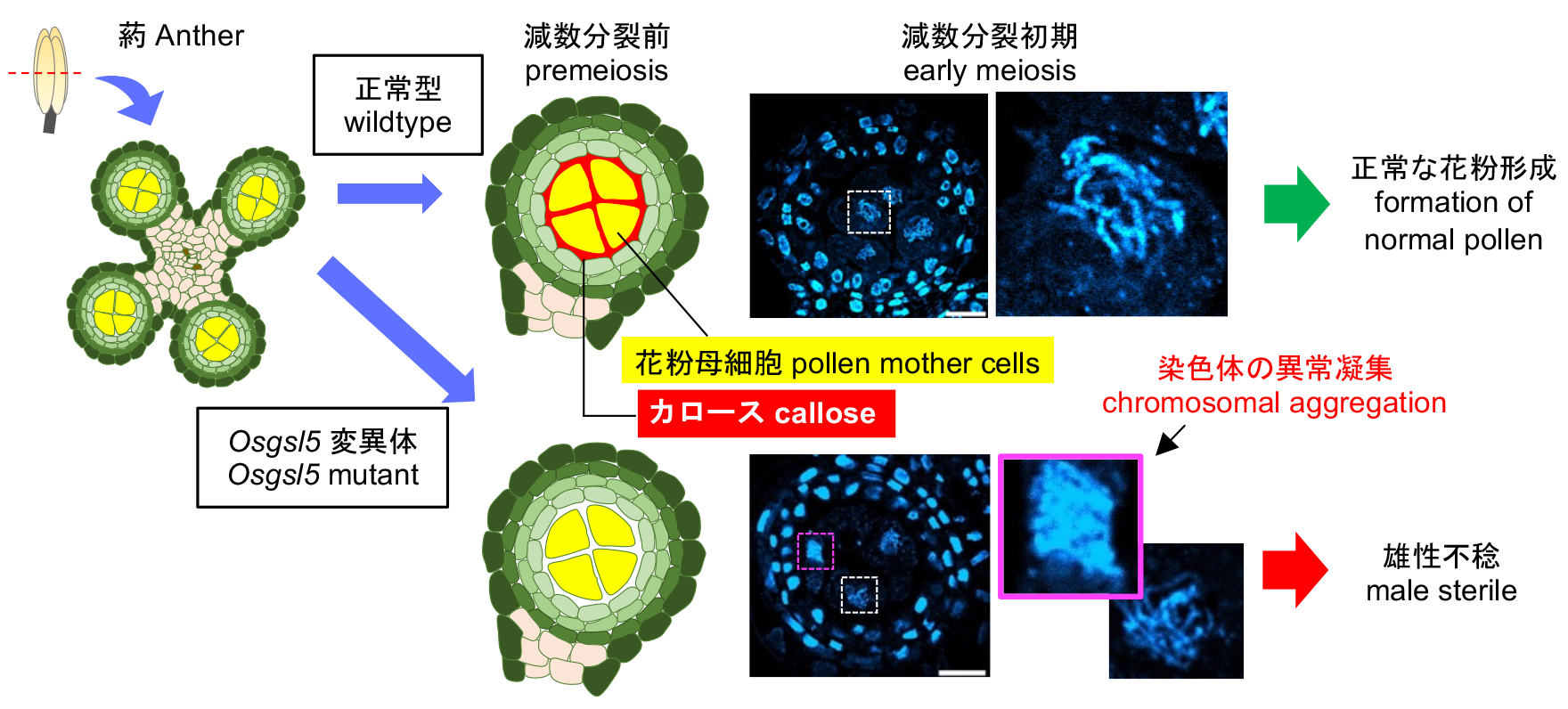
多くの顕花植物の葯では、減数分裂の直前に細胞壁要素のひとつカロースが高蓄積しますが、その生物機能は不明でした。イネのカロース合成酵素OsGSL5の変異体解析から、減数分裂の直前のカロース蓄積は、雄性減数分裂の開始・進行に必須の役割をもつことがわかりました(主要論文1の成果)。
研究活動の紹介動画
Selected Publications
Somashekar H, Mimura M, Tsuda K, Nonomura KI. Rice GLUCAN SYNTHASE-LIKE5 promotes anther callose deposition to maintain meiosis initiation and progression. Plant Physiol. 2023 Jan 2;191(1):400-413.
Tsuda K, Suzuki T, Mimura M, Nonomura KI. Comparison of constitutive promoter activities and development of maize ubiquitin promoter- and Gateway-based binary vectors for rice. Plant Biotechnol (Tokyo). 2022 Jun 25;39(2):139-146.
Ishikawa R, Castillo CC, Htun TM, Numaguchi K, Inoue K, Oka Y, Ogasawara M, Sugiyama S, Takama N, Orn C, Inoue C, Nonomura KI, Allaby R, Fuller DQ, Ishii T. A stepwise route to domesticate rice by controlling seed shattering and panicle shape. Proc Natl Acad Sci U S A. 2022 Jun 28;119(26):e2121692119.
Ono S, Liu H, Tsuda K, Fukai E, Tanaka K, Sasaki T, Nonomura KI. EAT1 transcription factor, a non-cell-autonomous regulator of pollen production, activates meiotic small RNA biogenesis in rice anther tapetum. PLoS Genet. 2018 Feb 12;14(2):e1007238. doi: 10.1371/journal.pgen.1007238. Erratum in: PLoS Genet. 2019 Mar 6;15(3):e1008033.

















