森研究室・ゲノム多様性研究室
ゲノムの多様性と環境との相互作用の解明
教員
Research Summary
生物は多様なゲノムを持ち、様々な環境に適応しております。我々の研究室では、コンピュータを用いた比較ゲノム解析やメタゲノム解析等の手法によって、数万年前に絶滅した動物の古いDNAからゲノム配列情報を取得し進化系統樹を推定する研究や、メタゲノム配列から新規性の高い微生物の高精度なゲノム配列を構築してデータベース化する研究等に取り組んでおります。また、ゲノム・メタゲノム解析を高度化するための様々な情報解析技術・ツールの開発を行い、先端ゲノミクス推進センターと強く連携して、開発した情報解析技術を活かした国内外の研究機関との多数の共同研究を進めております。
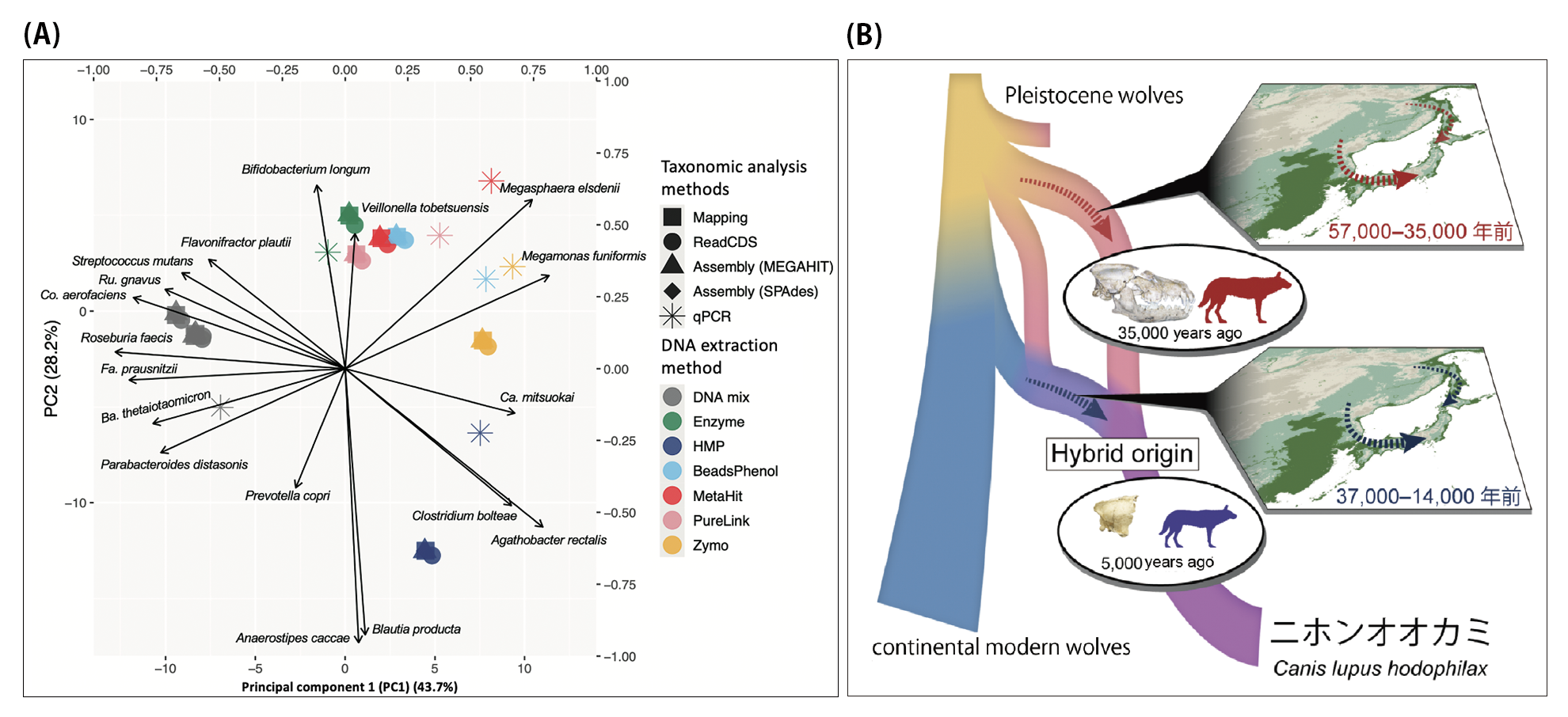
(B)3万5千年前の日本の更新世オオカミのAncient DNA解析によって推定されたニホンオオカミの系統の成立過程。我々の研究から、巨大な更新世オオカミが日本に移入した後、別の系統のオオカミが移入しそれらが交雑してニホンオオカミが成立した進化史が推定された。
研究活動の紹介動画
Selected Publications
Mori H, Ishikawa H, Higashi K, Kato Y, Ebisuzaki T, Kurokawa K. PZLAST: an ultrafast amino acid sequence similarity search server against public metagenomes. Bioinformatics. 2021 Jul 7;37(21):3944–3946.
Segawa T, Yonezawa T, Mori H, Akiyoshi A, Allentoft ME, Kohno A, Tokanai F, Willerslev E, Kohno N, Nishihara H. Ancient DNA reveals multiple origins and migration waves of extinct Japanese brown bear lineages. R Soc Open Sci. 2021 Aug 4;8(8):210518.
Segawa T, Yonezawa T, Mori H, Kohno A, Kudo Y, Akiyoshi A, Wu J, Tokanai F, Sakamoto M, Kohno N, Nishihara H. Paleogenomics reveals independent and hybrid origins of two morphologically distinct wolf lineages endemic to Japan. Curr Biol. 2022 Jun 6;32(11):2494-2504.e5.
Mori H, Kato T, Ozawa H, Sakamoto M, Murakami T, Taylor TD, Toyoda A, Ohkuma M, Kurokawa K, Ohno H. Assessment of metagenomic workflows using a newly constructed human gut microbiome mock community. DNA Res. 2023 Jun 1;30(3):dsad010.
















