Archive
- Home
- 2026/3
- 2026/2
- 2026/1
- 2025/12
- 2025/11
- 2025/10
- 2025/9
- 2025/8
- 2025/7
- 2025/6
- 2025/5
- 2025/4
- 2025/3
- 2025/2
- 2025/1
- 2024/12
- 2024/11
- 2024/10
- 2024/9
- 2024/8
- 2024/7
- 2024/6
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
2018年度「先進ゲノム支援」による支援課題の公募を開始
2018年度「先進ゲノム支援」による支援課題の公募を開始
シンポジウム「遺伝学の最先端研究から見る生命誌」を開催(5月19日)
染色体生化学研究室 村山准教授が平成30年度文部科学大臣表彰 若手科学者賞を受賞

村山准教授(受賞式後)
新分野研究センター染色体生化学研究室の村山准教授が平成30年度文部科学大臣表彰 若手科学者賞を受賞しました。
本賞は、萌芽的な研究、独創的視点に立った研究等、高度な研究開発能力を示す顕著な研究業績をあげた40歳未満の若手研究者に授与されるものです。
授賞式日時:平成30年4月17日(火)
授賞式場所:文部科学省3階 講堂
受賞名:平成30年度文部科学大臣表彰 若手科学者賞
受賞テーマ:コヒーシン複合体による姉妹染色体接着形成の分子機構の研究
平成30年度科学技術分野の文部科学大臣表彰受賞者等の決定について
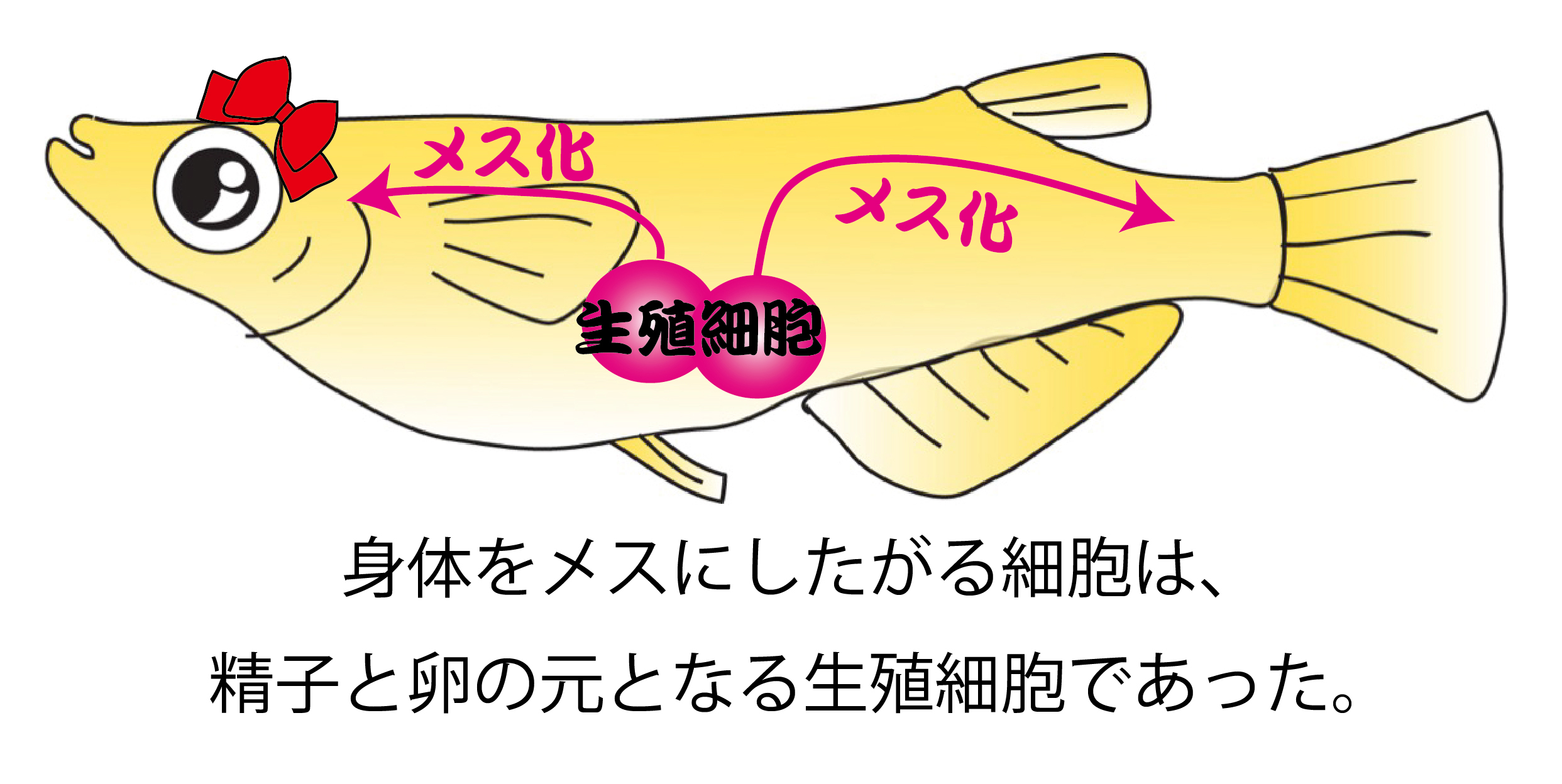
どんなときでも身体をメスにしたがる細胞:生殖細胞 〜この特質によって身体はメスになる〜
![]()
Germ cells in the teleost fish medaka have an inherent feminizing effect
Toshiya Nishimura, Kazuki Yamada, Chika Fujimori, Mariko Kikuchi, Toshihiro Kawasaki, Kellee R. Siegfried, Noriyoshi Sakai, Minoru Tanaka
PLoS Genetics Published: March 29, 2018 DOI:10.1371/journal.pgen.1007259
名古屋大学大学院理学研究科の 田中 実 教授、西村 俊哉 助教の研究グループは、国立遺伝学研究所の 酒井 則良 准教授のグループ及びUniversity of Massachusetts BostonのKellee Siegfried博士との共同研究により、身体をメスにしたがる特質の細胞がいることを、メダカを利用した実験において見出しました。
哺乳類もメダカもY染色体を持っていると身体はオスになります。ところが、「生殖細胞」は身体がY染色体を持っていようがいまいが、もともと、身体をメスにしたがる働きを持っているだけでなく、この特質がないとメダカはメスにはなれないこともわかりました。「生殖細胞」は精子と卵(配偶子)の元となる細胞、すなわち、子孫を作り出すのに必須の細胞なのです。生殖細胞は、精子と卵のどちらにもなれる能力を持っています。興味深いことに、このメスにさせる特質は、生殖細胞が精子もしくは卵のどちらになるのかが決まる前の状態から発揮され、また「精子になる」と決まった生殖細胞にもこの特質があることがわかりました。
身体をメスにしたがる細胞の特質がわかったことにより、今後、身体の性が決まる仕組みの理解が一層深まると期待されます。
この研究成果は、平成30年3月30日付(日本時間午前3時)米国科学雑誌「PLOS Genetics」オンライン版に掲載されました。
また、この研究は文部科学省科学研究費補助金、住友財団及びノバルティス科学振興財団の支援のもとで行われたものです。
カイコモデル系によるヒト薬物吸収性の評価
Construction of a simple evaluation system for the intestinal absorption of an orally administered medicine using Bombyx mori larvae
Fumika Ichino, Hidemasa Bono, Takeru Nakazato, Atsushi Toyoda, Asao Fujiyama, Kikuo Iwabuchi, Ryoichi Sato, Hiroko Tabunoki
Drug Discoveries & Therapeutics, 12(1) 7-15, 2018 DOI:10.5582/ddt.2018.01004
ヒトの腸管吸収はヒト結腸癌由来の細胞株Caco-2細胞や腸灌流、哺乳類モデルを用いて見積もられていますが、これらの系にはその能力において限界がありました。マウスやラットなどの実験動物によるin vivoの評価系には倫理的な問題があり、また創薬段階における大規模な薬剤スクリーニングが困難となっていました。そこで、東京農工大学農学部 天竺桂弘子講師を中心とするグループは、薬剤腸管吸収の評価にカイコガ(Bombyx mori)幼虫を用いる手法を提案し、さらに経口投与による腸管透過性を評価する系を開発しました。
遺伝学研究所 比較ゲノム解析研究室 豊田敦特任教授、同研究所先端ゲノミクス推進センター 藤山秋佐夫特任教授はカイコ中腸のトランスクリプトーム配列解読を行いました。ライフサイエンス統合データベースセンター(DBCLS) 坊農秀雅特任准教授、仲里猛留特任助教はそのカイコ中腸の遺伝子発現データ解析を行い、公共遺伝子発現データセットから得たCaco-2細胞ならびにヒト小腸の遺伝子発現データとの比較トランスクリプトーム解析をおこなうことで本研究に貢献しました。
本研究は、国立遺伝学研究所が有する遺伝研スーパーコンピュータシステムを利用し、また国立遺伝学研究所公募型共同研究 NIG-JOINT (2014-A171 & 2015-A155)の支援を受けました。
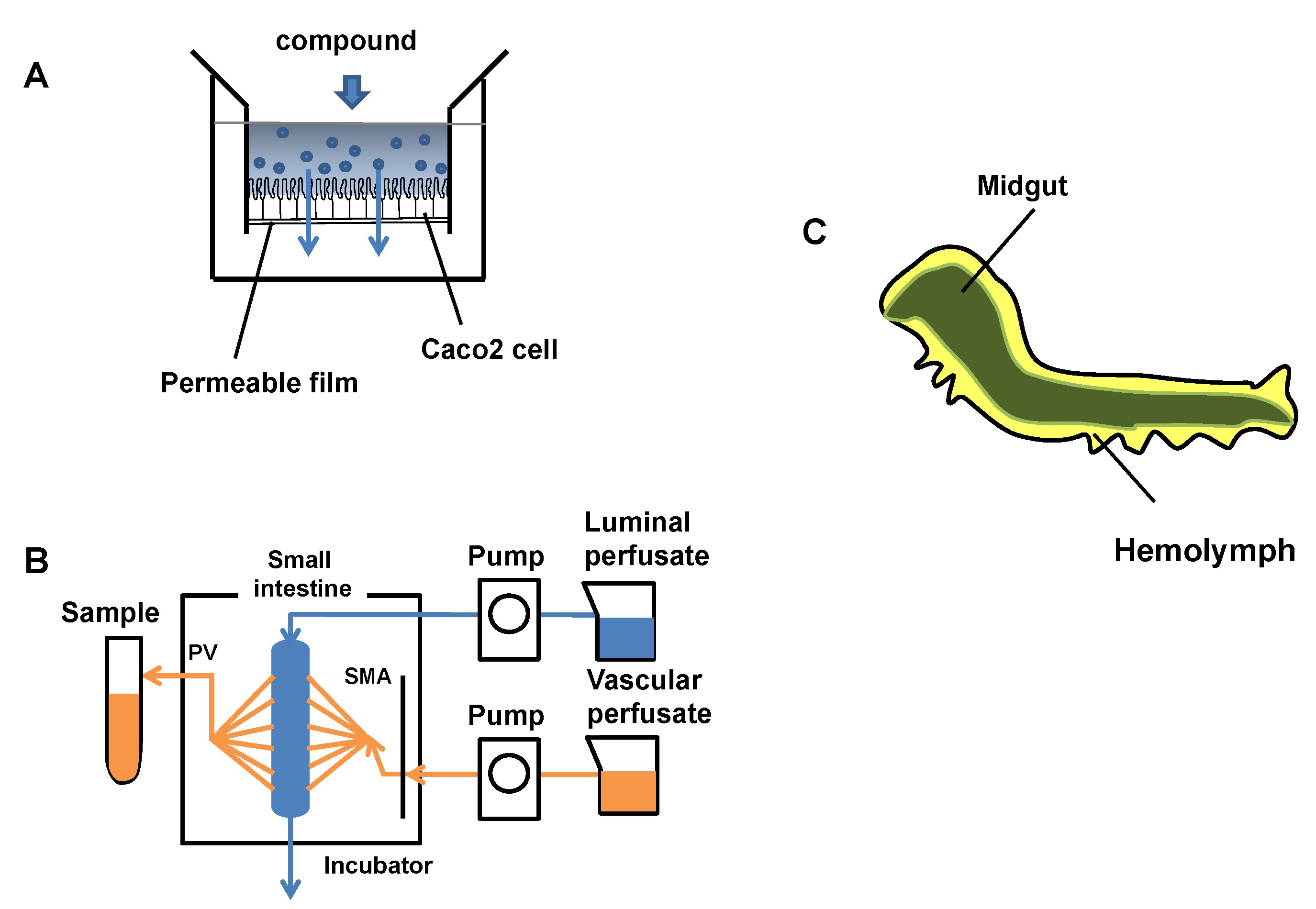
図:腸管透過性のモデル系の評価とカイコガ幼虫の内部構造。体内の大部分が中腸(Midgut)によって占められており(緑色)、それらは血リンパ(Hemolymph)によって取り囲まれている(黄色)。
全国のソメイヨシノの花びらに付着したDNAを解析し、環境中に存在するDNAの由来を調査
Collaborative environmental DNA sampling from petal surfaces of flowering cherry Cerasus × yedoensis ‘Somei-yoshino’ across the Japanese archipelago
Ohta T, Kawashima T, Shinozaki NO, Dobashi A, Hiraoka S, Hoshino T, Kanno K, Kataoka T, Kawashima S, Matsui M, Nemoto W, Nishijima S, Suganuma N, Suzuki H, Taguchi Y, Takenaka Y, Tanigawa Y, Tsuneyoshi M, Yoshitake K, Sato Y, Yamashita R, Arakawa K, Iwasaki W
Journal of Plant Research 2018, Epub ahead of print. DOI:10.1007/s10265-018-1017-x
概要
複数の研究機関から研究者が参加した研究プロジェクト「お花見プロジェクト」によって、ソメイヨシノの花びら表面に付着した環境DNAのサンプルを収集し、DNAの由来を解析しました。プロジェクトはクラウドソーシングのアプローチを採用し、150名以上の協力者を得て、2015年春に全国の577地点からサンプルを収集しました。プロジェクトチームは、収集された環境DNAの塩基配列を高速DNAシーケンサーによって解析し、データベースに登録された配列と比較することで、その由来を調べました。その結果、ソメイヨシノの花びらの表面から、ソメイヨシノ自身のものと思われるDNAの他に、スギを始めとする異なる植物由来と考えられるDNAが存在することがわかりました。本研究プロジェクトは全ての研究データを公開しており、関連する環境DNAの研究にとって貴重な参照データとなることが期待されます。また、短い期間のうちに全国規模でサンプルを集めた世界でも珍しいプロジェクトとして、科学研究におけるクラウドソーシングの有効性を示すことができました。

図1: サンプリングの様子。桜の花びらを綿棒状の採取キットでぬぐうことで、表面に付着する環境DNAを採取する。
背景
生命の設計図とも呼ばれるDNAは、生物の組織や分泌物の飛散によって、環境中のいたるところに存在していることがわかっています。このように特定の生体サンプルではなく、環境中から検出されたDNAは環境DNAと総称され、人工物の表面や生物の体表面からも見つかります。環境DNAの由来を調べることで、そのDNAが見つかった場所が、どのような生物が存在する環境であるかを知ることができます。樹木などの植物の表面にも環境DNAは付着しており、これらを調べることは植物の生育している環境のことを知る手がかりになります。特に花を咲かせる植物において、開花直前まで外界に晒されていない花びら表面に付着する環境DNAを調べることで、開花してからの環境の情報を読み取ることができると考えられます。
ソメイヨシノ (学名 Cerasus × yedoensis ‘Somei-yoshino’) は沖縄県や北部北海道を除く日本全国で見ることのできる桜の一種で、観賞用にも人気のある桜の品種として親しまれています。ソメイヨシノは特定の気候条件が整った際に、一週間程度の短い期間だけ開花するという特徴を持っています。さらに、ソメイヨシノは自家不和合性の品種であり、接木で増やされ全国に広まりました。つまり、全てのソメイヨシノは同じゲノムを持つクローンだということです。そのため、ソメイヨシノの開花後、花びら表面に付着する環境DNAを全国から集めて解析し、比較することによって、環境DNAを通した生息環境の比較を行うことができると考えられます。しかし、開花の期間が短く、また全国の様々な場所で見られるソメイヨシノに付着したDNAのサンプルを大量に得ることは、単独の研究チームでは時間と費用の両面が問題となり、困難でした。そこで、全国に点在する研究者に呼びかけ、ソメイヨシノからDNAをサンプリングしてもらい、集まったサンプルを比較解析する「お花見プロジェクト」が2015年に企画されました。
成果
お花見プロジェクトでは、最新の高速DNAシーケンサーに関する研究交流を行っている「NGS現場の会」を通して、全国の大学や研究所、企業に所属する研究者に参加を呼びかけ、環境DNAのサンプリングを依頼しました。その結果、約150名の協力者を得て、577ものサンプルを集めることに成功しました。サンプルは沖縄県を除く全ての都道府県から届き、2015年の最も早い開花時期であった3月中頃から、最も遅い開花時期であった5月前半のうちにサンプリングが完了しました。協力者には、環境DNAを採取するための綿棒状のキットを送付し、ソメイヨシノの花びらの表面をぬぐうことで環境DNAを採取してもらいました (図1)。
研究チームは、集められた環境DNAサンプルから微生物の系統判別に用いられる 16S rRNA と呼ばれるDNA配列を対象に、高速DNAシーケンサーで配列の解読を行いました。これによって、開花して数日から一週間という短い期間においても、環境DNAが花びらに付着していることが明らかになりました。さらに、得られた塩基配列をデータベースに登録された情報と比較することで、環境DNAが何の生物に由来するものかを調べました。結果として、桜の花びらから様々な生物に由来するDNAが見出されました。特徴的なものとして、スギと思われる植物に由来するDNAが付着しているサンプルが多く見出されました。この結果は、開花後間もないソメイヨシノの花びらに、スギ花粉が飛散し、付着している可能性を示唆しています。しかし同時に、ソメイヨシノの花びらに付着した環境DNAの由来をさらに正確に調べるためには、より精度の高いサンプリング方法と、DNA配列解析の工夫が必要であることもわかりました。
展望・波及効果
本プロジェクトでは、短期間に広範囲の地点から環境DNAのサンプルを得るために、クラウドソーシングを採用し、同手法が科学研究においても重要な手段となり得ることを示しました。さらに、開花後1週間前後という短い期間にしか採取できないサンプルを、日本全国という大規模な範囲から得ることに成功した世界でも例の少ないプロジェクトとなりました。また、同じ条件で開花するクローン植物というユニークな性質をもつソメイヨシノに付着する環境DNAを数多く採取し解析した結果は、関連するプロジェクトにとって優れた参照データになる可能性があります。ソメイヨシノという日本に馴染みの深い植物を通して、環境DNAの解析による様々な研究成果にも目が向けられることを期待しています。
研究プロジェクトについて
お花見プロジェクトは、NGS現場の会 第四回研究会 の主催者チームによって企画されました。同研究会の参加者を通してお花見プロジェクトへの参加者を募り、サンプルの収集を行いました。収集されたDNAの塩基配列解析は研究会主催者チームによって行われました。塩基配列データの解析は、プロジェクト参加者からの希望者によって行われました。塩基配列データや、関連するサンプルの情報は、全て国立遺伝学研究所 DNA Data Bank Japan (DDBJ) に寄託され、一般に公開されています。プロジェクトにおけるサンプリングキットの購入や配布、DNA解析の費用は NGS現場の会 第四回研究会 の運営費から支出されました。データの解析の一部は、遺伝学研究所のスーパーコンピューターシステムを利用しました。
補足情報
Journal of Plant Research に掲載された論文の Fig. 1b において、説明文では “ID 005789” とありますが、これは正しくは “ID 005754” の誤りです。論文のデータの解釈や結論には影響を与えないため、修正 (Erratum) は出版されませんが、この場で訂正させていただきます。
バフンウニのゲノムを解読 〜研究・教育推進のためにゲノムデータベースを公開〜
![]()
HpBase: a genome data base of a sea urchin, Hemicentrotus pulcherrimus
Sonoko Kinjo, Masato Kiyomoto, Takashi Yamamoto, Kazuho Ikeo, Shunsuke Yaguchi
Development Growth & Differentiation DOI:10.1111/dgd.12429
筑波大学 生命環境系 谷口俊介准教授(下田臨海実験センター)は、情報・システム研究機構国立遺伝学研究所 遺伝情報分析研究室 池尾一穂准教授、金城その子研究員、お茶の水女子大学 湾岸生物教育研究センター 清本正人准教授、広島大学 大学院理学研究科 山本卓教授との共同研究により、バフンウニ(Hemicentrotus pulcherrimus)のゲノム配列を解読しました。
バフンウニは北海道南端より南の地域の海岸線でよく見られるウニの一種であり、地域によっては貴重な漁獲対象物となっています。また、その採集のしやすさ、卵や精子といった配偶子取得の容易さから、発生生物学、細胞生物学等の優れた研究材料としてだけでなく、動物の発生を学ぶ教育現場においても、我が国では長い間利用されてきました。本研究では、バフンウニのゲノム配列を解読し、研究・教育の過程で利用できるデータベース「HpBase」(http://cell-innovation.nig.ac.jp/Hpul/)を作成しました。
本研究の成果は2018年3月13付で日本発生生物学会の機関誌「Development Growth & Differentiation」にて先行公開されました。データベースは3月19日公開の予定です。
本研究は、AMEDおよび文部科学省の助成金によって実施されました。
なお、国立遺伝学研究所にて開発した次世代シーケンサーデータの解析ツールを以下のurlにて提供しています。
http://cell-innovation.nig.ac.jp/maser/
参考文献:Kinjo, S. et.al. 2018. Maser: one-stop platform for NGS big data from analysis to visualization. DATABASE. In press.
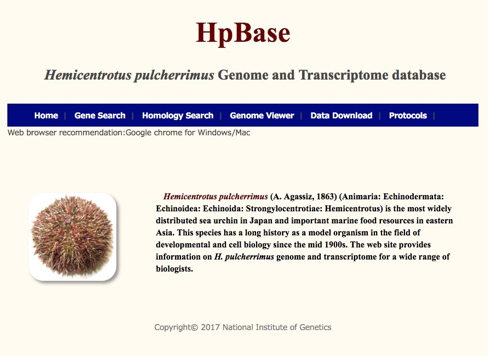
図キャプション:HpBaseのトップページ
生体高分子研究室 今井さん が「森島奨励賞」を受賞

総合研究大学院大学 遺伝学専攻が独自に行っている「森島奨励賞」の選考が、2017年度後期の学位申請者に対して行われ、
生体高分子研究室 前島研究室に所属する今井亮輔さんが受賞しました。
・今井亮輔(生体高分子研究室 前島研究室)
「DNA transactions and physical environment of chromatin」
授与式が2018年3月13日に行われ、桂専攻長から賞状と研究奨励金が贈られました。
森島奨励賞とは
総研大遺伝学専攻で優秀な研究成果を発表して学位を取得した学生に、その研究内容を称えるとともに今後のさらなる発展を促す目的で贈られます。遺伝学の先達
「二つの顔」を持つタンパク質 ~転写促進とメッセンジャーRNAの分解抑制~
![]()
形質遺伝研究部門
Mbf1 ensures Polycomb silencing by protecting E(z) mRNA from degradation by Pacman
Kenichi Nishioka, Xian-Feng Wang, Hitomi Miyazaki, Hidenobu Soejima, Susumu Hirose
Development 2018 145:dev162461 DOI:10.1242/dev.162461
Mbf1はストレス耐性などにかかわる転写因子で、いわば細胞を守る「正義の味方」です。Mbf1は核内で様々なストレス耐性にかかわる遺伝子の転写を促進することがわかっていました。
本成果では、Mbf1が転写促進活性とは異なる新たなメカニズムによってポリコーム抑制やストレス防御に関わっていることを見出しました。細胞には、パックマンと呼ばれる「悪役」酵素が存在し、メッセンジャーRNAを分解しています。このメッセンジャーRNAにMbf1が結合し、パックマンによる分解からメッセンジャーRNAを守っていることがわかったのです。Mbf1によって守られているメッセンジャーRNAが作るタンパク質には、パックマンの発現を抑えるポリコーム抑制やストレス耐性にかかわるものなどがありました。したがって、Mbf1は、ポリコーム抑制を強固にすると共に、「ストレス耐性遺伝子の転写の促進」と「メッセンジャーRNAの分解抑制」という二つの機能によってストレス防御を制御していたのです。
本研究は、情報・システム機構国立遺伝学研究所の広瀬進名誉教授らと佐賀大学医学部の西岡憲一講師らによる共同研究の成果で、科学技術振興機構さきがけ、日本学術振興会科研費と九州大学生体防御研究所機器利用型共同研究プロジェクトの支援を受けておこなわれました。
本研究成果は、英国科学雑誌Developmentに平成30年3月9日(グリニッジ標準時)に掲載されました。
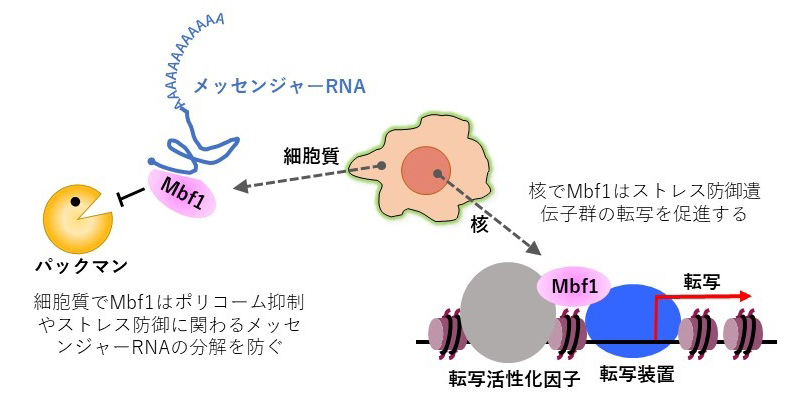
図キャプション:二つの顔を持つMbf1
遺伝研 一般公開2018 : 4月7日(土)開催 – 講演の動画を配信中
大量遺伝情報研究室 藤澤貴智 特任研究員がLODチャレンジ2017 アイデア賞優秀賞を受賞

(JODチャレンジ2017審査委員長)、
DBCLS 山本泰智 特任准教授、
大量遺伝情報研究室 藤澤貴智 特任研究員
大量遺伝情報研究室 藤澤貴智 特任研究員は、DBCLS 山本泰智 特任准教授と連名で、LODチャレンジ2017 において、アイデア賞優秀賞を受賞しました。LOD (Linked Open Data)は、世界中のだれもが意味情報を付与したデータを公開することで、インターネット上を一つの知識ベースとして扱うことを可能にすることのできる仕組みです。LODチャレンジは、そのデータ公開や技術開発に関するコンテストであり、受賞した構想は、データ公開を促進するためのアイデアです。ROIS URAステーションにおいて共同研究打合せ中に生まれ、現在、研究開発の実施を計画しております。
▶ Linked Open Data チャレンジ Japan 2017 受賞作品発表
LODチャレンジ2017は、さまざまな分野でデータ作りやデータの活用にチャレンジされている方々による活動の発表の場を提供するために、新たなデータづくり、データ公開、データ共有の仕掛けやオープンデータ活用のアイディア、アプリケーションなどを「作品」として募集し表彰するものです。
▶ 大量遺伝情報研究室
ウンシュウミカンの全ゲノムを解読 -カンキツ品種改良の効率化に期待-
![]()
Draft sequencing of the heterozygous diploid genome of Satsuma (Citrus unshiu Marc.) using a hybrid assembly approach
Tokurou Shimizu, Yasuhiro Tanizawa, Takako Mochizuki, Hideki Nagasaki, Terutaka Yoshioka, Atsushi Toyoda, Asao Fujiyama, Eli Kaminuma, Yasukazu Nakamura
Frontiers in Genetics 05 December 2017 DOI:10.3389/fgene.2017.00180
1.農研機構は情報・システム研究機構国立遺伝学研究所と共同で、ウンシュウミカン「宮川(みやがわ)早生(わせ)」の全ゲノム配列を解読しました。ゲノムの大きさは約3億6000万塩基対でした。
2.ゲノム配列中に約2万9千個の遺伝子が存在すると推定し、その中からカンキツの着色に関わるカロテノイド生合成の遺伝子や、結実性に関わるジベレリンの生合成遺伝子91個を特定しました。
3.ウンシュウミカンの両親であるキシュウミカンとクネンボの塩基配列も解析して比較したところ、クネンボの片親がキシュウミカンであること、つまり、ウンシュウミカンはキシュウミカンの子供にさらにキシュウミカンが交配されて生まれたことがわかりました。
4.本成果で得られた全ゲノム配列を利用すれば、ゲノムワイド関連解析を利用した果実形質や栽培性に関わる重要遺伝子の機能推定が高速化され、カンキツの品種育成を効率化できると期待されます。
5.本成果は国際科学雑誌「Frontiers in Genetics」に12月5日に掲載されました。
遺伝研の貢献
国立遺伝学研究所 比較ゲノム解析研究室 豊田敦特任教授、同研究所先端ゲノミクス推進センター 藤山秋佐夫特任教授は宮川早生品種のDNA解読をおこない、断片状の塩基配列を生成しました。その断片塩基配列データをもちいて、大量遺伝情報研究室 谷澤靖洋特任研究員、望月孝子特任研究員、神沼英里助教、中村保一教授は、染色体配列を再構築するゲノムアセンブリ解析や遺伝子注釈解析を行いました。解析には遺伝研スーパーコンピュータを使用しました。
研究支援
本研究は、農林水産省委託プロジェクトゲノム情報を活用した農産物の次世代生産基盤技術の開発プロジェクト「多数の遺伝子が関与する形質を改良する新しい育種技術の開発」(NGB)、ROIS新領域融合プロジェクト「生命システム」サブテーマ 1 超大量ゲノム情報(代表:藤山秋佐夫)、科学研究費補助金の支援を受けて実施されました。
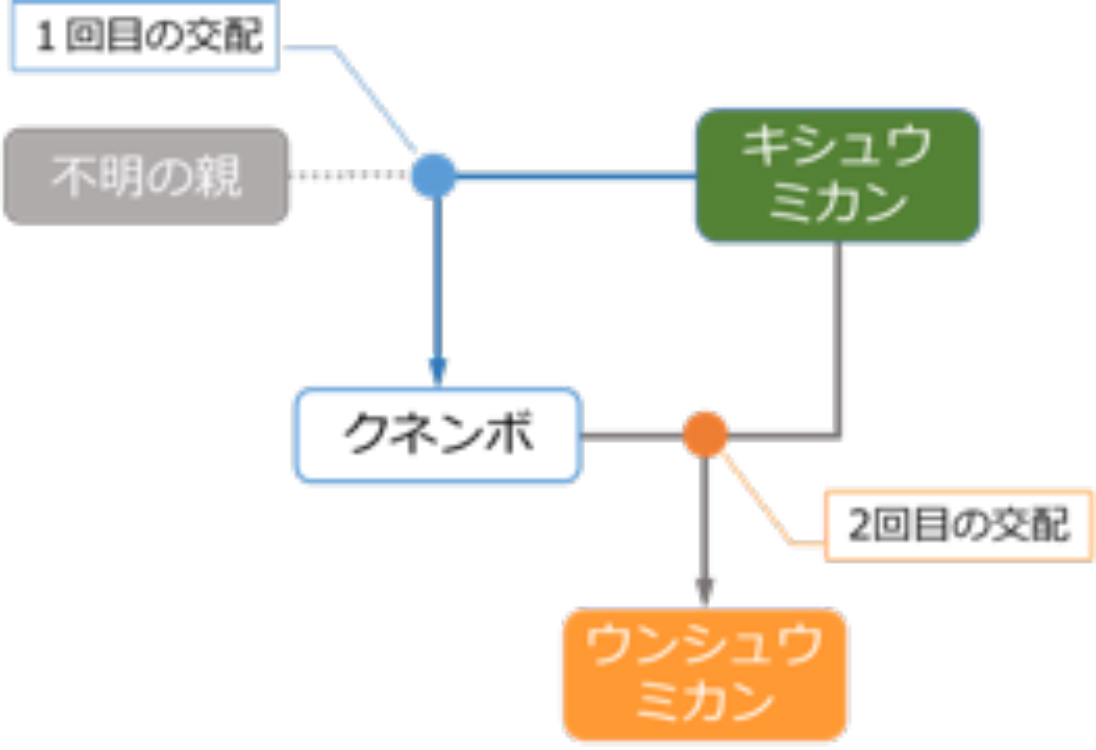
図1.全配列解析から明らかとなったウンシュウミカンの成立経過
ウンシュウミカンの片親であるクネンボはキシュウミカンの子であることを全配列解析から確認しました。すなわち、ウンシュウミカンはキシュウミカンの子供に、さらにキシュウミカンが交配された結果生まれた品種であることがわかりました(クネンボのもう一方の片親は不明です)。

図2:ウンシュウミカン、キシュウミカン、クネンボの果実
プレスリリース資料中でご紹介した成果「DNAの違いから、芽生え段階でカンキツの様々な果実特性を高精度に予測」についてこちらでお読みいただけます
イネの葯組織特異的な小分子RNA生合成を誘導する転写因子を特定
EAT1 transcription factor, a non-cell-autonomous regulator of pollen production, activates meiotic small RNA biogenesis in rice anther tapetum.
Seijiro Ono, Hua Liu, Katsutoshi Tsuda, Eigo Fukai, Keisuke Tanaka, Takuji Sasaki, Ken-Ichi Nonomura.
PLOS Genetics, 14 (2), e1007238, (2018) DOI:10.1371/journal.pgen.1007238
小分子RNAは、遺伝子の転写や翻訳の抑制を介して、個体発生やウィルス防御などで重要な働きをします。私たちは今回、葯を構成するタペート組織で発現するイネEAT1転写因子が、減数分裂期特異的に24塩基長の小分子RNA生産を誘導することを発見しました(図1A, B)。EAT1は、小分子RNAの素材となる前駆体RNA、および前駆体を24塩基長に切断するDCL5酵素をコードする遺伝子の転写を直接活性化していたのです(図1C)。
また、小分子RNAの一部はタペート細胞に隣接する減数分裂細胞へと移動する可能性が、減数分裂細胞で発現するMEL1タンパク質の解析から明らかになりました(図1C)。小分子RNAが、植物の減数分裂細胞と周辺の体細胞の協調的な発生制御を取り持つ伝達役として機能する可能性を示唆する成果です。
本研究成果は、米国オンラインジャーナル「PLOS Genetics」に平成30年2月12日に掲載されました。
本研究は、遺伝研と包括連携協定を結ぶ東京農業大学、および新潟大学との共同研究の成果です。また、文科省科研費・新学術領域「植物新種誕生原理」(17H05849)、科研費・基盤研究(A) (25252004)、東京農業大学生物資源ゲノム解析センター生物資源ゲノム解析拠点事業、遺伝研共同研究 (NIG-JOINT 2015-61、2016-65、87A2017)、および遺伝研博士研究員制度による支援を受けました。
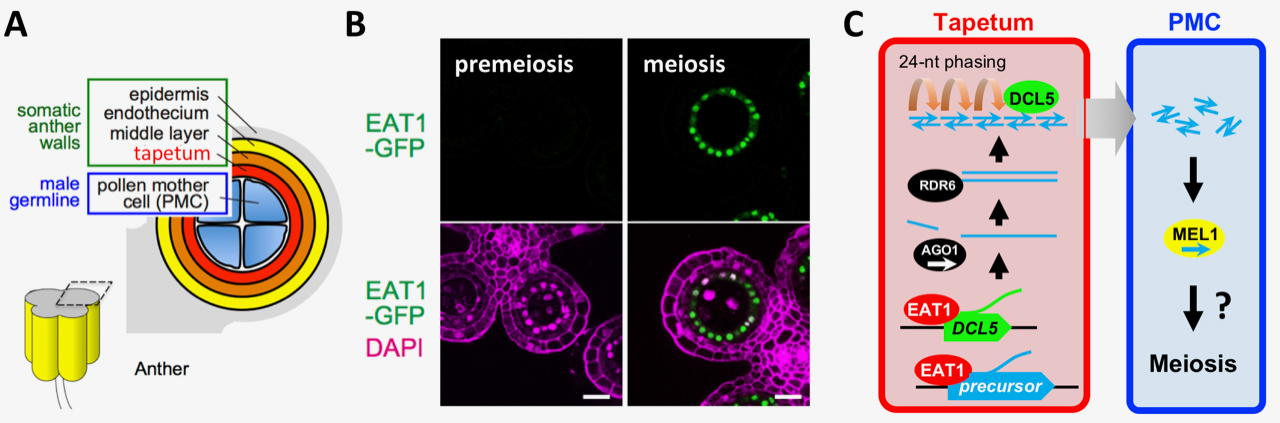
図1. EAT1転写因子は葯タペート組織で24塩基長の減数分裂small RNA (sRNA) の生合成を促進
(A) 葯 (anther) の断面図。タペート組織 (tapetum) は減数分裂細胞(PMC)と接する。(B) EAT1転写因子(緑)は減数分裂期 (meiosis) タペート細胞の核に蓄積するが、前減数分裂期 (premeiosis) では発現しない。スケールバー; 20µm。(C) EAT1はタペートでsRNA前駆体とDCL5遺伝子の転写を活性化する。前駆体RNAは切断・二本鎖化を経てDCL5により24塩基に切断される。PMCで発現するMEL1と24塩基長sRNAの結合から、sRNAの細胞間移動が示唆される。
「先進ゲノム支援」情報解析講習会のご案内
斎藤成也教授の論文引用が5万件を超えました
斎藤成也教授が1987年にMolecular Biology and Evolution に発表された下記論文の引用が5万件(Google Scholar Citations)を超えました。(2018年2月6日現在)
▶ 論文:The neighbor-joining method: a new method for reconstructing phylogenetic trees.
N Saitou, M Nei
Molecular biology and evolution 4 (4), 406-425, 1987
論文引用とは:優れた論文は、ほかの論文に引用される回数が多くなることから、論文が引用される回数は、その論文の質を表す指標のひとつと考えることができます。このため、被引用回数が多い論文は知的貢献度が高いと見なされています。
Google Scholar Citations では、著者ごとの論文とその引用回数を明示的に確認することができます。
細胞分裂期の染色体凝縮はマグネシウムイオンの増加によって起こる ―生細胞イメージングにより新たなメカニズムを検証―
![]()
A transient rise in free Mg2+ ions released from ATP-Mg hydrolysis contributes to mitotic chromosome condensation
Kazuhiro Maeshima, Tomoki Matsuda, Yutaka Shindo, Hiromi Imamura, Sachiko Tamura, Ryosuke Imai, Syoji Kawakami, Ryosuke Nagashima, Tomoyoshi Soga, Hiroyuki Noji, Kotaro Oka, Takeharu Nagai
Current Biology Published Online January 18, 2018 DOI:10.1016/j.cub.2017.12.035
細胞が分裂する際、ヒトでは全長2メートルにもおよぶゲノムDNAからコンパクトに凝縮した「染色体」と呼ばれるDNAの束が作られ、2つの細胞に正確に分配されていきます。半世紀以上前、細胞に大量に存在するマグネシウムイオン(Mg2+)がゲノムDNA凝縮の鍵となりうることが提唱されたことがありましたが、当時は細胞内Mg2+濃度を測定する手段が無かったため証明されぬまま忘れられていました。
国立遺伝学研究所の前島一博 教授、大阪大学の永井健治 教授、慶應義塾大学の岡浩太郎 教授、京都大学の今村博臣准教授らの共同研究グループは、蛍光タンパク質技術を駆使してMg2+濃度の変化を高感度で感知できる蛍光センサー MARIO を開発し、生細胞内のMg2+濃度を蛍光イメージングにより可視化することに成功しました。そして細胞分裂の際にMg2+濃度が一過的に上昇することを示すとともに、負の電気を帯びているDNA同士の反発を弱め、染色体の凝縮を促進していることを明らかにしました(図1)。本研究によって、実際にMg2+が細胞のなかで染色体の凝縮にかかわっていることが初めて証明されました。また、Mg2+は通常ATPと結合していますが、細胞分裂でのATPの消費により放出されることで、Mg2+濃度が上昇することも分かりました(図1)。
染色体の形成の失敗はゲノムDNAの損傷を引き起こし、細胞に「死」や「がん化」などのさまざまな異常、さらには疾病をもたらすと考えられています。また細胞のなかに多量に存在するMg2+は多くのタンパク質の働きを助けており、欠乏するとさまざまな細胞異常が現れることが知られています。今回の蛍光センサー開発と生物学的知見の発見は、このような細胞の異常が起こるしくみの解明につながると期待されます。
本研究成果は、2018年1月19日(金)午前2時(日本時間)に「Current Biology」に掲載されました。 Dispatchesセクションにおいてハイライトされています。
本研究は、国立遺伝学研究所・前島一博教授・田村佐知子テクニカルスタッフグループ、大阪大学産業科学研究所・ 永井健治教授・松田知己准教授グループ、慶應義塾大学理工学部・岡浩太郎教授・新藤豊特任助教グループ、京都大学生命科学研究科・今村博臣准教授・東京大学工学研究科・野地博行教授グループ、慶應義塾大学環境情報学部・曽我朋義教授グループの共同研究成果です。
なお、本研究は文部科学省科学研究費・新学術領域「少数性生物学」(領域代表:大阪大学 永井健治教授)、科学技術振興機構 戦略的創造研究推進事業(CREST) (JPMJCR15G2, JPMJCR15N3)、および科学研究費(16H04746)、日本医療研究開発機構の革新的先端研究開発支援事業 AMED-CRESTの支援を受けました。
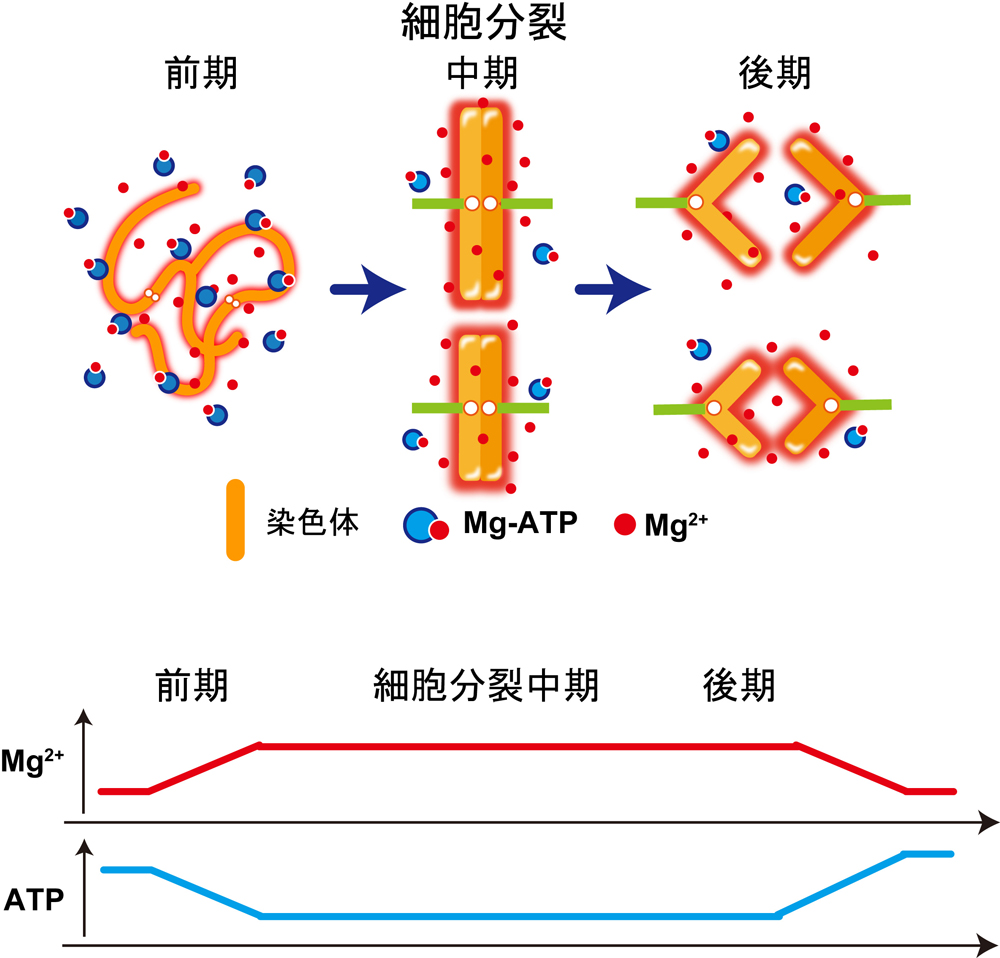
図1:細胞が分裂する際にMg2+が増加し、染色体の凝縮が促進される。ATPの減少によりMg-ATPから遊離したMg2+が供給される。
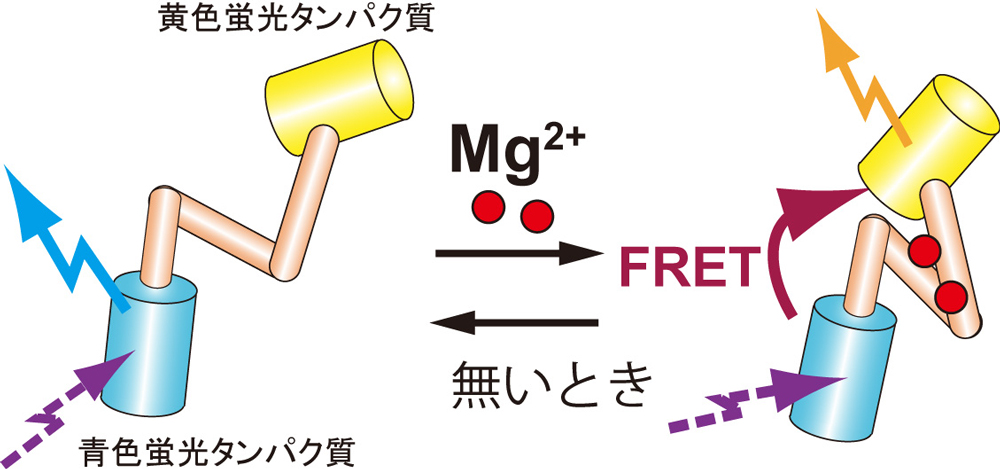
図2:新しい蛍光Mg2+センサーMARIOの検出原理 青色と黄色の2種類の蛍光タンパク質が、検出タンパク質(肌色)でつながれている。Mg2+が検出タンパク質に結合すると、2種類の蛍光タンパク質が引き寄せられ、FRETと呼ばれるエネルギーの移動が起こり、紫の光を照射したときに放射される蛍光の色が青から黄にシフトする。
APCは紡錘体を引っ張る力のブレーキ がん抑制遺伝子APCの新しい機能の発見
![]()
Tumor suppressor APC is an attenuator of spindle-pulling forces during C. elegans asymmetric cell division
Kenji Sugioka, Lars-Eric Fielmich, Kota Mizumoto, Bruce Bowerman, Sander van den Heuvel, Akatsuki Kimura, Hitoshi Sawa
PNAS January 18, 2018 DOI:10.1073/pnas.1712052115
大腸ガン患者では高頻度でがん抑制遺伝子APCに変異があることが知られています。これは、Wntシグナルという細胞内シグナル伝達経路が過剰に活性化することが主な原因です。これに加えて、APCタンパク質は微小管を制御する働きが知られていますが、その働きと細胞のがん化との関係はよくわかっていませんでした。
本研究では、細胞の表層に局在したAPCタンパク質が、染色体の分配を担う紡錘体を構成する微小管の末端に結合して、微小管の安定性を制御していることが明らかになりました。APCタンパク質の微小管への結合は、微小管を引っ張る力を弱めることによって、染色体の移動を制御していたのです。
APC遺伝子が変異した大腸がん細胞では染色体異常が頻繁に観察されます。また、染色体異常は染色体の分配をおこなう紡錘体にかかる力の異常によっても引き起こされることが指摘されています。本研究で明らかになったAPCタンパク質の紡錘体を引っ張る力を制御する働きががん化に関与している可能性があります。
本研究は、情報・システム機構国立遺伝学研究所の元研究員杉岡賢史、澤斉教授と同研究所細胞建築研究室およびユトレヒト大学、オレゴン大学との国際共同研究の成果です。
本研究成果は、米国科学誌「米国科学アカデミー紀要(PNAS)」に平成30年1月18日(米国東部標準時間)に掲載されました。
本研究は、科学研究費補助金(JP22127005、JP15H04732 、JP15KT0083)上原記念生命科学財団および遺伝学研究所共同研究(NIG-JOINT:2013-A60)の支援を受けておこなわれました。
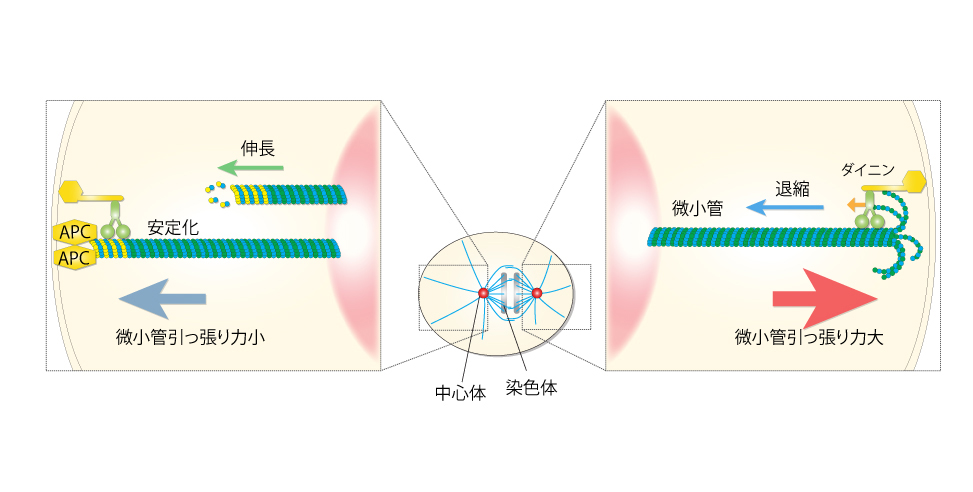
図:APCタンパク質による微小管引張り力の抑制機構
微小管が細胞表層に到達した後、退縮する際に、モータータンパク質であるダイニンの働きで引っ張る力が発生する。APCタンパク質が微小管の末端に結合することで退縮を阻害し、引っ張る力を弱める。
線虫の非対称分裂の動画をこちらでご覧いただけます
右:野生型では後極から強く引っ張られるため紡錘体が後方に移動する。この際、後ろ側の中心体は、強い力を受けて縦方向に振動する。前極に局在するAPCが引っ張る力を抑制するため前側の中心体はほとんど動かない。左:APCを阻害すると、前方の中心体も振動する。
姉妹染色分体間接着の形成機構を解明〜コヒーシンはDNAの結束バンド !?〜
![]()
Establishment of DNA-DNA Interactions by the Cohesin Ring
Yasuto Murayama, Catarina P. Samora, Yumiko Kurokawa, Hiroshi Iwasaki and Frank Uhlmann
CellPublished Online January 18, 2018 DOI:10.1016/j.cell.2017.12.021
生命の設計図であるDNAは非常に長い“糸”で、細胞核の中で様々なタンパク質と結合し、染色体を形成しています。染色体は細胞が分裂する毎にコピーされ、分配されます。染色体はコピーされた直後、物理的に接着しています(姉妹染色分体間接着)。この染色体の物理的接着がなくなると染色体が正確に分配されなくなることがわかっています。この染色体の物理的接着には「コヒーシン」と呼ばれるリング状のタンパク質の働きが重要です(図)。コヒーシンはDNAと直接結合することがわかっていますが、姉妹染色分体を接着する仕組みはわかっていませんでした。
情報・システム研究機構 国立遺伝学研究所の村山泰斗准教授と東京工業大学の岩崎博史教授らの研究グループは、コヒーシンを細胞から分離精製し、コヒーシンとDNAの複合体の形成を試験管内で再現することにより、コヒーシンによる姉妹染色分体接着の仕組みの一端を明らかにしました。コヒーシンは「結束バンド」のように2本のDNAを束ねることがわかったのです(図)。
本成果によりコヒーシンの性質を詳細に明らかにしたことが、コヒーシンの機能欠損が原因とされている様々な疾患や不妊の原因解明に繋がると期待されます。
本研究成果は、平成30年1月18日 (米国東部時間) に米国科学雑誌 Cell に掲載されました。
本研究は、情報・システム研究機構・村山泰斗准教授、黒川裕美子研究員、東京工業大学・岩崎博史教授、英国フランシスクリック研究所・Frank Uhlmann グループとの共同研究としておこなわれました。
本研究は、科学研究補助金 (16H06160、16H01404、15H059749)、日本分子生物学会若手研究助成富沢純一・圭子基金の支援を受けておこなわれました。
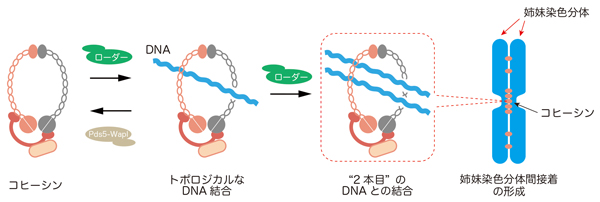
図:コヒーシンによる姉妹染色分体間接着の形成モデル。
コヒーシンは、自身のリングの一部を開いて、その内側に通すようにしてDNAと結合し、“2本目”のDNAと結合する。これにより、DNA−コヒーシン−DNAの構造をつくって、2つの姉妹染色分体の間に接着を形成すると考えられる。















