Archive
- Home
- 2026/2
- 2026/1
- 2025/12
- 2025/11
- 2025/10
- 2025/9
- 2025/8
- 2025/7
- 2025/6
- 2025/5
- 2025/4
- 2025/3
- 2025/2
- 2025/1
- 2024/12
- 2024/11
- 2024/10
- 2024/9
- 2024/8
- 2024/7
- 2024/6
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
エピゲノム研究室/ゲノムダイナミクス研究室 齋藤さんが「研究科長賞」を受賞

総合研究大学院大学の各研究科において、特段に顕彰するに相応しい研究活動を行い、修了する学生に対し、研究科長賞が授与されます。
エピゲノム研究室 角谷研究室(2020年9月まで) / ゲノムダイナミクス研究室 前島研究室(2020年10月より)に所属する齋藤 絡さんが、2020年度後期の生命科学研究科 研究科長賞を受賞しました。
・齋藤 絡(エピゲノム研究室 角谷研究室 / ゲノムダイナミクス研究室 前島研究室)
「Evolution, biochemical properties and single-molecule dynamics of transposon-encoded anti-silencing factor」
齋藤さんは今回、遺伝学専攻の「森島奨励賞」も受賞しています。
「独習 Pythonバイオ情報解析」を先進ゲノム支援情報解析班が羊土社より出版
 ゲノムサイエンスの分野で圧倒的な支持が得られている「Python」。Pythonでバイオインフォに取り組み、いずれは機械学習など始めたい方に最適の教科書です。本書では、汎用的なテーブルデータ解析、可視化ライブラリを用いて、生命科学特有のシングルセル、RNA-Seq解析を実装しつつ学ぶことができます。
ゲノムサイエンスの分野で圧倒的な支持が得られている「Python」。Pythonでバイオインフォに取り組み、いずれは機械学習など始めたい方に最適の教科書です。本書では、汎用的なテーブルデータ解析、可視化ライブラリを用いて、生命科学特有のシングルセル、RNA-Seq解析を実装しつつ学ぶことができます。
| 実験医学別冊 「独習 Pythonバイオ情報解析」 Jupyter、NumPy、pandas、Matplotlibを理解し、実装して学ぶシングルセル、RNA-Seqデータ解析 先進ゲノム解析研究推進プラットフォーム/編 |
|
| 羊土社 書籍のページ 文部科学省科学研究費助成事業 先進ゲノム解析研究推進プラットフォーム 国立遺伝学研究所 先端ゲノミクス推進センター |
ミドリイガイのゲノム解析からわかった足糸の耐久性の秘密
Press release

Genomics and transcriptomics of the green mussel explain the durability of its byssus
Koji Inoue*, Yuki Yoshioka, Hiroyuki Tanaka, Azusa Kinjo, Mieko Sassa, Ikuo Ueda, Chuya Shinzato, Atsushi Toyoda, Takehiko Itoh Scientific Reports 11, 5992 (2021) DOI:10.1038/s41598-021-84948-6東京大学大気海洋研究所の井上広滋教授らは、ミドリイガイ(写真)の全ゲノム配列の解読を行い、高精度なゲノム情報の再構築に成功しました。ミドリイガイはムール貝の仲間で、熱帯・亜熱帯沿岸生態系の主要種であり、また、東南アジアで広く養殖される水産重要種です。さらに、本種には海水中のマイクロプラスチック粒子や汚染物質を蓄積する性質があり、海洋汚染の指標生物としても注目されています。今回解明した全ゲノム配列は極めて精度が高く、生理学、生態学、水産食品学など様々な研究分野に今後大きく貢献することが期待されます。
さらに、井上教授らは、解明したゲノム配列から、ミドリイガイが海中基盤に付着するために合成する「足糸」の耐久性のしくみを解明しました。ムール貝類の足糸は、微生物や酵素による分解に耐性があることが知られていましたが、そのしくみはこれまでわかっていませんでした。今回解明したゲノム上の遺伝子の中から、足糸を合成する「足」で主に発現し、かつ発現量が高い81遺伝子について詳しく調べると、タンパク質分解酵素阻害物質や生体防御関連の遺伝子がその約30%を占めていました。すなわち、足糸の耐久性は、頑丈な構造を作るだけでなく、分解から防御するための遺伝子を多数動員して実現していることが初めて明らかになったのです。この成果は、海中施設や船舶への貝類の付着防除対策に新たな手掛かりをもたらすものです。
本研究は、科学研究費助成事業新学術領域研究「学術研究支援基盤形成」先進ゲノム解析研究推進プラットフォーム(16H06279 (PAGS))、基盤研究(B)(18H02261)、東京大学—日本財団「FSI海洋ごみ対策プロジェクト」の支援を受けて実施したものです。
本研究成果は、英国科学雑誌「Scientific Reports」に2021年3月16日午後7時(日本時間)に掲載されました。
遺伝研の貢献 illumina社のHiSeq2500システムを用いて、熱帯・亜熱帯性のムール貝の一種ミドリイガイのゲノム情報を整備しました。本解析は、2018年度先進ゲノム支援の支援課題としておこなわれたものです。
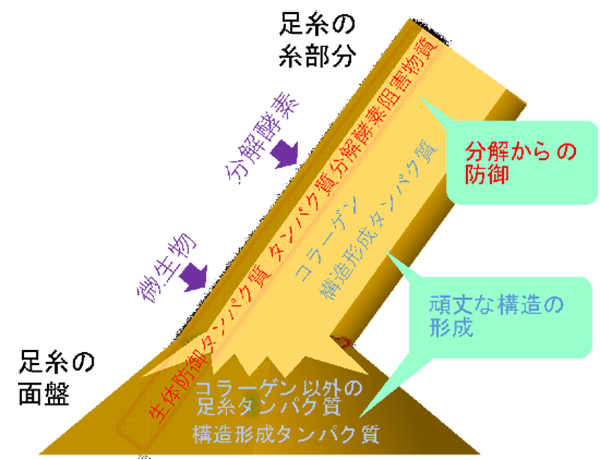
図: イガイ類が足糸の耐久性を高めるしくみ
アサガオの花びら(花冠)がまっすぐに伸びる力学的な仕組みを解明
Press release

Reduction in organ-organ friction is critical for corolla elongation in morning glory.
Ayaka Shimoki, Satoru Tsugawa, Keiichiro Ohashi, Masahito Toda, Akiteru Maeno, Tomoaki Sakamoto, Seisuke Kimura, Takashi Nobusawa, Mika Nagao, Eiji Nitasaka, Taku Demura, Kiyotaka Okada, Seiji Takeda.
Communications Biology 4, 285 (2021) DOI:10.1038/s42003-021-01814-x
京都府立大学の武田征士准教授(細胞工学研究室)、奈良先端科学技術大学院大学の津川暁特任助教(植物代謝制御研究室)らの共同研究グループは、江戸時代から知られている変化アサガオのひとつで、花びらが折れ曲がる「台咲(だいざき)」系統を材料に、花びらがまっすぐに伸びる力学的な仕組みを明らかにしました。花器官表面にあるミクロ構造「分泌腺毛」が、器官どうしの摩擦を軽減することで、狭いつぼみの中でも花びらが伸長できることが分かりました。この仕組みを応用し、観賞用の花の形を自在に制御する園芸技術につながる可能性があります。
本研究は、文部科学省および日本学術振興会の科学研究費補助金(JP18K06366, JP18H05484, JP18H0548, JP18H04787)文部科学省の私立大学戦略的研究基盤形成支援事業(S1511023)、国立遺伝学研究所NIGJOINT(44A2020)の支援を受けて行われました。
本研究成果は、国際学術誌「Communications Biology」に、令和3年3月5日10時 (GMT)に掲載されました。
遺伝研の貢献
マイクロフォーカスX線CT装置を用いてアサガオ蕾のCTスキャンを行い、蕾内部の花弁等の主要器官や維管束構造の観察に貢献しました。本解析は2020年度国立遺伝学研究所共同研究(NIG-Joint)として行われたものです。
▶︎ 国立遺伝学研究所 3D Imaging Roomウェブサイト
▶︎ 生物の複雑な構造を3次元で解き明かす
本研究はNBRPのアサガオのリソースを活用して遂行されました。

図: 野生型(左)と台咲(右)の花
野生型では花弁がまっすぐ伸長して漏斗状になる。台咲では花弁が2度折れ曲がり、花の中央に筒状の「台」と呼ばれる構造を作る。
動画: 本成果のmicroCTによるアサガオ蕾の解析部分を抜粋し要約。
①スキャンしたサンプルと装置
②3次元表示動画による外観観察
③注目部位を拡大スキャンしたデータの2次元および3次元表示動画による詳細な観察
- 本研究を紹介したコラムはこちら
エピゲノム研究室/ゲノムダイナミクス研究室 齋藤さんが「森島奨励賞」を受賞

総合研究大学院大学 遺伝学専攻が独自に行っている「森島奨励賞」の選考が、2020年度後期の学位出願者に対して行われ、エピゲノム研究室 角谷研究室(2020年9月まで) / ゲノムダイナミクス研究室 前島研究室(2020年10月より)に所属する齋藤 絡さんが受賞しました。
・齋藤 絡(エピゲノム研究室 角谷研究室 / ゲノムダイナミクス研究室 前島研究室)
「Evolution, biochemical properties and single-molecule dynamics of transposon-encoded anti-silencing factor」
授与式が2021年3月9日に行われ、花岡専攻長から賞状と研究奨励金が贈られました。
森島奨励賞とは
総研大遺伝学専攻で優秀な研究成果を発表して学位を取得した学生に、その研究内容を称えるとともに今後のさらなる発展を促す目的で贈られます。遺伝学の先達
遺伝子が密に並んだゲノム上で転写を調節する仕組み
Chromatin-based mechanisms to coordinate convergent overlapping transcription.
Soichi Inagaki, Mayumi Takahashi, Kazuya Takashima, Satoyo Oya, Tetsuji Kakutani.
Nature Plants 2021 March 1 DOI:10.1038/s41477-021-00868-3
タンパク質をコードする遺伝子のみならず、タンパク質をコードしない非コード領域も含め、予想より多くのゲノム領域で転写が起きていることが、さまざまな生物でわかってきましたが、ゲノム上で近接していたり、オーバーラップしていたりする転写を調節する仕組みは大部分が未解明でした。
本研究では、ゲノムがコンパクトで、遺伝子が密に並んでいるシロイヌナズナという植物を用いた解析により、数百もの遺伝子の内部において逆向きにオーバーラップする転写が起きていること、また、これがクロマチン修飾によって調整されていることを見出しました。両方向性の転写が起きている領域では、ヒストン修飾の一つ、ヒストンH3の4番目のリジンのモノメチル化(H3K4me1)が除去されることで、転写伸長の抑制が起きていました。またこの制御にはDNAのねじれを防ぐDNAトポイソメラーゼが関与していることもわかり、遺伝子が密に転写することで起きうるDNA のねじれを防ぐのに重要な役割を果たしていることが予想されます。面白いことに、この両方向性の転写を介した遺伝子制御は、植物が春に花を咲かせるのを可能にする、冬を経験したことの「エピジェネティックな記憶」にも関与していました。本研究の成果は、ゲノム上での近隣遺伝子との相互作用が遺伝子の制御に与える影響を示唆しており、今後、この未開拓な経路の分子機構や生物学的インパクトの理解につながることが期待されます。
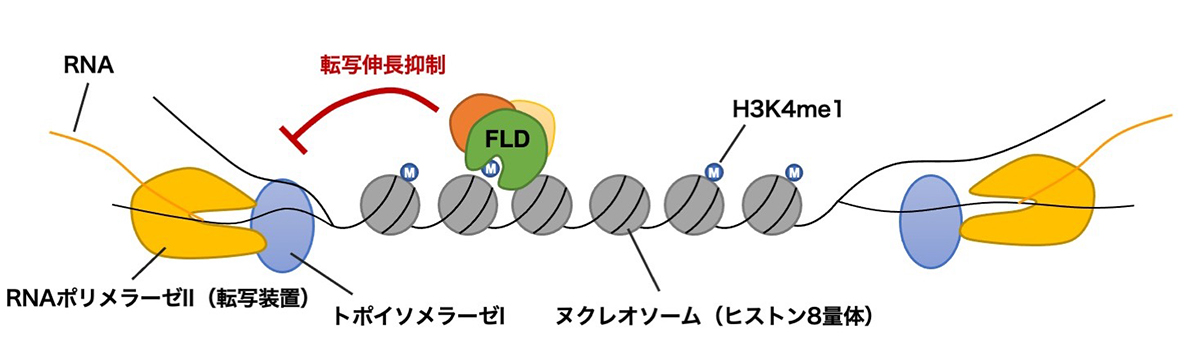
図:FLDによる制御モデル。
RNAポリメラーゼIIはDNA二本鎖をほどきながら遺伝子の転写を進める。トポイソメラーゼIは転写装置と協調して働き、DNAのねじれをほどき、転写の進行(転写伸長)を助ける。遺伝子の転写領域にはH3K4me1が局在しているが、両方向から転写するような遺伝子の場合、FLDがH3K4me1を除くことで転写の伸長を抑制し、両方向からの転写を円滑にしていると推察される。
派手な雄は何のため?
〜熱帯メダカのゲノム解析が明らかにする性差の多様性の遺伝基盤〜
Press release

(月刊アクアライフ・橋本直之撮影)
Genome editing reveals fitness effects of a gene for sexual dichromatism in Sulawesian fishes.
S Ansai, K Mochida, S Fujimoto, D F Mokodongan, B K A Sumarto, K W A Masengi, R K Hadiaty, A J Nagano, A Toyoda, K Naruse, K Yamahira, J Kitano
Nature Communications 12, 1350 (2021) DOI:10.1038/s41467-021-21697-0
クジャクの羽のようなオス特有の派手な装飾はどうやって進化してきたのでしょうか?オス特有の派手な装飾は、異性に「モテる」ため、同性を打ち負かすために進化したと考えられていますが、どのような遺伝子によって派手な装飾が生まれたのかはよくわかっていませんでした。
インドネシアのスラウェシに生息するメダカの一種、ウォウォールメダカのオスは「赤いヒレ」が特徴です。本研究では、ウォウォールメダカを用いてオスのヒレを赤くする遺伝子を特定し、赤いヒレを持つことの意義を明らかにすることに挑戦しました。
まず、近縁でオスのヒレが赤くないセレべスメダカの全ゲノム配列を決定し、ヒレが赤くなるウォウォールメダカとの違いを解析することで、csf1という遺伝子がヒレを赤くする候補遺伝子であることを特定しました。ゲノム編集でウォウォールメダカのcsf1を破壊するとオスのヒレの赤色がなくなりました。csf1がヒレを赤くする原因遺伝子だということがわかったのです。csf1は男性ホルモンを投与することで発現量が上昇することもわかったので、オスにのみ赤色が強く発色することを説明できました。
さらに、ゲノム編集によってヒレの赤色を失ったオスを利用して行動実験を行ったところ、メスは「ヒレが赤くないオス」にあまり惹きつけられませんでした。さらに、捕食者は「ヒレが赤くないオス」を捕まえようとしました。
他の生物種でも類似の研究を実施することで、「派手なオス」の出現という進化の謎に迫ることが期待されます。
本研究は、国立遺伝学研究所、琉球大学、東北大学、基礎生物学研究所、龍谷大学、インドネシア科学院、サム・ラトゥランギ大学の共同研究として実施されました。
また、本研究は、科研費(18K14769、16K14792、26291093、19K16232、16H06279)、先進ゲノム支援(16H06279)、学術振興会PD(16J0553)、基礎生物学研究所 共同利用研究 (17-313)、琉球大学熱帯生物圏研究センター 共同利用研究、国立遺伝学研究所 共同利用研究NIG-JOINT(20A2018, 20A2019, and 5B2020)の支援を受けました。
本研究成果は、英国科学雑誌「Nature Communications」に2021年3月1日午後7時(日本時間)に掲載されました。
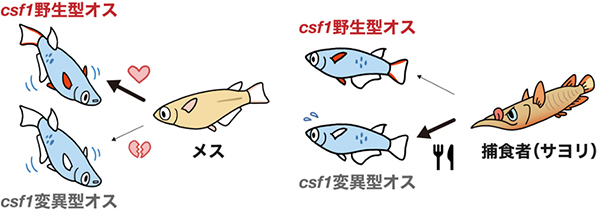
図: 行動実験の結果の概要
- 本研究を紹介したブログ記事はこちら
現代ヤポネシア人(日本列島人)の内なる二重構造
Press release
Genome-wide SNP data of Izumo and Makurazaki populations support inner-dual structure model for origin of Yamato people
Timothy Jinam, Yosuke Kawai, Yoichiro Kamatani, Shunro Sonoda, Kanro Makisumi, Hideya Sameshima, Katsushi Tokunaga, and Naruya Saitou
Journal of Human Genetics 2021 January 25. DOI:10.1038/s10038-020-00898-3
日本列島人(ヤポネシア人)の形成に関する「二重構造」モデルは、在来の採集狩猟民(象徴的に「縄文系」と呼ぶ)と弥生時代以降に日本列島に渡来した稲作農耕民(象徴的に「弥生系」と呼ぶ)が混血したと仮定しています。その結果、縄文系の要素は北方に居住するアイヌ人と南方に居住するオキナワ人の双方で高い一方、中央部に居住するヤマト人は弥生系の要素が高くなっています。このモデルは私たちが以前おこなった遺伝子解析で支持されましたが、その解析で使われたヤマト人は主に東京周辺に居住する人々でした。今回私たちは島根県の出雲出身者45名と鹿児島県の枕崎出身者72名のゲノム規模SNPデータを生成し、それらとバイオバンクジャパンのデータを含む東アジアの他の人類集団のゲノムデータと比較解析しました。主成分分析、系統ネットワークなどのさまざまな統計手法を用いた結果、出雲・枕崎・東北地方の集団は、関東(東京を含む)・東海・近畿と遺伝的にすこし異なっていました。日本列島中央部内に居住するヤマト人内にこのような内部構造が生じたのは、縄文時代以降に東アジア大陸部から複数回の渡来があったことを示唆します。これは、「二重構造」モデルをすこし変更した「うちなる二重構造」モデルを支持しています。
本研究は、集団遺伝研究室のTimothy Jinam助教と斎藤成也教授、国立国際医療研究センターの河合洋介副プロジェクト長と徳永勝士プロジェクト長、東京大学大学院新領域創成科学研究科の鎌谷洋一郎教授、鹿児島県枕崎市医師会の園田俊郎博士、牧角寛郎博士、鮫島秀弥博士らによって実施されました。また、出雲地方出身者のDNA収集には、東京いずもふるさと会の岡垣克則会長、荒神谷博物館の藤岡大拙館長にお世話になりました。現代人の進化に関する総合研究大学院大学の共同研究経費、文部科学省の新学術領域研究「ヤポネシアゲノム」(18H05505)、大学共同利用機関4機関連携の文理融合研究、およびジェネシスヘルスケア社との共同研究の支援を受けました。
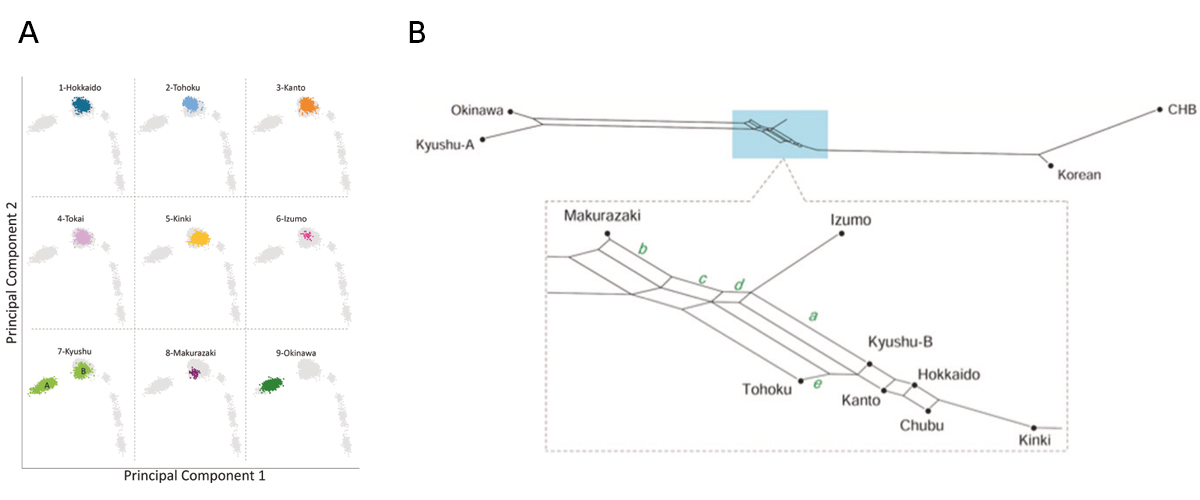
図: ヤポネシアの9集団といくつかの大陸集団の遺伝的関係
(A) 主成分分析の結果。出雲 (6)と枕崎 (8)のDNAデータは今回あらたに決定し、他の7集団のデータはバイオバンクジャパンから提供を受けました。大陸の5集団は右下に位置しています。九州 (7)は 沖縄 (9)と近いクラスターAとヤマト人(他の7集団)と近いクラスターBに分かれました。 (B) Neighbor-Net法を用いた系統ネットワーク。沖縄と九州-Aが左に、韓国人と北京の漢族 (CHB)が右に位置しています。枕崎集団は左側のクラスターにもっとも近く、次に出雲集団が近くなっています。逆に近畿集団は右側の大陸クラスターにもっとも近く位置します。
魚類と羊膜類における視覚系から終脳への中継核の収斂進化
Non-thalamic origin of zebrafish sensory nuclei implies convergent evolution of visual pathways in amniotes and teleosts
Solal Bloch, Hanako Hagio, Manon Thomas, Aure´ lie Heuze´, Jean-Michel Hermel, Elodie Lasserre, Ingrid Colin, Kimiko Saka, Pierre Affaticati, Arnim Jenett, Koichi Kawakami, Naoyuki Yamamoto, Kei Yamamoto Elife 9, e54945 (2020) DOI:10.7554/eLife.54945哺乳類の視覚情報を皮質に伝える視蓋徘徊性経路(視蓋から視床を経由して皮質へ投射する経路)に類似した上行性の投射は、脊椎動物種にひろく見られますが、それらの経路の相同性、発生学的起源についてはよくわかっていませんでした。その進化について、より深い洞察を得るために、我々はゼブラフィッシュの視床類似構造、糸球体前核群(PG)の発生学的起源を解析しました。羊膜類の視床核と同様に、PGの外側核は視蓋の情報を受け取り終脳に投射します。トランスジェニックゼブラフィッシュを用いた細胞系譜解析により、PGの細胞の大部分が羊膜類の視床とは異なり中脳に由来することが明らかになりました。このことは、真骨魚類のPGは、前脳由来の細胞で形成される羊膜類の視床と相同ではないことを示唆しています。魚類の機能的に視床皮質経路に類似した投射は、非前脳細胞集団から形成されていたということは、脊椎動物の感覚系の収斂進化の驚くべき多様性を示しています。
1. 背景 脊椎動物の脳は発生学的に3つの主要な区分から成っています。すなわち、前脳、中脳、および後脳です。脳機能を決定するニューロンの接続は、後の発生段階で確立されます。機能的な接続は、異なる脊椎動物グループ間で保存されています。接続パターンは脊椎動物間で類似していることが多いため、それらの発生学的起源が同じか否かはよくわかっていませんでした。
2. 結果 ゼブラフィッシュの視覚情報を視蓋から終脳(外套、哺乳動物の皮質に相当する)へ投射する中継核―糸球体前核群(PG)―を特異的にラベルする遺伝子トラップ系統を作製し解析しました。Cre-loxPシステムを用いた細胞系譜の解析等を行い、ゼブラフィッシュのPGは中脳由来であることをつきとめました。これは、羊膜類(哺乳類と鳥を含むグループ)脳の感覚情報を外套(皮質)に投射する視床核―皮質経路が前脳内の経路であるのとは異なります。このように、脊椎動物脳のニューロンの接合パターンは、類似した機能を維持しているが発生学的起源を異にしており、収斂進化により形成されたと考えられます。
3. 今後の期待 魚類脳と羊膜類や哺乳動物脳の構造と機能を解析することにより、私たちはどのようにして複雑な脳を獲得してきたのか?について理解と洞察を得ることができます。
本研究は、パリ=サクレ大学・フランス国立科学研究センター(CNRS) 山本渓博士、名古屋大学大学院生命農学研究科山本直之博士との共同研究として行われました。本研究は部分的に、NBRP、NBRP基盤技術整備プログラムおよびNIG-JOINT (2013-A15)に支援されました。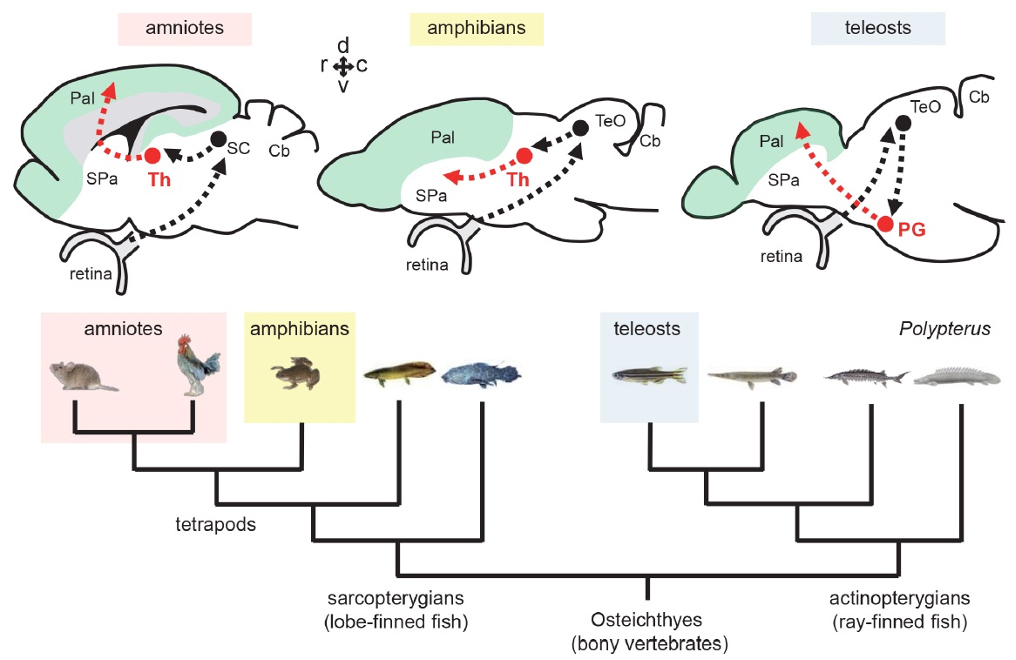
図1:さまざまな脊椎動物種における視蓋徘徊経路
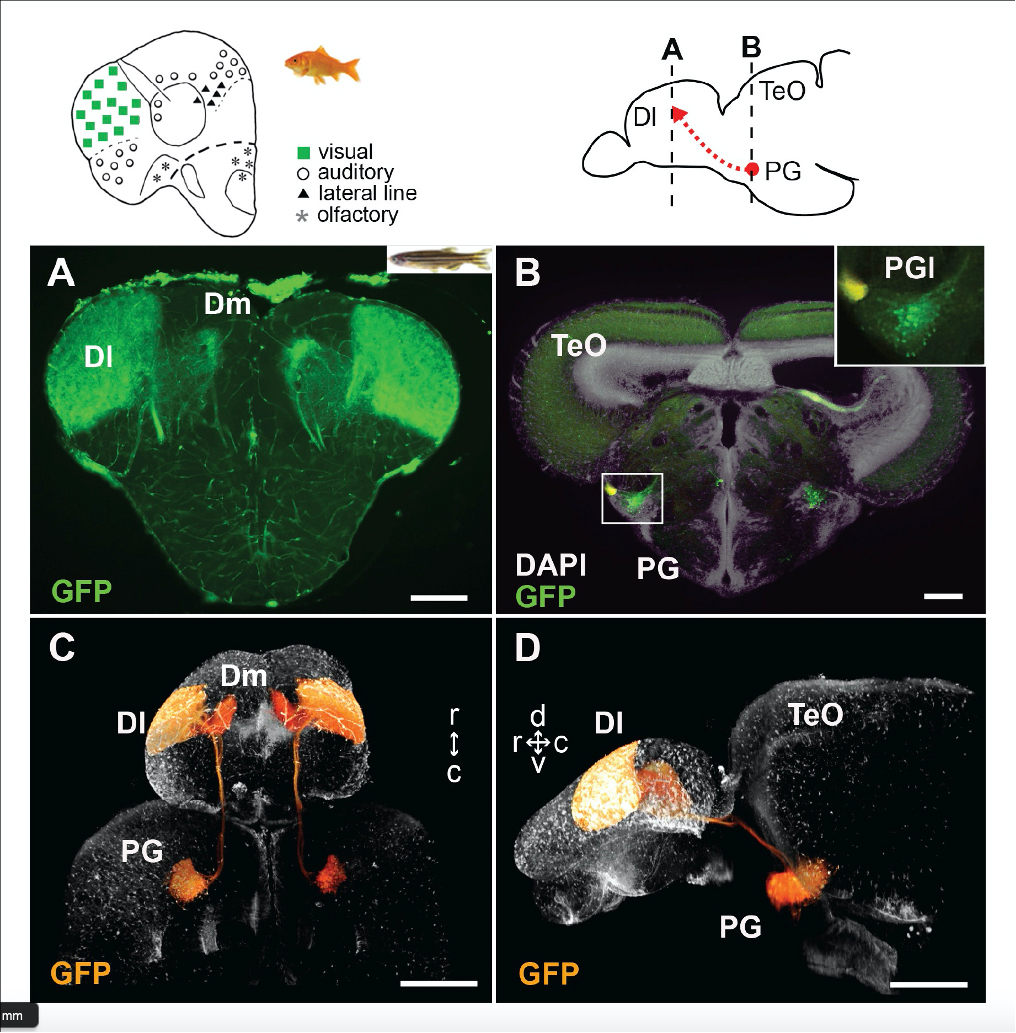
図2:遺伝子トラップゼブラフィッシュ系統においてGFP発現でラベルされた糸球体前核群(PG)細胞および終脳への投射。緑色(A, B)、オレンジ色(C,D)。
ゲノム概念誕生百周年記念国際シンポジウム
2021年度入試日程
ヘキサンジオールは細胞内のクロマチンの動きを止める
1,6-hexanediol rapidly immobilizes and condenses chromatin in living human cells
Yuji Itoh, Shiori Iida, Sachiko Tamura, Ryosuke Nagashima, Kentaro Shiraki, Tatsuhiko Goto, Kayo Hibino, Satoru Ide and Kazuhiro Maeshima
Life Science Alliance 4, e202001005 (2021) DOI:10.26508/lsa.202001005
液―液相分離によって細胞内で形成される液滴は、膜の無い構造体であり、ある分子の濃度を高め、細胞の機能を時間的・空間的に制御するために重要です。脂肪族アルコールの一つである、1,6-ヘキサンジオール (1,6-HD) は、液滴の形成に必要な、弱い疎水性のタンパク質―タンパク質相互作用、及びタンパク質―RNA相互作用を阻害するため、液滴を溶かす作用があります。このため、細胞質や核内の構造体形成の過程を調べるために盛んに使われてきました。しかし、生きた細胞の中で、1,6-HD がクロマチンにどのような影響を与えるかは、不明でした。
情報・システム研究機構 国立遺伝学研究所・伊藤優志 日本学術振興会特別研究員(元遺伝研博士研究員)、飯田史織 総研大生、田村佐知子 テクニカルスタッフ、井手聖 助教、前島一博 教授らのグループは、生細胞内の単一ヌクレオソームイメージングを用い、1,6-HDがヒト細胞内のクロマチンの動きを著しく抑制し、クロマチンを凝縮させることを発見しました。その効果は、1,6-HDの濃度が高いほど強く、5%以上の濃度では不可逆的になりました。このクロマチンの凝縮は、1,6-HDが液滴を可溶化する作用とは異なるメカニズムによって引き起こされていました。1,6-HDのようなアルコールは、クロマチンの周りの水分子を取り除き、局所的にクロマチンを凝縮させると考えられます。これらの結果は、クロマチンが関わる液滴に対して用いた場合、得られた結果を注意深く解釈・考察する必要があることを示しています。
本研究は、国立遺伝学研究所・ゲノムダイナミクス研究室の伊藤優志 日本学術振興会特別研究員、飯田史織 総研大生、田村佐知子 テクニカルスタッフ、井手聖 助教、日比野佳代 助教、永島崚甫 元総研大生、前島一博 教授、筑波大学 白木賢太郎 教授、帯広畜産大学生 後藤達彦 助教との共同研究成果です。日本学術振興会 (JSPS) 及び文部科学省科研費 (19K23735, 20J00572, 18K06187, 19H05273, 20H05936)、科学技術振興機構 (JST) 戦略的創造研究推進事業 (CREST) (JPMJCR15G2)、武田科学振興財団、上原記念生命科学財団、国立遺伝学研究所博士研究員、JSPS特別研究員(PD)の支援を受けました。
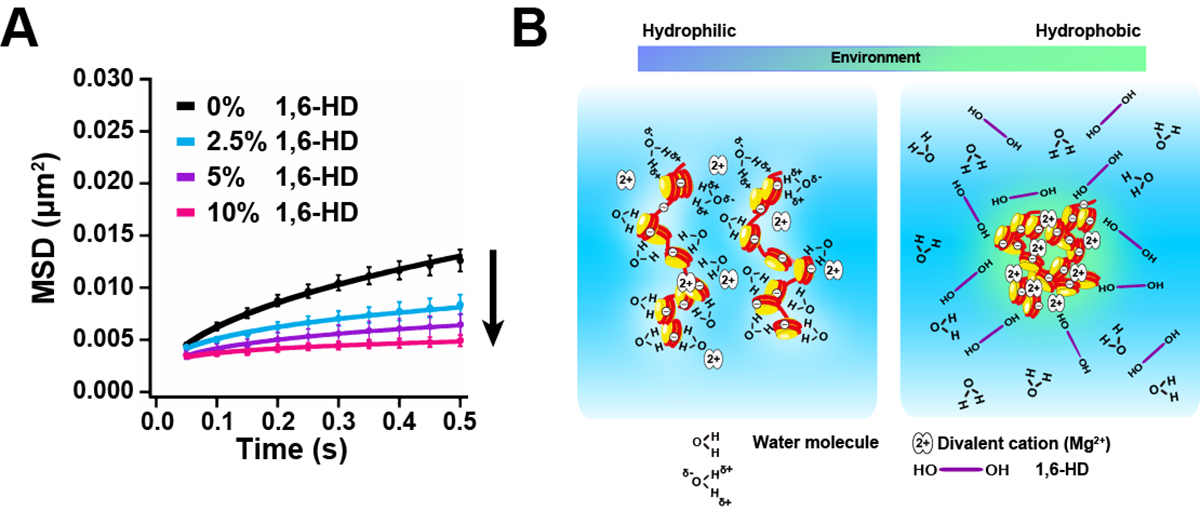
図:1,6-HDによるクロマチンの動きの抑制効果とクロマチン凝縮作用の模式図
(A) 生細胞のヌクレオソームの動きは1,6-HDによって濃度依存的に抑制される。単一ヌクレオソーム測定データを平均二乗変位MSDとして表した。(B)(左) クロマチンは静電相互作用で多くの水分子と会合している。(右) 1,6-HDのようなアルコールは、クロマチンの周囲の水分子を取り除くと思われ、クロマチンの周りの環境はより疎水的になる。この環境の変化が、クロマチンの凝縮を促進する。この模式図は単純化されたものであり、分子の大きさの比率は正確でない。また、1,6-HDが分子レベルでどのようにクロマチンに作用するか、分かっていない。
日本人に感染しているEBウイルスが他のアジア地域のEBウイルスと異なることを発見
Press release
A global phylogenetic analysis of Japanese tonsil-derived Epstein–Barr virus strains using viral whole-genome cloning and long-read sequencing
Misako Yajima, Risako Kakuta, Yutaro Saito, Shiori Kitaya, Atsushi Toyoda, Kazufumi Ikuta, Jun Yasuda, Nobuo Ohta and Teru Kanda J Gen Virol. 2021 January 12 DOI:10.1099/jgv.0.001549東北医科薬科大学 医学部の神田 輝(かんだ てる)教授(微生物学教室)と太田 伸男(おおた のぶお)教授(耳鼻咽喉科学教室)らの研究グループは、国立遺伝学研究所、東北大学 東北メディカル・メガバンク機構との共同研究により、日本人の扁桃組織に潜伏感染しているEBウイルスが、他のアジア地域でみられるEBウイルス株とは異なる系統であることを明らかにしました。EBウイルスが関連する疾患にはアジアの特定の地域に好発するものがあり、本研究は、アジアにおけるEBウイルス株の地域分布とEBウイルス関連疾患の地域偏在が一致する可能性があることを示した初めての報告です。
本研究結果は2021年1月12日付で国際専門誌Journal of General Virology誌のオンライン版に掲載されました(doi: 10.1099/jgv.0.001549)。
なお、本研究は日本学術振興会科学研究費助成事業 基盤研究(C)、文部科学省科学研究費助成事業 新学術領域研究 先進ゲノム支援(PAGS)、日本医療研究開発機構、三菱財団研究助成の支援を受けて行われたものです。
遺伝研の貢献 Pacific Biosciences社のSequelシステムを用いて、日本人に感染しているEBウイルスのゲノム情報を整備しました。本解析は2018年度先進ゲノム支援の支援課題としておこなわれたものです。
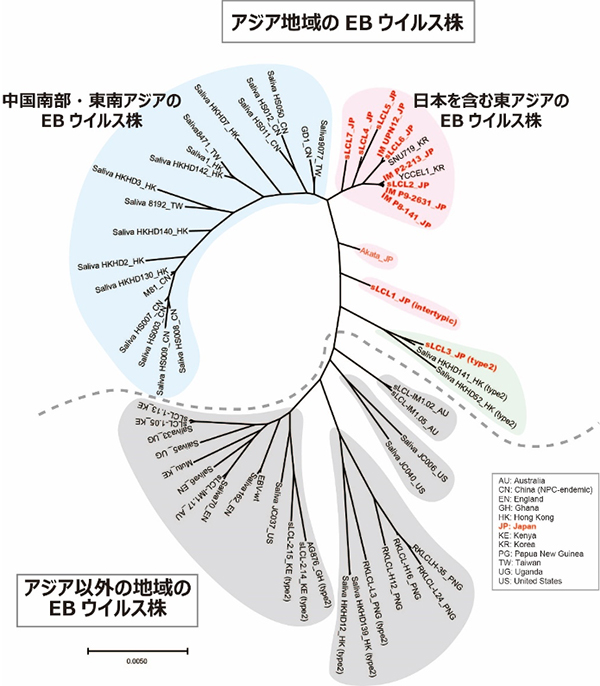
図: 世界のEBウイルス株の系統樹 EBウイルス関連疾患ではない検体(扁桃、唾液、血液など)から得られたEBウイルス株のウイルスゲノム全長の塩基配列情報を用いて系統樹を作成した。アジア地域のEBウイルス株の中で、日本を含む東アジアの株と中国南部・東南アジアの株は異なるグループを形成していることが明らかになった。
トマト祖先種のゲノム情報を高精度に解読
〜品種改良に役立つ遺伝子同定の手がかりに〜
Press release
De novo genome assembly of two tomato ancestors, Solanum pimpinellifolium and S. lycopersicum var. cerasiforme, by long-read sequencing.
Takei H, Shirasawa K, Kuwabara K, Toyoda A, Matsuzawa Y, Iioka S, Ariizumi T DNA Research 2020 January 20 DOI:10.1093/dnares/dsaa029トマトは世界で最も生産される野菜の一つであり、農業的かつ経済的に非常に重要な作物です。近年、特に健康志向の高まりや地球規模での気象変動により、より安定的に生産できる品種や、機能性成分がより含まれたトマト品種の開発が世界的に重要な課題となっています。形質を改良する育種の過程においては、様々な品種間、あるいは雑種の間で交雑を繰り返して、より優れた品種を作り上げますが、有用な形質を保有する遺伝資源との交雑が優れた品種を作り上げる鍵となります。
トマトの祖先種であるSolanum pimpinellifoliumとSolanum lycopersicum var. cerasiformeは、いずれも現在栽培されているトマトとの交雑が可能です。これらの種は、環境ストレスへの耐性など、栽培種トマトにはない優れた形質を含んでおり、品種改良における有用遺伝子のドナーとなり得ますが、そのゲノム情報は、あまり明らかにされていませんでした。
そこで本研究チームは、長いDNAを解読する技術(ロングリードシーケンシング)を用いて、この二つの祖先種のゲノム情報を高精度に解読し、ゲノムにコードされる遺伝子情報を明らかにしました。本研究成果は、今後、有用な遺伝子を同定する手がかりとなり、トマトの品種改良に貢献すると期待されます。
本研究は、イノベーション創出強化研究推進事業(30010A)、科研費・基盤B(17H03761)、科研費・特別研究員奨励費(18J20505)、先進ゲノム支援(16H06279)のプロジェクトの一環として実施されました。
本研究成果は、2021年1月20日にDNA Researchに掲載されました。
遺伝研の貢献 ロングリードが取得できるPacific Biosciences社のSequelシステムを用いて、トマトの祖先と考えられる2つの野生種ゲノムのショットガンシーケンスや機能遺伝子の全長配列を決定し、ゲノム解読の基盤となる情報を整備しました。本解析は2019年度先進ゲノム支援の支援課題としておこなわれたものです。

図: 本研究でゲノム配列を決定した二つのトマト祖先種(SP: Solanum pimpinellifolium, SLC: Solanum lycopersicum var. cerasiforme) (A)成熟果実、(B) 成熟果実の横断面、(C) 花序の形態
国立遺伝学研究所建物等警備業務
抗がん性成分を生産する植物チャボイナモリの全ゲノムを高精度に解読
Press release
Chromosome-level genome assembly of Ophiorrhiza pumila reveals the evolution of camptothecin biosynthesis
Amit Rai, Hideki Hirakawa, Ryo Nakabayashi, Shinji Kikuchi, Koki Hayashi, Megha Rai, Hiroshi Tsugawa, Taiki Nakaya, Tetsuya Mori, Hideki Nagasaki, Runa Fukushi, Yoko Kusuya, Hiroki Takahashi, Hiroshi Uchiyama, Atsushi Toyoda, Shoko Hikosaka, Eiji Goto, Kazuki Saito and Mami Yamazaki Nature Communications 12, 405 (2021) DOI:10.1038/s41467-020-20508-2千葉大学、理化学研究所、かずさDNA研究所、国立遺伝学研究所の研究チームは、抗がん剤の原料となるカンプトテシンを生産する薬用資源植物、チャボイナモリの全ゲノム配列を染色体レベルで高精度に解読しました。さらに他の植物のゲノムと比較することにより、カンプトテシンならびに類縁化合物の生産能力がどのように進化してきたかを明らかにしました。
本研究成果は、なぜ植物が薬になる成分を作るようになったのか、という根本的な疑問を解明するとともに、今後、抗がん剤原料(カンプトテシン)の持続的生産に寄与することが期待されます。
本研究は、2021年1月15日(日本時間)にNature Communicationsに掲載されました。
遺伝研の貢献 チャボイナモリゲノムのショットガンシーケンス(ロングリードとショートリード)やHi-C解析データなどゲノム解読の基盤となる情報を整備しました。 本解析は2017年度先進ゲノム支援の支援課題としておこなわれたものです。

図: チャボイナモリ
The NIG Webinar: AID2 enables rapid target protein degradation in living mammalian cells and mice
The NIG international webinar by Prof. Kanemaki will be held on February 2nd at 4 pm, Eastern Time (on February 3rd at 6:00 am, Japan Standard Time). He will talk about a new genetic tool that enables rapid and reversible depletion of target proteins in living cells and animals. A Zoom link of the webinar will be obtained by free registration at the following URL (https://rois.zoom.us/meeting/register/tJAudOuprD4oHdV4dRlS6mAIdKX2F-jlAIEJ).
Time and Date:
Eastern Standard Time (EST): 4:00 pm, February 2nd
Pacific Standard Time (PST): 1:00 pm, February 2nd
Greenwich Mean Time (GMT): 9:00 pm, February 2nd
Central European Time (CET): 10:00 pm, February 2nd
Japan Standard Time (JST): 6:00 am, February 3rd
Registration (Zoom URL will be obtained by the free registration):
https://rois.zoom.us/meeting/register/tJAudOuprD4oHdV4dRlS6mAIdKX2F-jlAIEJ
Title:
AID2 enables rapid target protein degradation in living mammalian cells and mice
Speaker:
Prof. KANEMAKI, Masato
Molecular Cell Engineering Laboratory
National Institute of Genetics
Summary:
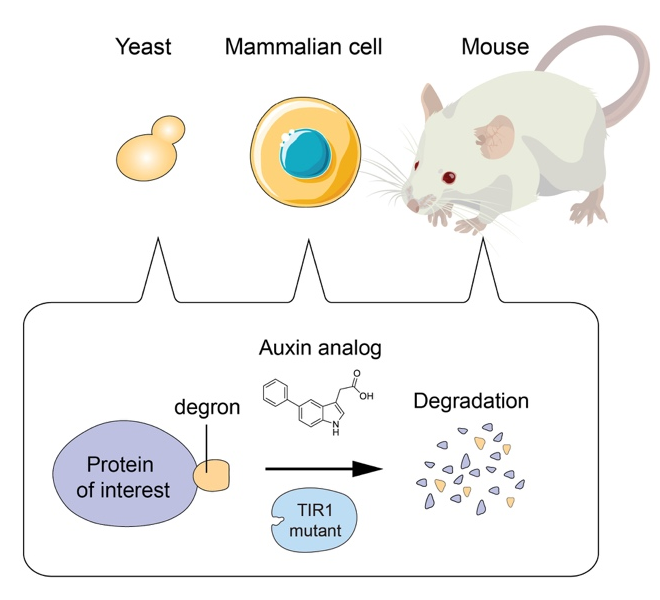
Genetic perturbation is a powerful way to analyze the function of proteins in living cells. For this purpose, we pioneered to develop the auxin-inducible degron (AID) technology by which a degron-fused protein can be rapidly degraded after the addition of the plant hormone auxin (Nishimura et al., Nat. Methods, 2009). By combining with CRISPR-based genome editing, it was possible to generate AID conditional mutants of human cells (Natsume et al., Cell Reports, 2016). The AID system became one of the popular genetic tools to study the function of proteins. However, leaky degradation and high doses of auxin for inducing degradation have been major drawbacks. Moreover, nobody has successfully applied the AID system to control protein degradation in living mice. We recently overcame these problems by taking advantage of chemical biology and successfully established the AID2 system (Yesbolatova et al. Nature Communications, 2020). By using AID2, we can now sharply control protein degradation in yeast, mammalian cells and mice.
• Link to KANEMAKI laboratory
• EurekAlert! link about the paper, Yesbolatova et al. Nature Communications, 2020
• Link to an interview of Prof. KANEMAKI
Chairperson: MAESHIMA, Kazuhiro















