Archive
- Home
- 2026/3
- 2026/2
- 2026/1
- 2025/12
- 2025/11
- 2025/10
- 2025/9
- 2025/8
- 2025/7
- 2025/6
- 2025/5
- 2025/4
- 2025/3
- 2025/2
- 2025/1
- 2024/12
- 2024/11
- 2024/10
- 2024/9
- 2024/8
- 2024/7
- 2024/6
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
嗅覚入力に依存した脳左右半球での非対称的な神経細胞細胞の特異化
Kishimoto, N., Asakawa, K., Madelaine, R., Blader, P., Kawakami, K., and Sawamoto, K.
Nature Neuroscience 16, 884-888, 2013 doi:10.1038/nn.3409
脊椎動物の脳は左右非対称です。今回私たちは、モデル脊椎動物ゼブラフィッシュにおいてトランスポゾンを用いた遺伝子トラップ法による大規模なスクリーンを行い、脳の左右半球において非対称的にGFP(Gal4)を発現するトランスジェニックゼブラフィッシュを見出しました。このトランスジェニックフィッシュにおいては、zinc finger蛋白質をコードするMyt1遺伝子内にトランスポゾンが挿入され、Myt1発現神経細胞を可視化することができました。Myt1発現細胞は、脳室帯—脳室下帯で新生され嗅球に移動する一群の細胞に含まれており、左半球により多く存在していました。私たちは、Notchシグナルが右側の脳室帯—脳室下帯におけるMyt1陽性神経細胞の産生を負に制御していること、ゼブラフィッシュの左側の鼻を塞ぐと左半球におけるNotchシグナリングが亢進しMyt1陽性神経細胞が減少すること、Notchシグナリングを阻害すると左側の鼻に依存した行動に異常が見られること、を明らかにしました。このことから、嗅覚入力がNotchシグナルを介して機能的神経細胞の左右脳半球における非対称的な新生を制御しているモデルを提唱しました。
この研究は名古屋市立大学・岸本博士、澤本博士との共同研究として行なわれました。
ゼブラフィッシュ終脳、脳室帯—脳室下帯において新生されるMyt1陽性細胞の左右非対称な分布。
血流により誘導されるマイクロRNAが心臓弁形成を制御する
Toshihiro Banjo,Janin Grajcarek,Daisuke Yoshino,Hideto Osada,Kota Y. Miyasaka,Yasuyuki S. Kida,Yosuke Ueki,Kazuaki Nagayama,Koichi Kawakami,Takeo Matsumoto,Masaaki Sato & Toshihiko Ogura
Nature Communications 4,Article number:1978,2013 doi:10.1038/ncomms2978
心拍や血流は、正常な心臓形成のために重要なはたらきをしています。発生過程において、心拍を停める、あるいは血流を攪乱すると心臓形成に異常を生じます。今回私たちはモデル脊椎動物ゼブラフィッシュを用いて研究を行い、マイクロRNAのひとつ、miR-21が血流依存的に心臓内皮に発現していることを見出しました。さらに、miR-21の機能を阻害すると心臓の弁形成が正常に行なわれないこと、miR-21の新しいターゲットのひとつがSprouty2であることを明らかにし、miR-21は、MAPキナーゼカスケードの阻害活性をもつSprouty2の発現を抑制することにより、正常な心臓弁形成に重要な役割を果たしている、というモデルを提唱しました。
本研究は、東北大学加齢医学研究所・小椋博士との共同研究として行なわれました。
A:ゼブラフィッシュ野生型胚における正常な心臓弁形成。
B:miR-21の機能が阻害されたゼブラフィッシュ胚における異常な心臓弁形成。
細胞分裂のタイミングの調節による球菌から桿菌への復帰
Daisuke Shiomi, Hironori Niki
MicrobiologyOpen (DOI: 10.1002/mbo3.116)
細菌ごとに形や大きさは決まっている。大腸菌や枯草菌は、長楕円の形、すなわち桿状をしており「桿菌」と呼ばれている。そして桿状の形態は、細胞壁により維持されている。細胞壁は抗生物質の標的であることから、細菌の形態維持の機構は盛んに研究されるようになった。私達の研究室では桿状形態を制御する因子であるRodZについて研究を行なってきた。RodZが欠損した大腸菌は、球状の形態に変化する。この球状に変化した大腸菌から、再び桿状に戻った復帰変異体を分離し調べることによって、桿状形態の制御の仕組みについて調べている。今回、復帰変異体の一つが細胞分裂の制御に関わるZipA蛋白質の発現変異により、球状から桿状に戻ることを明らかにした。ZipAは細胞分裂の際に、分裂面の形成時期を決める。ZipA蛋白質の発現が増加すると、分裂面の形成が遅れるのである。しかし、遅れている間も細胞の伸長はつづき、分裂が遅れた分だけ通常よりも長い細胞になるのである。このように細胞分裂の遅延により、球菌から桿菌への形態の復帰が起きたのである。自然界にはストレプトコッカスのような極性をもったレンサ球菌がいるが、今回の発見はこのタイプの球菌から桿菌は生じる仕組みとして適用できるものである。
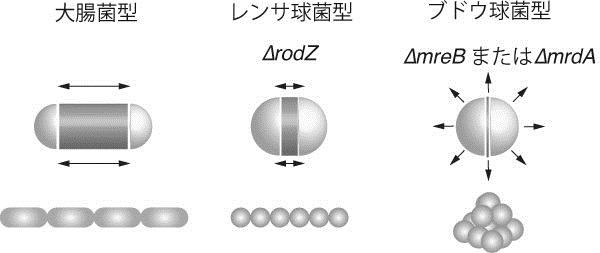
大腸菌型は、細胞の中央領域の細胞壁が伸長する。また細胞極性は常に一定であるため、分裂した細胞は直鎖上に並ぶ。レンサ球菌型は半球の間の細胞壁がわずかに伸長するだけである。RodZ欠損株もこの型である。他方、ブドウ球菌型は細胞全体が膨らみ、また細胞極性も変わるため分裂した細胞は固まりとなる。大腸菌でもMreBやMrdAの欠損株がこの型となる。
細胞核内のDNAの濃度が染色体の凝縮に影響する
Yuki Hara, Mari Iwabuchi, Keita Ohsumi, Akatsuki Kimura
Mol. Biol. Cell August 1, 2013 vol. 24(15) 2442-2453 doi: 10.1091/mbc.E13-01-0043
私たちヒトをはじめとする真核生物において、遺伝情報を担う染色体は、細胞が分裂する際には凝縮し、一本一本の染色体が「分裂期染色体」と呼ばれる棒状の構造を形成します。同じ個体の中でも、DNAの塩基配列の長さは変わらないのに、分裂期染色体の長さは変わることが知られています。その理由としてこれまで、発生ステージによって染色体を凝縮させる蛋白質の種類や量がかわることによることがわかっていました。今回、細胞建築研究室の原裕貴(総研大遺伝学専攻、現在はドイツEMBL研究員)と木村暁は、名古屋大学の大隅圭太博士、岩渕万里博士との共同研究により、染色体凝縮が「細胞核内のDNAの濃度」により制御されるとする新しいメカニズムを提唱しました。まず、線虫C. elegansを用いて、初期胚発生過程において、細胞核が徐々に小さくなる過程で、分裂期染色体の長さも徐々に短くなることを見いだしました。遺伝子操作により細胞核を大きくしたり小さくしたりすると、それに相関して同じ発生ステージでも分裂期染色体も長くなったり短くなったり変化しました。さらには、細胞核内のDNAの量を減らすと分裂期染色体は長くなり、DNA量を増やすと分裂期染色体は短くなることも明らかにしました。このことは、間期核内で染色体一本あたりの核の大きさが大きいほど分裂期染色体は長くなることを示しています。この制御の一般性を検討するためにカエル卵の無細胞系を利用して細胞核の大きさを小さくしてから分裂期染色体を形成させると、やはり分裂期染色体は通常よりも短くなることを見いだしました。以上の観察は、染色体の凝縮が核の大きさやDNAの量と言った物理的な制約に影響をうけることを示す新たな知見です。
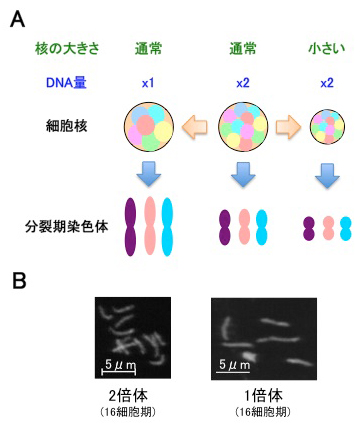
(A)細胞核内DNA濃度が染色体凝縮に影響する。核が小さくなると分裂期染色体も短くなる。核内の染色体DNAの量が減ると、分裂期染色体は長くなる。
(B)分裂期染色体の例(線虫のサンプル)。
トランスポゾンのコードする抗サイレンシング因子を発現させるとトランスポゾンが動いた
Yu Fu, Akira Kawabe, Mathilde Etcheverry, Tasuku Ito, Atsushi Toyoda, Asao Fujiyama, Vincent Colot, Yoshiaki Tarutani, Tetsuji Kakutani
EMBO Journal, advance online publication 30 July 2013; doi:10.1038/emboj.2013.169
ゲノム中を動く配列であるトランスポゾン(TE)はゲノムの進化に大きな影響を与えるが、潜在的に有害であり、そのほとんどはDNAメチル化などのエピジェネティックな機構で抑制されている。
今回、宿主のエピジェネティックな抑制機構に対抗する活性を持つTEについて報告する。シロイヌナズナで飛雲(Hi )と呼ぶTEは可動性であるが末端の逆位反復配列が壊れている。
Hi は正常個体では抑制されているがDNAメチル化がなくなるとゲノム中を動く。
Hi 配列をゲノム中に導入すると、もともとあったHi コピーが動く。また、導入配列はもとの配列の転写活性化と脱メチル化を引き起こす。
さらに、このような活性化は転移を触媒する酵素でなく別のタンパク質に仲介されている。
この抗サイレンシング因子と構造の似たタンパク質は、末端逆位配列の壊れた一群のTEに広く分布しており、これらのTEの自然集団中での増殖に貢献しているかもしれない。
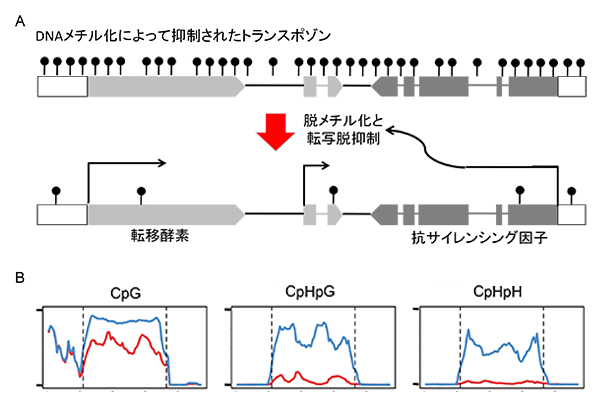
(A) トランスポゾンHiun (Hi , 飛雲)の構造。このトランスポゾンは転移酵素と抗サイレンシング因子をコードする。抗サイレンシング因子は脱メチル化と転写活性化を引き起こす。
(B) 非形転換体(青)とHi を発現する形質転換体(赤)での内在Hi 配列のDNAメチル化。Hi 配列の導入で内在コピーのDNAメチル化が低下する。
ゼブラフィッシュ側線神経系において Wnt/Dkkの負のフィードバック機構が感覚器のサイズを制御する
H. Wada, A. Ghysen, K. Asakawa, G. Abe, T. Ishitani, K. Kawakami.
Current Biology, 23 (16), p1559–1565, DOI: 10.1016/j.cub.2013.06.035
体の様々な器官は幹細胞を持ち、細胞が新しく置き換わったり、受けた損傷を修復したりして器官を一定の大きさに保ちます。どのようにして、器官は一定のサイズを認識するのでしょうか。
ゼブラフィッシュの側線器官(感丘)は、中央の感覚細胞(有毛細胞)とそれを取り囲む支持細胞からなります(図A)。感丘は、出芽によって増え、クラスターを形成します。すべての感丘は、同じ大きさをしています。我々は、Wntシグナル活性が、増殖細胞で高いこと(図C)、一方、感覚細胞がWntシグナルの抑制因子Dkk2を発現することを見出しました(図D)。Wntシグナル、Dkkシグナル、それぞれの機能阻害・獲得実験から、Wntシグナルは細胞の増殖を促進し、DkkはWntシグナルを阻害することによって細胞増殖を抑えることがわかりました(図B)。つまり、Wnt/Dkkは負のフィードバック・ループを形成し、器官の大きさを一定に保っています。負のフィードバックは、多くの恒常的システムに必須のコンポーネントです。本研究は、器官サイズ調節における、負のフィードバックの分子機構を明らかにしました。
本研究は、科学技術振興機構さきがけの助成のもと行われました。
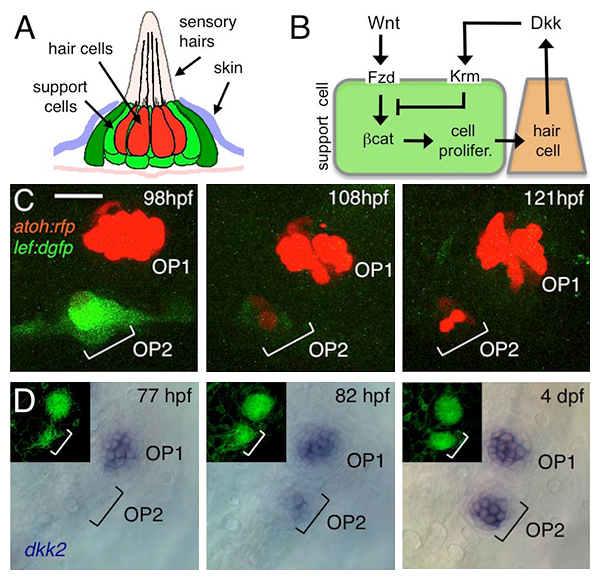
(A)ゼブラフィッシュ側線器官(感丘)模式図。
(B)Wnt/Dkkシグナルの作用機序。
(C)出芽細胞におけるWntシグナル活性(緑)が、有毛細胞(赤)の形成と共に失われる様子。
(D)有毛細胞ではWnt抑制因子Dkk2が発現する。
シーラカンス5頭の全ゲノム配列の解読に成功
![]()
Coelacanth genomes reveal signatures for evolutionary transition from water to land.
Nikaido et al.
Genome Research 22 July 2013 in press
生きた化石と呼ばれている希少種シーラカンスの全ゲノム配列(約27億塩基対)の決定に、東京工業大学、国立遺伝学研究所、東京大学などの研究チームが成功しました。
今回、研究チームは、タンザニア産3頭、コモロ産とインドネシア産のそれぞれ1 頭のシーラカンス(現存する全2種を網羅)について全ゲノム配列を決定し、大規模な比較ゲノム解析を実施しました。
特に、四肢形成や嗅覚に関連する遺伝子について詳しく調べたところ、シーラカンスは水中で生息する魚類であるにも関わらず、そのゲノム中には陸生の四足動物(カエルや哺乳類など)に特徴的な遺伝子がすでに数多く存在していることが明らかになりました。
これは、かつて水中に生息していた四足動物の祖先となる魚類が徐々に陸上化を遂げていくにあたり、ゲノムレベルではどのような変化が起きていたのかを知るための重要なてがかりを与えるものと考えられます。
また、シーラカンスの遺伝的多様性が極めて低いことも明らかとなり、この希少種の絶滅回避に向けた本格的な保全活動が急務であることが示されました。

今回のゲノム解読に用いた個体と同腹のシーラカンス稚魚
(国際科学振興財団主席研究員 岡田典弘(東工大名誉教授)提供)
太平洋クロマグロ全ゲノムを解読、クロマグロは微妙な青緑色の違いを認識
![]()
Evolutionary changes of multiple visual pigment genes in the complete genome of Pacific bluefin tuna
Yoji Nakamura・Kazuki Mori・Kenji Saitoh・Kenshiro Oshima・Miyuki Mekuchi・Takuma Sugaya・Yuya Shigenobu・Nobuhiko Ojima・Shigeru Muta・Atushi Fujiwara・Motoshige Yasuike・ Ichiro Oohara・Hideki Hirakawa・Vishwajit Sur Chowdhury・Takanori Kobayashi・Kazuhiro Nakajima・Motohiko Sano・Tokio Wada・Kosuke Tashiro・Kazuho Ikeo・Masahira Hattori・Satoru Kuhara・Takashi Gojobori・Kiyoshi Inouye
PNAS July 2, 2013 vol. 110 no. 27 11061-11066 doi: 10.1073/pnas.1302051110
水産総合研究センター、東京大学、九州大学と行ってきた共同研究の成果が、6月18日付けの米国科学アカデミー紀要オンライン版に掲載されました。
太平洋クロマグロの全ゲノムを解読し、他の魚類とは異なり、青~緑色がよく見えるように視覚の遺伝子が進化しているという特徴を明らかにしました。
独立行政法人水産総合研究センター(FRA)では、平成21年度より東京大学、九州大学および国立遺伝学研究所と共同で、世界初となる太平洋クロマグロ(以下「クロマグロ」)の遺伝情報全体であるゲノムの全ての塩基配列の解読に取り組み、クロマグロの全ゲノムの解読に成功しました。
解読したゲノムの解析により視覚にかかわる遺伝子にクロマグロに特有の特徴を発見しました。これは、海洋表層を高速で泳ぎ回るクロマグロが、赤色の乏しい海中の景色の中で微妙な青緑色の違いを認識できることを示していると考えられます。
本成果は、クロマグロの行動特性に関する基礎的な知見を与えるものであり、今後のまぐろ養殖生産技術の改善にもつながるものです。
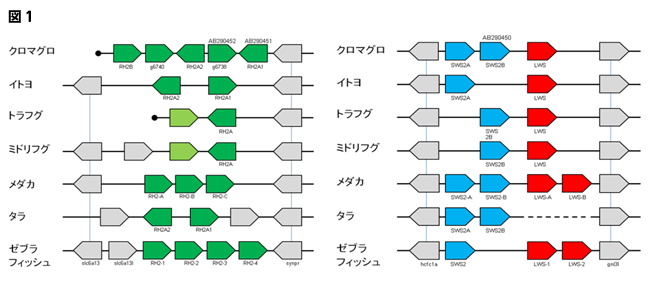
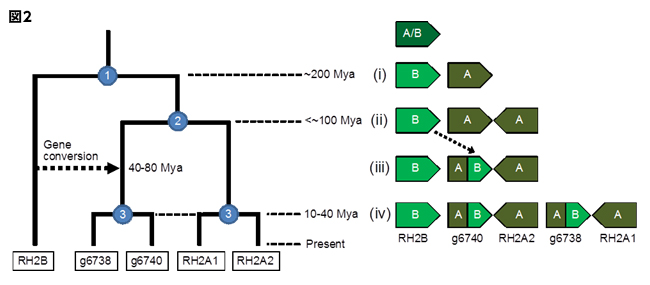
図1 クロマグロおよび他の魚種のゲノム上に存在する可視光感受性オプシン遺伝子。緑オプシン遺伝子(左)、青および赤オプシン遺伝子(右)。図中の遺伝子の色は吸収する可視光の色に対応し、向きは転写の方向を示します。
同色のオプシン遺伝子であっても各オプシン遺伝子の吸収波長は微妙に異なるため、ロドプシン遺伝子数の増加により、より微妙な色の違いが認識できるようになると推定されます。
フグ類2種の黄緑色は、緑オプシンの機能を失った偽遺伝子であることを表します。また、青色と赤色のオプシン遺伝子はゲノム上で隣り合って存在しています。灰色の遺伝子はオプシンとは無関係の遺伝子ですが、縦線で結ばれたものは各魚種で保存されている遺伝子同士であることを示しています。
図2 クロマグロの5つの緑オプシン遺伝子の推定進化シナリオ。両図とも上から下(現代)に年代が進行します。(①の進化が生じたのは約2億年前と推定)。 A/BはAとBの共通の祖先を、系統樹中の①-③の数字は、遺伝子の倍加が起こったことを示します。③では2つの遺伝子が同時に倍加して4つになったと推定されています。点線の矢印は遺伝子変換(gene conversion)のイベントを示します。 それぞれのイベントに対応する遺伝子構造の変化を図の右側に示しています。各イベント(遺伝子重複:i,ii,iv、遺伝子変換:iii)に対して、起きた時期が推定されています(単位は百万年前=Mya)。
マウスゲノム内の超複雑な繰り返し領域の解析(新領域融合プロジェクトの成果です)
Umemori J, Mori A, Ichiyanagi K, Uno T, and Koide T.
BMC Genomics14: 455, 2013.
コピー数多型 (Copy number variation, CNV)は、ヒトおよびマウスゲノムの10%以上を占め、個体や系統間の遺伝的な多様性を決める重要な要素であると考えられています。
CNVはしばしば集合して、コピー数多型領域(complex CNV region, complex CNVR)を構成し、ゲノムの部分重複(Segmental duplication, SD)と呼ばれる、複雑な繰り返し配列を持つ領域をつくりだしています。
しかし、その非常に複雑な領域の解析は、ゲノム配列のほとんどが解読された現在においても、困難を極めています。今回我々は、国立情報学研究所との共同研究により、この複雑な領域の新たな解析方法を確立しました。
先ず我々は、国立情報学研究所、宇野毅明准教授が開発した、高速相同性検出アルゴリズム、Similarity/Homology Efficient Analyze Procedure (SHEAP)を用いることにより、マウスのlarge SD領域をゲノムワイドに検出しました。
その結果、large SDは、それぞれが独自の「タータンチェック模様」として視覚化されました (図1)。次に、遺伝的不適合性の原因領域である、第13番染色体上のSD (SD13M)に着目し(投稿準備中)、独自に開発した画像解析プログラム、blast-based Systematic analysis of HErPlot to Extract Regional Distinction (SHEPHERD)を用いて、繰り返しのコアとなっている配列を同定しました (図2)。
さらに、この領域について比較ゲノムハイブリダイゼーション(Array-comparative genomic hybridization, aCGH)を三島の野生マウス由来の系統MSMとブルガリアの野生マウス由来系統BLG2、実験用マウス系統C57BL/6Jについて行い、それぞれのコア配列の系統間のCNVを実験的に検出しました。この結果、これらのコア配列は、CNVsを持つものと、持たないものに分けられること、これらは含有配列などの性質が大きく異なることが示唆されました。
これらの解析結果は、今後complex CNVRの進化や機能を明らかにするための足がかりになると期待されます (図3)。
本研究は、情報・システム研究機構、新領域融合プロジェクト・育成融合プロジェクトの助成のもと行われました。
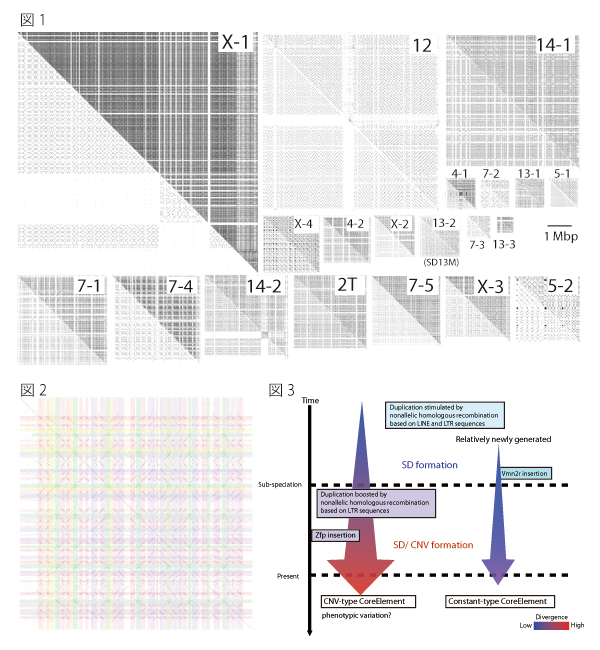
図1 SHEAPにより視覚化されたSDのタータンチェック構造
左上の数字はSDの染色体番号を示している。セルフプロットで示されたそれぞれのSDの左下はトランスポゾンなどの既知の配列をマスクしたもの、右上はマスクしていない配列を用いて解析を行なったもの。SD内の斜線は、長い繰り返し配列を表している。
図2 セルフプロットで示されたSD13Mと反復単位の抽出
斜線として視覚化された繰り返し配列について、X軸方向とY軸方向で重複するものを画像解析により間引き、コアとなる配列を選別する。
図3 SD13Mの進化および機能についての仮説
CNVsを持つコア配列は、LTR型のレトロトランスポゾンの割合およびDivergenceが高い。これは、CNVsを持つコア配列は古い年代からそのコピー数を増加させ、LTR型のレトロトランスポゾンの挿入による非アリル性の相同組換えが、そのコピー数の変動を加速させた可能性を示唆している。
DNA複製のスタート地点の決定における、クロマチンの役割
Kohji Hizume, Masaru Yagura, and Hiroyuki Araki
Genes to Cells , 24 JUN 2013 DOI: 10.1111/gtc.12073. [Epub ahead of print]
真核生物のDNAの複製は、染色体上に点在する複製開始点と呼ばれる領域からスタートします。その複製開始点には、origin recognition complex (ORC)と呼ばれるタンパク質複合体が特異的に結合して、複製開始に必要な他のタンパク質が複製開始点に集合する目印となります。これまで、ORCと複製開始点との特異的な結合には、クロマチンが何らかの役割を示していることが示唆されてきましたが、ORCとクロマチンとの結合を試験管内で直接調べ、その役割を明らかにする研究は行われてきませんでした。
私たちは、試験管内再構成したクロマチンとORCとの結合の様子を生化学的に解析し、さらに原子間力顕微鏡(atomic force microscopy, AFM)によるORC–クロマチン複合体の観察を行いました。その結果、複製開始点が裸のDNAであるよりもクロマチンファイバーを形成しているほうが、ORCがより安定かつ特異的に複製開始点に結合することを見出しました。また、ORCの結合は、複製開始点付近のヌクレオソーム配置に変化を促し、複製開始点がリンカーDNA領域となる様子を検出しました。これらのことから、ORCが、リンカーDNAとなった複製開始点と結合し、さらに近傍のヌクレオソームとも結合を確立して、安定かつ特異的なORC–複製開始点結合を確立する仕組みが解りました。
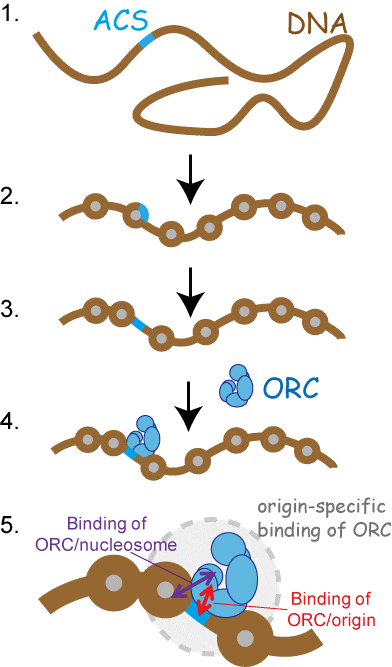
(1) 複製開始点特異的配列(ACS)を含むDNA。
(2) クロマチンを形成させるとACS上にヌクレオソームが配置される。
(3) ORCの添加によりACSがリンカーDNAとなる。
(4) リンカーDNAとなったACS上にORCが結合する。
(5) ORCは、リンカーDNAとなったACS配列との結合と、近傍のヌクレオソームとの結合両方を介して安定に複製開始点と相互作用する。
サンゴ礁の白化現象の原因の解明に向け一歩前進
![]()
Draft assembly of the Symbiodinium minutum nuclear genome reveals dinoflagellate gene structure
Eiichi Shoguchi, Chuya Shinzato, Takeshi Kawashima, Fuki Gyoja, Sutada Mungpakdee, Ryo Koyanagi, Takeshi Takeuchi, Kanako Hisata, Makiko Tanaka, Mayuki Fujiwara, Mayuko Hamada, Azadeh Seidi, Manabu Fujie, Takeshi Usami, Hiroki Goto, Shinichi Yamasaki, Nana Arakaki, Yutaka Suzuki, Sumio Sugano, Atsushi Toyoda, Yoko Kuroki, Asao Fujiyama, Mónica Medina, Mary Alice Coffroth, Debashish Bhattacharya, and Nori Satoh
Current Biology, 11 July 2013 DOI:10.1016/j.cub.2013.05.062
プレスリリース資料
(OIST 沖縄科学技術大学院大学ニュースセンター「サンゴ共生カッチュウソウのゲノム解読」)
サンゴは、クラゲやイソギンチャクの仲間であるサンゴ虫といわれる生き物が本体です。サンゴは環境の変化などにより白化現象が発生しますが、これはサンゴの細胞の中に共生するカッチュウソウが減少することが原因です。サンゴは、カッチュウソウの光合成に頼ってエネルギーを補給しているので、カッチュウソウが失われるとサンゴは白化し、死滅するといわれています。しかし、なぜカッチュウソウが失われていくのか、詳しいことは不明でした。
今回、国立遺伝学研究所(大学共同利用機関 情報・システム研究機構)は、沖縄科学技術大学院大学(OIST)、独立行政法人理化学研究所、東京大学大学院新領域創成科学研究科と共同研究にて、カッチュウソウの全遺伝子解析(ゲノム解析)を行いました。ゲノムとは、ある生物の全遺伝情報のことで、ゲノム解読は生物の設計図を手に入れたといっても過言ではありません。2003年のヒトゲノム解読により、その後のヒト遺伝子研究が飛躍的に進んだことと同様、今後、サンゴとカッチュウソウがどのように共生し、温度変化や海水汚染などにより、どのようにサンゴの白化が引きおこされるか、解明できることが期待されます。
今回、ゲノムの解読は、三島市にある国立遺伝学研究所(遺伝研)にて行われました。すでに遺伝研では、サンゴ、シーラカンス、イネなどのゲノム解読の功績があり、今後とも生物のゲノム解析に大きな貢献が期待できます。
本研究成果は、文部科学省科学研究費新学術領域研究「ゲノム支援」(代表 小原雄治)の支援を得ました。
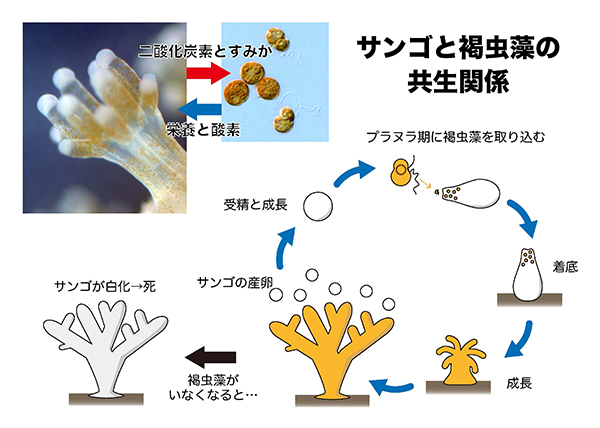
サンゴとカッチュウソウの共生関係
半開放型の核分裂に必要な核膜動態制御
Keita Aoki, Yuh Shiwa, Hiraku Takada, Hirofumi Yoshikawa and Hironori Niki
Genes to Cells 21 JUN 2013 DOI: 10.1111/gtc.12072. [Epub ahead of print]
真核細胞の分裂期は、核膜が崩壊し染色体が細胞内で露になる開放型の核分裂と、核膜が崩壊しない閉鎖型の核分裂の2つがあります。しかし真菌類ではその中間型も存在します。
カンジダ菌やジャポニカス分裂酵母では、核膜の一部分が消失する半開放型の核分裂が起きます。半開放型の核分裂は、開放型、閉鎖型両方の性質を備えているため、両者の特質を考える上でよい研究対象といえます。
私たちは、ジャポニカス分裂酵母を用いて、半開放型核分裂が起こるしくみについて研究を行っています。今回、半開放型核分裂には、脂肪酸合成因子が関わっていることを明らかにしました。
ジャポニカス分裂酵母の変異株ライブラリーから、半開放型核分裂が起こらない変異株を見つけました。その変異はE3ユビキチンリガーゼであるAPC/Cにありました。この変異株は増殖が高温で停止します。
この停止が手直しされ高温でも増殖できるようになったサプレッサー変異株とよばれるものを取り、調べました。サプレッサー変異は、脂肪酸合成因子Oar2に変異が起こっていました。
図のように、APC/C変異株では半開放型核分裂に異常がみられます。しかし、APC/C変異株にOar2の変異が加わったサプレッサー変異株では、その異常が部分的ですが修復されていました。
Oar2にはAPC/Cによって認識される配列がありました。また、Oar2のタンパク質の量がAPC/C変異株内では増量していました。
これらのことから、APC/CがOar2のタンパク質の量を制御すると思われます。脂肪酸合成因子Oar2は、核膜を作る脂質の合成に関係しています。
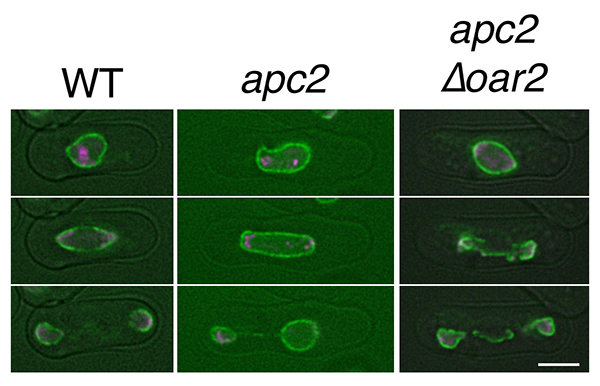
核膜の動態を、野生型、apc2変異株、apc2∆oar2変異株の中でそれぞれ観察した。野生型で見られた半開放型分裂は、apc2変異株で減退し、apc2∆oar2変異株では部分的に回復した。
緑:核膜マーカー(Cut11-GFP)、マゼンタ:染色体マーカー(H2A-mCherry)。スケールバー:5µm
線虫胚の紡錘体における幅、長さと染色体量の定量的な関係性
Yuki Hara, Akatsuki Kimura
Mol. Biol. Cell 24 (9), 1411-1419, 2013 doi:10.1091/mbc.E12-07-0528
遺伝情報を担う染色体を分配する装置である「紡錘体」は、「ひし形」を回転させたような美しい形をしています。大きい紡錘体でも小さい紡錘体でも似たような「ひし形」ができあがるので、ひし形の縦と横のサイズの比は細胞内で何らかの制御を受けていると予想されますが、そのような制御についてはわかっていませんでした。細胞建築研究室の原裕貴(総研大遺伝学専攻、現在はドイツEMBL研究員)と木村暁は線虫胚発生時に生じる様々なサイズの紡錘体を定量化しました。その結果、紡錘体の縦と横の長さが単純な比例関係(isometry)にはなく、「ひし形の横幅が、斜辺の約0.6乗に比例する」という、少し複雑だがきれいな関係性(allometry)があることを見いだしました。染色体の量を変えると、ひし形の横幅が染色体量の約0.4乗に比例することも見いだしました。これらの関係性から、原研究員らは紡錘体のひし形構造を制御する新たな力学的機構について提唱しました。
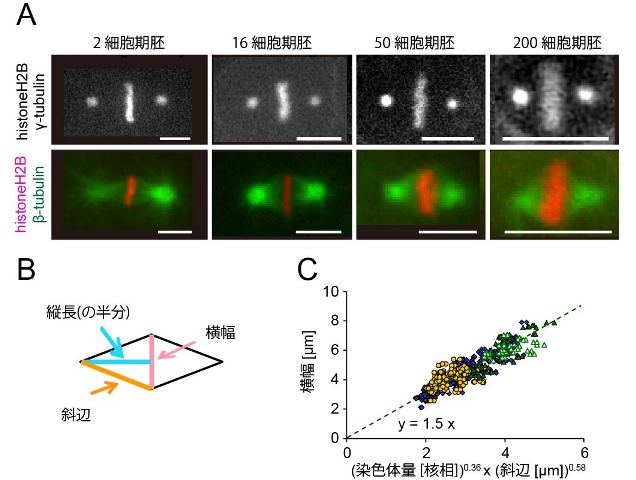
(A) 線虫胚における紡錘体の例(スケールバー=5 μm)。(B) 紡錘体の横幅(ピンク)、縦長の半分(青)と斜辺(オレンジ)の説明。(C) 通常(2倍体)の線虫(青)、1倍体の線虫(黄色)および多倍体の線虫(緑)について、紡錘体の横幅を染色体量(核相)の0.36乗と斜辺の0.58乗の積に対してプロットしたもの。(論文図を改変)
65回日本細胞生物学会大会で北川研究室・白土玄さん、前島研究室・野崎慎さんが同時受賞
 |
 |
|
| 受賞時の野崎さん | 受賞時の白土さん |
 第65回日本細胞生物学会大会
第65回日本細胞生物学会大会 新分野創造センター・北川研究室
新分野創造センター・北川研究室 構造遺伝学研究センター・前島研究室
構造遺伝学研究センター・前島研究室集団遺伝研究部門の隅山健太助教が第17回 日本細胞生物学会論文賞を受賞

- 論文名: Live Imaging of Protein Kinase Activities in Transgenic Mice Expressing FRET Biosensors
- Cell Structure and Function 37: 65-73(2012)
- 著者: Yuji Kamioka1, Kenta Sumiyama2, Rei Mizuno1, Yoshiharu Sakai1, Eishu Hirata1, Etsuko Kiyokawa1, Michiyuki Matsuda1
- 1京都大学, 2国立遺伝研
DNA複製:真核生物間で保存された、効率よい複製開始のためのタンパク間相互作用
Seiji Tanaka, Yayoi Komeda, Toshiko Umemori, Yumiko Kubota, Haruhiko Takisawa, and Hiroyuki Araki
Mol. Cell. Biol.(2013), 33 2614-2622. doi:10.1128/MCB.00431-13
真核生物の染色体DNA複製は、特定の領域(=複製起点)から開始します。染色体DNAは一度の細胞分裂周期の間に、一度だけ過不足なく正確に倍加されなくてはならないため、複製起点の活性化は細胞周期の間で厳密に制御されています。これまでの解析から、複製起点の活性化は大きく2段階の反応で起きることがわかってきました。すなわち、1) 細胞周期のG1期に、複製時ヘリカーゼ(DNA2本鎖巻き戻し酵素)が不活性型として複製起点にロードされ、2) S期ではヘリカーゼが活性型に変換し、DNA複製が開始します。活性型ヘリカーゼの形成は、タンパクキナーゼによるタンパクリン酸化をきっかけとした、複製起点上での新たなタンパク群の集合・リモデリングを含む複雑な反応です。私たちは、真核細胞のモデル細胞である出芽酵母を用いた研究から、Dpb11と呼ばれるタンパクがリン酸化された2種類のタンパクと特異的に結合することが、この反応の鍵となっていることを示してきました。
今回の研究では、このDpb11が、上記結合とは別の領域で活性型ヘリカーゼにのみ含まれるGINSというタンパク複合体と直接結合すること、この結合が効率よい複製起点の活性化に必要であることを見出しました。また、脊椎動物のDpb11ホモログであるTopBP1においても、酵母のDpb11同様、GINSと結合し複製開始に重要な働きをしている領域、すなわち機能的に同じ働きを持つ領域を見出しました。このことは、今回見出した Dpb11/TopBP1-GINS相互作用が真核生物のDNA複製開始において共通の重要な役割を果たしていることを示すものであり、今後、真核生物の染色体DNA複製開始の詳細なメカニズムの理解に役立つものです。
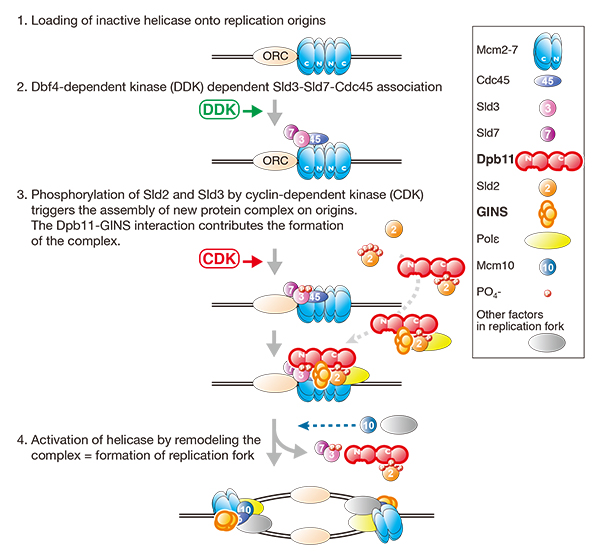
出芽酵母(S. cerevisiae)DNA 複製開始機構のモデル。Dpb11-GINSの相互作用が、複製開始に必要となる新たなタンパク複合体の形成に寄与している。
シースルー運動カラム
Kazuhide Asakawa, Gembu Abe, and Kawakami Koichi
Frontiers in Neural Circuits, 2013 May 28;7:100. DOI: 10.3389/fncir.2013.00100
私たちの行動は、600種類を超えるとも言われる大小様々な種類の筋肉が、特定の組み合わせでタイミングよく収縮・弛緩することで生まれます。この筋肉の収縮リズムを決めている脳や脊髄からの神経シグナルは、「運動ニューロン」と呼ばれる神経細胞を介して筋肉に伝えられます。このため、運動ニューロンの多様性や、筋肉との接続様式を明らかにすることは、行動のメカニズムを理解するのに必要なだけでなく、運動ニューロンの変性によって神経シグナルが筋肉に伝達されなくなってしまう疾患(運動ニューロン病)の病理を解明する上でもとても重要です。
我々は運動ニューロンの多様性や、筋肉との接続様式を明らかにする為に、左右およそ60個ずつの運動ニューロンを含む節(脊髄節)が30個連なってカラム構造を形成しているゼブラフィッシュ幼魚に着目しました。身体の透明性を活かして、幼魚を生かしたまま単一の脊髄運動ニューロンや、カラム構造全体を可視化する遺伝学的技術を開発することで、脊髄運動ニューロンの細胞形態と、体幹筋との位置的な対応関係を詳細に明らかにしました。本研究によって、脊椎動物の脊髄運動ニューロンのカラム構造の微細構造が明らかになっただけでなく、将来、仮想行動中の運動ニューロン群の活動リズムをリアルタイムで観察することや、病態生理学的解析に適した運動ニューロン病のモデルを構築することが実現すると期待されます。
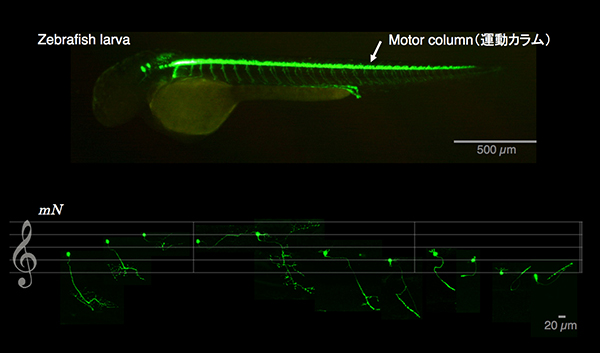
(上)ゼブラフィッシュ幼魚(zebrafish larva)の側面図。
(下)単一細胞染色によって同定された11種類の脊髄運動ニューロン。
これらの運動ニューロンの組み合わせで多様な運動リズムが生まれる。
日本産マウスのゲノム解読からみえてきた実験用マウス系統の起源
![]()
The ancestor of extant Japanese fancy mice contributed to the mosaic genomes of classical inbred strains
Takada, T., Ebata, T., Noguchi, H., Keane, K., Adams, D., Narita, T., Shin-I, T., Fujisawa, H., Toyoda, A., Abe, K., Obata, Y., Sakaki, Y., Moriwaki, K., Fujiyama, A., Kohara, Y. and Shiroishi, T.Genome Research, 2013 Aug;23(8):1329-1338. DOI: 10.1101/gr.156497.113
国立遺伝学研究所と理化学研究所からなる研究グループは、日本産マウス亜種(モロシヌス)由来の二つの系統、MSM/Ms(Mishima: MSM)及びJF1/Ms(Japan-Fancy 1: JF1)の全ゲノム解読を行い、 生物・医学研究分野で広く使われている標準的な実験用マウス系統のゲノム配列との比較解析を行いました。その結果、マウスゲノム多型情報が大幅に拡充されました。 さらに、この情報をもとに標準的な実験用マウス基準系統の成立史についての新知見が得られました。
MSM/Ms系統は静岡県三島市内で捕獲された日本産野生マウス亜種(モロシヌス)、また、JF1/Ms系統は25年ほど前にデンマークのペットショップで発見された日本産愛玩用マウスをもとにして、いずれも国立遺伝学研究所で樹立されました。 今回、これらのマウス系統の次世代シーケンサーによる全ゲノム解読を行い、C57BL/6 などの西ヨーロッパ産マウス亜種(ドメスティカス)由来である実験用マウスの基準系統の間で全ゲノム配列を比較解析しました。 この結果、両グループ間のゲノムの大半で1%程度のDNA配列上の違い(ゲノム多型)が見出されました。現在、多くの研究者がMSM/Ms、JF1/Msのゲノム多型性に起因する多様な形質の違い(表現型)の遺伝解析を進めています。 今回得られた日本産マウス亜種由来の両系統の全ゲノム配列情報や基準系統との間の多型情報は、マウス亜種間多型に基づくゲノム機能学を一層加速させるために重要な研究基盤となります。
さらに、実験用マウスの基準系統の比較ゲノム解析から、世界中で広く使用されている実験用マウス系統のゲノム中にJF1/Ms系統ときわめて類似度の高い配列が散在することを見出しました。 今回のゲノム解析の結果と日本やヨーロッパの古い文献の調査研究により、江戸末期に日本からヨーロッパに渡ったJF1/Msの祖先とヨーロッパ産愛玩用マウスの交配集団が、今日の実験用マウスの基準系統の起源となっていることがわかりました。 さらに、日本産マウスのゲノムが、実験用マウスの基準系統間の遺伝的多型に大きく貢献していることもわかりました。このように、本研究の成果によって、実験用マウス系統の成立に関する長い議論にも終止符を打つことができました。
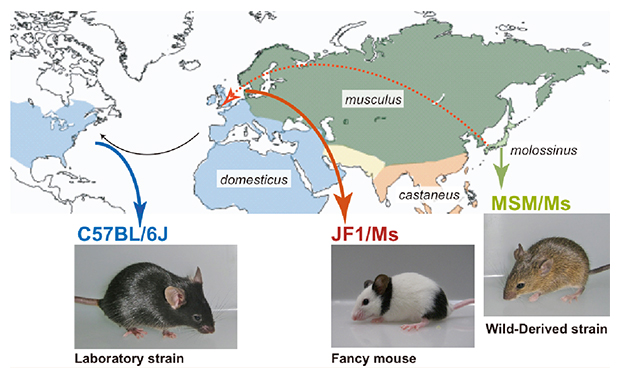
マウス亜種の世界的分布。実験用マウスの基準系統の成立に関与したとされる4つの亜種の分布域を地図上に色別してマップしました。 今回、日本産野生マウス由来のMSM/Ms系統および日本産愛玩用マウス由来のJF1/Ms系統をゲノム解読し、 それらの塩基配列を主に西ヨーロッパ産マウス亜種由来の実験用マウス系統であるC57BL/6Jの基準ゲノム配列と比較解析しました。
ヒト4番染色体長腕部分重複症の原因解明: Hand2遺伝子量効果による四肢・心臓の形態異常
Tamura, M., Hosoya, M., Fujita, M., Iida, T., Amano, T., Maeno, A., Kataoka, T., Otsuka, T., Tanaka, S, Tomizawa, S., and Shiroishi, T.
Human Molecular Genetics, 2013 Jun 15;22(12):2471-2481. DOI: 10.1093/hmg/ddt099
ダウン症候群などの染色体(部分)重複や欠損を伴う染色体異常疾患は、新生児の約1%に発症すると言われています。これらの疾患に特徴的な形態異常や神経症状は、遺伝子コピー数の変化によるものと考えられていますが、多くのケースではその原因遺伝子は特定されていません。
今回、哺乳動物遺伝研究室の田村勝助教(現理研バイオリソースセンター)、城石俊彦教授らのグループは、突然変異マウスRim4 がヒト染色体異常疾患である4番染色体長腕部分重複症のモデル動物であること、また多くの表現型を本疾患の患者さんと共有することを見出しました。更にRim4 とKnockoutマウスの交配実験による遺伝学的解析により、本疾患にみられる心臓中隔欠損や四肢形態異常が、bHLH型転写因遺伝子Hand2 の量的効果が原因であることを明らかにしました。この結果は、Hand2 の遺伝子量の形態形成への関与の解明に加えて、4番染色体長腕部分重複症表現型の分子メカニズムの理解や患者さんのQOL向上に貢献するものと期待されます。
本研究成果は、国立病院機構新潟病院富沢修一院長らのグループとの共同研究によるものです。
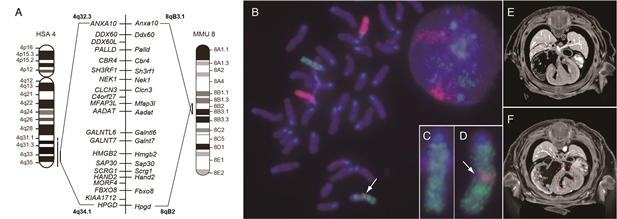
マウス変異Rim4にみられる染色体異常と心室中隔欠損
(A)ヒト4番染色体長腕 (HSA4) 4q32.3-34.1とマウス8番染色体 (MMU8) 8qB2-B3.1は、遺伝子の並びが良く保存されている。(B) Rim4/+マウスの染色体FISH像。6番染色体 (Chr6: 緑色)、8番染色体(Chr8: 赤色)。(C) 野生型Chr6。(D) Rim4 Chr6 の拡大像。矢印は挿入された8qB2-B3.1断片 。(E, F) 胸部micro-CT像。野生型 (E) にはみられない心室中隔欠損(点線丸)(F) がRim4/ Rim4では観察される。
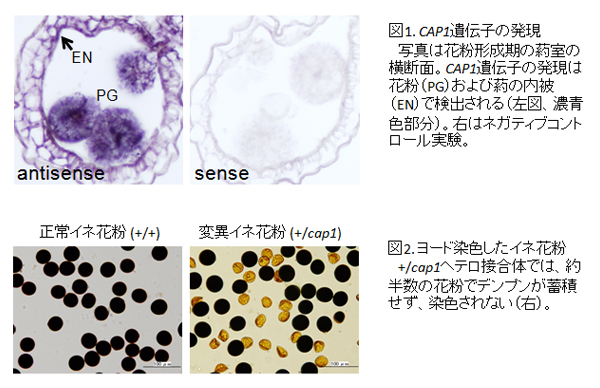
イネの花粉形成遺伝子を同定
Kenji Ueda, Fumiaki Yoshimura, Akio Miyao, Hirohiko Hirochika, Ken-Ichi Nonomura, and Hiroetsu Wabiko
Plant Physiology, 2013 Jun;162(2):858-871. DOI: 10.1104/pp.113.216523
栽培イネのゲノムに含まれる約3万2千個の遺伝子のうち、2万以上の遺伝子が花粉を含む葯で発現するといわれています。しかし、花粉形成過程における機能が解明された遺伝子は限られています。今回の研究では、花粉形成過程で特異的な働きをするイネ遺伝子の同定に成功し、COLLAPSED ABNORMAL POLLEN1 (CAP1 )遺伝子と命名しました。
被子植物の花粉は、栄養細胞のなかに2つの精細胞がとりこまれた、3細胞からなる特殊な構造をしています。CAP1 遺伝子は、精細胞が分裂する前の二細胞花粉期の葯で最も強く発現していました(図1)。遺伝子機能が欠損した花粉は、ほとんどの細胞成分が失われて花粉外壁(エキシン)だけの構造になり(図2)、花粉管伸長能が失われます。花粉形成以外のイネの成長には全く影響しないことから、CAP1 の花粉特異的な機能が明らかとなりました。
イネCAP1タンパク質は、植物のL-アラビノキナーゼとよく似ており、細胞壁代謝の過程で生じるL-アラビノースを再利用のためリン酸化している可能性が示唆されました。イネcap1変異体では、アラビノースが再利用されずに花粉内に異常に蓄積する、あるいは花粉の細胞壁代謝が阻害されて崩壊すると予想されます。遺伝子発現パターンがシロイヌナズナのアラビノキナーゼ様遺伝子の1つと酷似することから、CAP1 機能が被子植物で広く保存されており、花粉形成過程において非常に重要な役割を果たすことが示唆されました。
本研究は、秋田県立大学および農業生物資源研究所との共同研究であり、遺伝研共同研究経費(共同研究A、B)の助成を受けて実施されました。