Archive
- Home
- 2026/2
- 2026/1
- 2025/12
- 2025/11
- 2025/10
- 2025/9
- 2025/8
- 2025/7
- 2025/6
- 2025/5
- 2025/4
- 2025/3
- 2025/2
- 2025/1
- 2024/12
- 2024/11
- 2024/10
- 2024/9
- 2024/8
- 2024/7
- 2024/6
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
三島北高校の生徒が遺伝研を訪問
静岡県立三島北高校の生徒11名が12月24日、国立遺伝学研究所を訪問しました。分子生命史研究室の工樂樹洋教授による「DNAで探る生命の進化」をテーマとした講義のほか、サメが飼育されている工樂研の見学も行われました。
講義では、DNA配列を比較することで、外見だけでは判断できない生物どうしのつながりや、これまでにたどってきた進化の道筋を明らかにする研究について解説しました。さらに、実際のDNA配列データベースに多様な生物の情報が豊富に含まれる様子を紹介。系統樹を作成する演習や、生き物の系統関係を学ぶボードゲームを通して、生徒たちはDNAや進化に関する研究への理解を深めました。
工樂教授は講義の中で、「DNAの情報は次々と読み取られ、データベースとして誰でもウェブ上で利用できる時代になっています。しかし、まだ解明されていないことは数多く残されています。そうした情報をもとに、俯瞰した視点で研究を進めていける研究者が必要です」と語りました。
清水東高校の生徒が遺伝研を訪問
さあ取りたまえ、このゆらぎを速さに変えるのは、分子モーターだ
Active fluctuations of cytoplasmic actomyosin networks facilitate dynein-driven transport
Takayuki Torisawa, Kei Saito, Ken’ya Furuta, Akatsuki Kimura
iScience (2025) Volume 28, Issue 12 114096 DOI: 10.1016/j.isci.2025.114096
細胞が生きていくためには、適切な物質が適切なタイミングで、適切な場所へと運ばれることが不可欠です。このような細胞内での物質輸送の多くを担っているのが、「分子モーター」と呼ばれるタンパク質酵素です。分子モーターは、細胞の周縁から中心へ、あるいは中心から周縁へ、神経細胞から伸びる突起の中や、やがて個体へと成長する胚の中など、生物のさまざまな文脈で多様な物質を運んでいます。
分子モーターがはたらく環境は、「生きている」細胞の内部です。「生きている」ということは、細胞内では輸送に限らず、さまざまな反応や運動が同時並行的に起こり、分子レベルで常に力や動きが生み出されているということを意味します。このような動的で、ある意味ノイズに満ちた環境は、分子モーターによる輸送に少なからず影響を与えていると考えられてきました。
実際、分子モーターの一種である「ダイニン」による細胞内輸送では、細胞からタンパク質を取り出して余計なものを除いて観察したときと比べて、生きている細胞の中ではより速く進むことが知られていました。しかし、なぜさまざまな分子で満たされ混雑している生体内で、むしろ輸送速度が増すのか、そのメカニズムは明らかになっていませんでした。
今回、細胞建築研究室の鳥澤嵩征助教(筆頭・責任著者)と木村暁教授、物理細胞生物学研究室の斎藤慧助教(当時)、情報通信研究機構の古田健也研究マネージャーらの研究グループは、細胞質中に存在するアクチンという細胞骨格繊維とミオシンという分子モーターがつくるネットワーク(アクトミオシンネットワーク)が生み出す「動的なゆらぎ (active fluctuation)」に着目し、このゆらぎによって細胞質内に生じるランダムな力が、ダイニンによる輸送を加速している可能性を示しました (図)。
遺伝学的および生化学的手法を用いて細胞内のアクトミオシンのゆらぎの程度を操作したところ、その活動を低下させると輸送速度は減少し、逆に活性を高めると輸送が加速することが定量的に確認されました(図)。さらに、細胞から精製したタンパク質を用い、光ピンセットとよばれる装置によって、ダイニンが周りの環境から与えられる力にどのように応答するかを調べました。これらの結果を合わせることで、細胞内で生じるランダムな力が、ダイニンによって一方向への運動へとうまく変換されている可能性が示唆されました。
本研究は、生きた細胞の内部環境が、分子モーターによる輸送を単にさまたげる存在ではなく、むしろ積極的に利用され、輸送を促進しうることを示したものです。細胞の中で起こる動きに対する理解を深めるとともに、将来的には、細胞質のアクティブなゆらぎを活用したあらたな輸送制御の考え方へとつながることが期待されます。
本研究はJSPS科研費 18KK0202、19K16094、 24K09405、18H02414 の支援を受けて行われました。
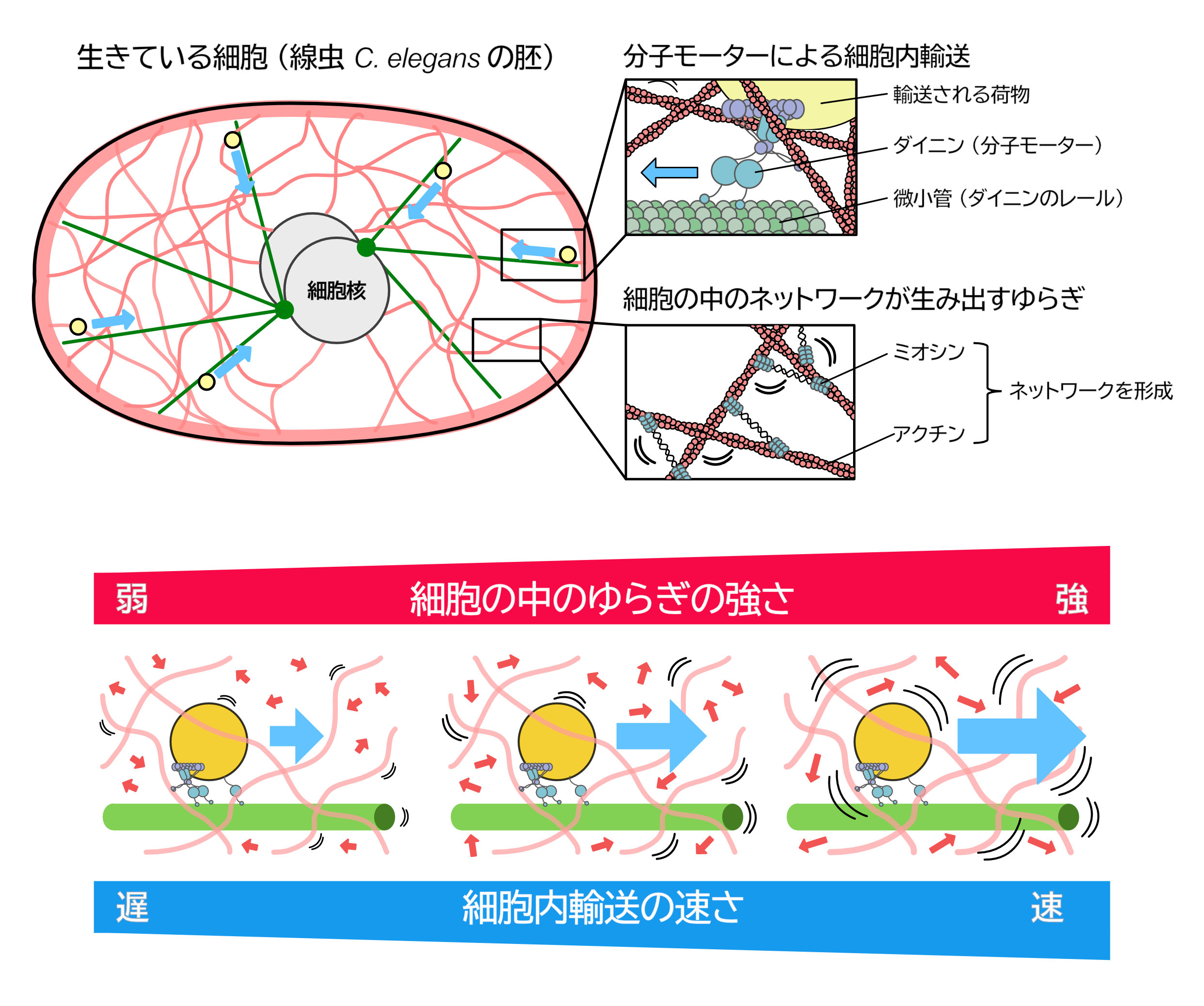
(上) 生きている細胞の中でおこる2つの動き。ダイニンという分子モーターが細胞の中で輸送をおこない、その周囲ではミオシンとアクチンという分子がネットワーク (アクトミオシンネットワーク) をつくり、ランダムな動きと力を生み出している。(下) アクトミオシンネットワークが生み出すランダムな動きが強いほどダイニンによる輸送は速くなる。
総研大生 Kim Jaehaさんが「Best Poster Award」を受賞

国立遺伝学研究所・森研究室・ゲノム多様性研究室のD4学生のKim Jaehaさんが、2025年11月14日(金)〜11月16日(日)に沼津市立図書館で開催された国際シンポジウム、2nd Asian Genetics Consortium Conference(AGCC 2025)にて、Best Poster Awardを受賞しました。
▶ 受賞ポスタータイトル:
Behavioral phase transitions in the migratory locust, Locusta migratoria, are related to changes in gut microbiome composition
国立遺伝学研究所 新分野創造センター 植物進化研究室 特任研究員(ポスドク)募集
植物進化研究室( https://kenji-fukushima-lab.github.io/ja/ )の一員として、国立遺伝学研究所の研究環境を存分に活かしながら研究活動に取り組んでいただく研究員を募集いたします。本研究室では、植物をおもな研究対象に、革新的な形質の進化機構解明を目指します。本公募の採用者が取り組む研究課題は、2026年度から開始予定の研究プロジェクト『「捕食」の起源の植物における再現と初期分子適応の解明』に基づき、本人の興味と経験を考慮してPIと相談の上で決定します。事前にお問い合わせいただければ、上記研究プロジェクトの詳細をご説明いたします。
パートナーでの応募を検討されている方は、国立遺伝学研究所博士研究員制度やその他の人材募集を併せてご参照ください。
(なお、2026年度国立遺伝学研究所博士研究員の募集は締め切りました。)
| 【募集人数】 | 1名 |
| 【募集期間】 | 2026年3月31日まで。応募のあったものから随時選考を行い、採用が決まり次第募集を締め切ります。選考・採用手続きには時間を要するため、4月1日の着任を希望される方は遅くとも1月末日までにご応募ください。 |
| 【着任時期】 | 2026年4月1日以降のなるべく早い時期を希望しますが、着任日については相談のうえ年度内の範囲で柔軟に対応します。 |
| 【勤務地】 | 静岡県三島市谷田1111(変更の予定なし) |
| 【配属部署】 | 新分野創造センター 植物進化研究室 |
| 【給与】 | 給与:年俸額410万円程度 情報・システム研究機構特定有期雇用職員就業規則に基づき支給します。 文部科学共済、雇用保険、労災保険、通勤手当(条件付き)が付帯します。 |
| 【勤務時間】 | 就業時間 : 08:30-17:15 休憩時間 : 12:00-13:00 休日 : 土・日曜日、祝日法に基づく休日、12月29日~1月3日は休日 夏季休業:8月12日~18日の土・日を除く連続する2日間(事業所ごとに指定) 時間外勤務 : 裁量労働制 |
| 【応募資格】 | 理系の博士号を取得した方、または着任までに取得見込みの方を募集します。 |
| 【業務における経験】 | 進化生物学・植物科学・分子生物学・生化学などの分野での研究経験がある方を特に歓迎します。植物を対象とした実験やバイオインフォマティクス解析の経験を有する方を歓迎しますが、必須要件ではありません。主体的に研究を遂行可能で、研究室の一員として協調性をもって職務に取り組んでいただける方を歓迎します。 |
| 【契約期間】 | 本雇用は年度契約とし、研究室PIの現在のテニュアトラック任期が終了する2029年3月31日まで更新可能です。ただし、テニュアトラック審査を通過するなどの理由により研究室PIの任期が延長された場合には、雇用財源となる研究費の助成終了時期(2031年3月31日)まで更新が可能となる場合があります。なお、本雇用契約締結直前に情報・システム研究機構との有期雇用契約を有している場合には、当該期間を通算して10年を超えて雇用することはできません。また、雇用契約の締結は、満65歳に達する日の属する事業年度を超えて行うことはできません。 |
| 【その他の待遇】 | 支給要件を満たした場合に通勤交通費の実費相当額を支給 文部科学省共済組合、雇用保険及び労災保険に加入 |
| 【就業場所における受動喫煙防止のための取組事項】 | 指定喫煙所を除き敷地内禁煙 |
| 【応募方法】 | (1)CV(履歴書):Eメールアドレスを明記し、日本語もしくは英語で自由書式にて作成してください。ただし、researchmap上の「基本情報」・「経歴」・「学歴」・「受賞」・「主要な論文」・「共同研究・競争的資金等の研究課題」の各項目が不足なく記述されている場合は、応募者のresearchmap URLをもって代替可能です。 (2)応募者について所見を求めることのできる方2名以上の氏名と連絡先 以上を一つのPDFファイルにまとめてEメール添付にて 国立遺伝学研究所植物進化研究室 福島 ( ) までお送りください。メール件名には「植物進化研究室 研究員応募」を明記してください。 |
| 【応募書類の返却】 | 応募書類はすべて当方にて責任をもって廃棄いたします。 |
| 【選考・結果通知】 | 応募を受け付けたのち、速やかに書類選考を開始します。書類選考通過者は、Zoomあるいは対面でPIと面接を行っていただきます。結果は、選考後速やかに本人あてにEメールにて通知します。 |
| 【備考】 | ※上記記載以外の待遇・勤務条件の詳細は、次の規則をご覧ください。 情報・システム研究機構特定有期雇用職員就業規則 ※情報・システム研究機構では、男女共同参画社会基本法(平成11年法律第78号)の趣旨に則って選考を行います。 ※情報・システム研究機構では、「障害者基本法(昭和45年法律第84号)」「障害者の雇用の促進等に関する法律(昭和35年法律第123号)」及び「障害を理由とする差別の解消の推進に関する法律(平成25年法律第65号)」の趣旨に則って選考を行います。 |
| 【連絡先】 | 本公募へのお問い合わせは、国立遺伝学研究所 植物進化研究室 福島 ( ) までお寄せください。 |
細胞のがん化によって変化するクロマチン動態
Aoi Otsuka, Masa A. Shimazoe, Shigeaki Watanabe, Katsuhiko Minami, Sachiko Tamura, Tohru Kiyono, Fumitaka Takeshita, and Kazuhiro Maeshima* (*Corresponding author)
Single-nucleosome imaging uncovers biphasic chromatin dynamics in inducible human transformed cells
Cell Structure and Function (2025) 先行オンライン公開 DOI:10.1247/csf.25147
真核細胞のゲノムDNAはヌクレオソームとして折りたたまれ、ユークロマチンとヘテロクロマチンを含む動的なドメインを形成します。がん細胞ではクロマチン状態や高次構造の変化が広く報告されていますが、がん化の進行に伴ってクロマチンの動態が時間的にどう変化するかは未解明でした。
ゲノムダイナミクス研究室の大塚碧 総研大生 (SOKENDAI特別研究員)、島添將誠 総研大生 (学振特別研究員DC1)、南克彦 特任研究員、田村佐知子 テクニカルスタッフ、前島一博 教授のグループは、国立がん研究センター 清野透 客員研究員 (公益財団法人 佐々木研究所 客員研究員併任)、渡邊重明 特任研究員、竹下文隆 部門長と協力し、ヒト上皮細胞に がん遺伝子として知られるヒトパピローマウイルス16 E6/E7、MYC、KRAS を導入し、ドキシサイクリンで誘導発現できる“EMR”細胞を樹立しました。このEMR細胞はがん遺伝子の誘導発現に伴い、がん様形質(増殖加速・接触阻害の回避・軟寒天内増殖・ヌードマウスでの腫瘍形成)を示します。生細胞1分子ヌクレオソームイメージングで、誘導後の日〜週スケールのクロマチン動態を追跡したところ、1–3日では変化がなく、5–7日に一過的に上昇し、4週には元のレベルへ戻る「二相性」の性質(図上段)が明らかになりました。また、動態上昇期にはヒストン H3/H4 アセチル化と転写が増加していました(図下段)。これらの結果から、がん遺伝子誘導が広範なアセチル化を伴う一時的なクロマチンの「ゆるみ」、転写活性化を引き起こし、クロマチンの再構築により動態が再安定化すると考えられます。
本研究は、がん化過程におけるクロマチンの物理的ふるまいが時間とともに再編成されることを1分子ヌクレオソームレベルで示し、クロマチン動態が細胞のがん化段階や適応状態を読み解く物理指標になり得ることを示しました。これらの知見はがん化に伴うクロマチン再編成の実体解明に役立つと考えられます。
本研究は、JSPSおよびMEXT KAKENHIの助成金(JP23K17398、JP24H00061、JP23KJ0998、JP24KJ1161)、JST のSPRING JPMJSP2104、および武田科学振興財団の助成を受けて実施されました。
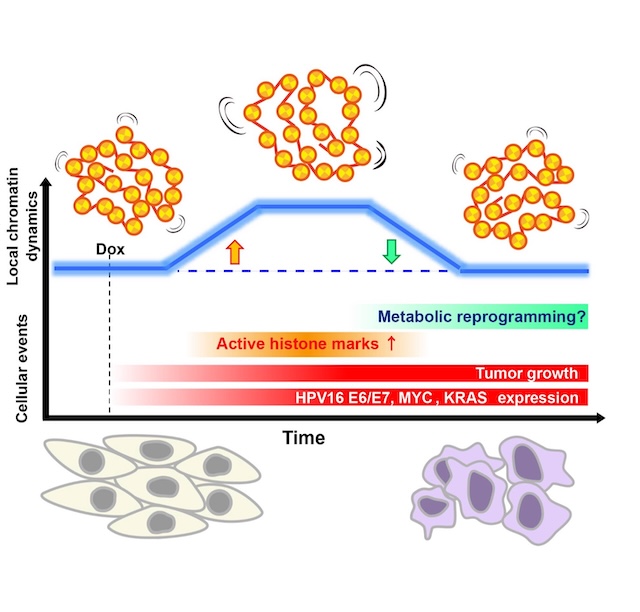
本研究結果のモデル図
EMR細胞のドキシサイクリン誘導によるがん化過程における局所クロマチン動態の二相性変化。ドキシサイクリン添加後、局所クロマチン動態(青の実線)が一過的に上昇(橙色矢印)し、その後、元のレベルへ戻る(緑色矢印)。上昇期には活性ヒストン修飾(H3/H4アセチル化など)と転写が増加する。時間経過とともに動態は再び安定化する一方、がん遺伝子の発現と腫瘍増殖は持続する。この過程には代謝リプログラミングが関与している可能性がある。
都市部の湖でイトヨ進化中──自然選択が生物の急速進化を駆動
Yamasaki, Y.Y., Yamaguchi, R., Nagano, A. J., Chen, B.J., Musto, N., Archambeault, S., Peichel, C.L., Schulien, J.A., Code, T.J., Beauchamp, D.A., and Kitano, J.
Inferring the strength of directional selection on armor plates in Lake Washington stickleback while accounting for migration and drift.
Evolution in press (2025) DOI:10.1093/evolut/qpaf254
生物の進化は、悠久の年月をかけて起こるものだけでなく、数年〜数十年という単位で観察できるものもあります。そのような急速進化を活用することで、自然選択(異なる遺伝型をもつ個体が異なる生存率を持つこと)がどのように生物集団で実際に作用しているのかを明らかにすることができます。
特に、近年の分子遺伝学の進展により、表現型に関わる原因遺伝子の特定が進み、これら遺伝子座における自然選択の強さを推定することが可能となりました。しかし、自然環境下では、異なる集団からの移住がある場合、選択圧を推定するのが困難になります。従って、これまでの選択圧を推定した研究は、閉鎖環境や実験環境下で推定されたものに限られていました。
このたび、生態遺伝学研究室の山﨑曜助教、北野潤教授、北海道大学の山口諒助教らの研究グループは、この課題を解決するべく、全ゲノムデータを用いることで移住率や有効集団サイズといった集団動態パラメータを推定し、このパラメータを予測モデルに組み込むことで自然選択圧を推定することに成功しました。
北米のシアトルの都市部にあるワシントン湖では、トゲウオ科のイトヨ(Gasterosteus aculeatus)において、1957年から2005年に、鱗板(骨化した外組織)が体の側面の全体をカバーする「完全型」と呼ばれる個体の出現頻度が上昇していました。これは、下水処理による湖水の透明度の上昇という人為的環境改変によって、視覚的に獲物を探索するマスによる捕食圧が増大したことに原因があると考えられていました。
本研究では、過去のデータを用いて、この鱗板を決定する遺伝子座に作用する選択圧を推定し、さらに、その予測モデルに基づいて現在の遺伝型頻度を予測し、実際に2022年に採集したサンプルを用いて予測を検証しました。
その結果、完全型は非完全型の個体に対して数パーセント生存率が高く、2005年以降も完全型の頻度は上昇し続けており、進化が継続していることが確認されました。加えて、2022年に観察された頻度は予測値を上回り、近年選択圧がさらに強まっている可能性が示されました。選択圧がさらに強まった要因の一つとして、例えば、夜間照明の上昇による捕食圧の上昇が考えられますが、現段階では証明できていません。
本研究は、分子遺伝学・集団ゲノミクス・シミュレーションを統合することで、移住や遺伝的浮動といった自然環境下で複雑に作用する因子も考慮に入れた上で選択圧を定量化できることを示したものであり、北米進化学会が刊行するEvolution誌に掲載されました。
本研究は科研費(22H04983, 20J01503, 21H02542, 22KK0105)、JST CREST(JPMJCR20S2)などの支援を得て、アメリカとスイスとの国際共同研究として実施しました。
北野教授のコメント
「20年ほど前にアメリカに留学していた時に、ワシントン湖のイトヨが急速進化していることを見出しました。このたび、2022年に再訪したところ、いまだにイトヨは進化中で、しかも、さらに選択圧が上昇していることにたいへん驚きました。ガラパゴスに行かなくても進化は観察できるのです。ワシントン湖は海や流入河川と繋がっており、周囲のイトヨ集団からの移住もあることから、正確な選択圧の推定が困難だったのですが、このたび、山﨑さんや山口さんの助けを借りて定量的に解析する方法論を確立できました。このように、急速進化の事例を詳細に調べることで、自然選択がどのように生物の進化を駆動するのかを実際に観察することが可能になります。」

図:ワシントン湖では、鱗板(骨化した外組織)が体の側面の全体をカバーする「完全型」と呼ばれる個体(上)の出現頻度が上昇している。
眼は脳に何を伝えるか── 網膜からの暗号情報を脳が解読する仕組みを解明
プレスリリース
Decoupling of visual feature selectivity in the retinocollicular pathway
Ole S. Schwartz, Akihiro Matsumoto, Haruka Yamamoto, and Keisuke Yonehara
Current Biology 2025 DOI: 10.1016/j.cub.2025.11.050
わたしたちが「ものが“見えた”」と感じるとき、中枢神経系は明るさや、色、動きなど膨大な視覚情報を処理しています。その起点となる網膜は、光を受容して情報を神経発火として暗号化し、脳へ送ります。しかし、脳がこの網膜暗号をどのようにして解読するのか不明でした。
本研究ではネズミを用い、生存に重要な「明るさの変化」と「動き」というの2種類の視覚情報に着目しました。相互情報量解析を応用し、網膜と視覚中枢である中脳上丘の神経活動を比較分析しました。その結果、網膜では、それらの情報が互いに予測可能な形で結びついていました。これは、異なる情報を一緒に “パッキング”することで脳への伝送が効率化されることを示しています。一方、中脳上丘では、その重複性が解消され、視覚特徴が再編成されていました。これは、中脳上丘がただの暗号の中継局ではなく、暗号解読の精度を高め、視覚応答の多様性を劇的に増やす役割を担うことを示します。
こうした処理は、注意、危険検知、素早い眼球運動など、中脳上丘が担う高度でしなやかな認知や行動に不可欠だと考えられます。本研究は、注意欠如や統合失調症など神経暗号の解読異常が関わると予想される疾患の理解にも寄与すると期待されます。
本成果は、オーフス大学生物医学部DANDRITE研究所のOle Schwartz大学院生、国立遺伝学研究所の松本彰弘助教、山本悠研究員、米原圭祐教授によるものです。
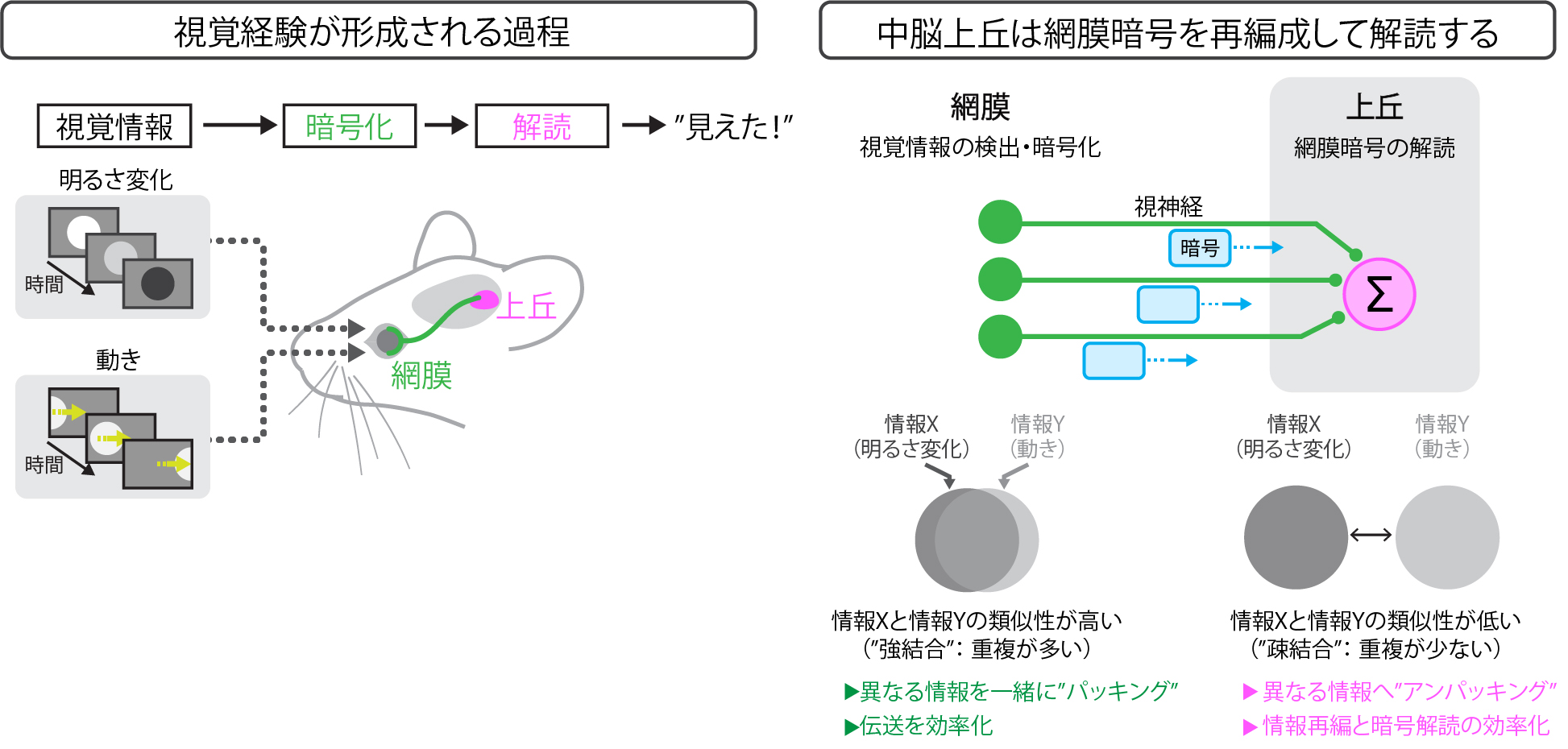
(左)視覚経験が形成される視覚情報処理経路。視覚情報は網膜において検出、暗号化され、上丘へ伝送される。(右)網膜では、「明るさ変化(情報X)」と「動き(情報Y)」の2種類の視覚情報を表現する神経活動の相互情報量が高い。上丘では、網膜暗号の重複性が解消される。
ゲノムダイナミクス研究室 技術補佐員募集
| 【職務内容】 | 技術補佐員(短時間雇用職員)として、研究室のメンテナンス業務を主に担当していただきます。勤務時間に応じて分子生物学実験補助をお願いする場合もあります。未経験の業務を担当する場合でも、適切な指導を行います。 |
| 【募集人数】 | 1名 |
| 【応募資格】 | 以下の条件を満たす方を募集しています。 1. 理系の専門学校、高専、大学を卒業している、着任までに卒業見込みであること、もしくは関連する職務経験を有すること。関連する職務経験とは、企業、大学、研究所での研究補助業務などを含みます。 2. マイクロソフトエクセル・ワードの取り扱いを含むパソコンの基本操作ができること。 3. 研究室の一員として協調性をもって職務に取り組む意欲があること。 |
| 【採用時期】 | 採用決定以降のできるだけ早い時期(応相談) |
| 【勤務地】 | 静岡県三島市谷田1111 国立遺伝学研究所(変更の予定なし) |
| 【勤務時間】 | 勤務は週3-5日、9:00~16:00(うち昼休憩1時間)。週12時間から30時間の間で、相談のうえ決定します。土・日曜日、祝日法による休日、12月29日から翌年1月3日、その他特に指定する日が休日となります。勤務条件に応じた日数の年次有給休暇が付与されます。詳細については、「情報・システム研究機構 短時間雇用職員就業規則」をご参照ください。ご家族の急な病気や学校行事にも相談に応じます。 |
| 【給与・待遇】 | 【給与・待遇】研究所の給与規定に準じて、1,371円(大卒新卒)~1,513円を支給します。勤務条件に応じて、健康保険、年金、雇用保険、労災保険に加入します。定期健康診断あり。支給要件を満たした場合に通勤手当等を支給します。車通勤も可能です。勤務条件の詳細については、「情報・システム研究機構 短時間雇用職員就業規則」をご参照ください。 |
| 【契約期間・更新】 | 雇い入れ時の契約期間は2026年3月31日までとなり、以降は事業年度(4月1日から翌年の3月31日まで)ごとに雇用契約を更新する可能性があります。更新の可否は、雇用中の勤務実績等を考慮の上判断します。詳細については、「情報・システム研究機構 短時間雇用職員就業規則」をご参照ください。 |
| 【応募方法】 | 履歴書のPDFファイルを添付の上、ゲノムダイナミクス研究室(055)981-6864 ・学歴 ・職歴(担った役割や業務内容) ・Eメールアドレス ・電話番号 ・その他、選考で参考となる情報 |
| 【応募締切】 | 応募のあったものから随時選考を行い、採用が決まり次第募集を締め切ります。 |
| 【備考】 | 応募日から一ヶ月以内に、書類選考の可否をEメールにて通知します。書類選考を通過された方のみ、対面による面接を実施します。応募書類は「情報・システム研究機構 個人情報保護規程」 に則り厳重に管理し、採用審査の用途に限り使用します。これらの個人情報は、正当な理由なく第三者に開示、譲渡及び貸与することはありません。 |
ナウマンゾウの古代 DNA 解析に成功 〜ユーラシア最古のパレオロクソドンの系統であることが判明〜

●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●
●●●●●● ●●●●●●, ●●●●●● ●●●●●●, ●●●●●● ●●●●●●, ●●●●●● ●●●●●●
●●●●●● ●●●●●● (20●●) ●●, e●●●●●●●●●●●● DOI:●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●

図:画像の説明です。画像の説明です。画像の説明です。画像の説明です。画像の説明です。画像の説明です。
画像の説明です。画像の説明です。画像の説明です。
動画の説明です。動画の説明です。動画の説明です。動画の説明です。動画の説明です。動画の説明です。
動画の説明です。動画の説明です。
対⽴が共存を⽣むアリの群集⽣態学 〜同じ環境に多数の種が共存するアリの謎を実験的に解明〜

●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●
●●●●●● ●●●●●●, ●●●●●● ●●●●●●, ●●●●●● ●●●●●●, ●●●●●● ●●●●●●
●●●●●● ●●●●●● (20●●) ●●, e●●●●●●●●●●●● DOI:●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●

図:画像の説明です。画像の説明です。画像の説明です。画像の説明です。画像の説明です。画像の説明です。
画像の説明です。画像の説明です。画像の説明です。
動画の説明です。動画の説明です。動画の説明です。動画の説明です。動画の説明です。動画の説明です。
動画の説明です。動画の説明です。
細菌共通の「合成困難なアミノ酸配列」確認──逆に利用するタンパク質群も発見
プレスリリース
Evolutionary Adaptation of Bacterial proteomes to Translation-Impeding Sequences
藤原圭吾、辻奈緒子、崎山歌恋、仁木宏典、千葉志信(Keigo Fujiwara, Naoko Tsuji, Karen Sakiyama, Hironori Niki, and Shinobu Chiba)
The EMBO Journal (2025)
細胞は、リボソーム上でアミノ酸をつなぎ合わせてタンパク質を合成します。細菌は短時間で多くのタンパク質を合成できるため、効率的に生育することが可能です。一方で、合成途中に現れる特定のアミノ酸配列が、リボソームを一時的に停止させる「難翻訳配列」として働く場合があり、大腸菌や枯草菌などの一部の細菌では、特殊な遺伝子発現制御系の中でいくつかの例が報告されています。
本研究では、ゲノム上に存在するタンパク質配列の中で、特定のアミノ酸配列の出現頻度を計算する独自のアルゴリズムを開発し、5万種を超える細菌を対象とした網羅的なゲノム解析を行い、難翻訳配列の探索を行いました。
その結果、地球上に生息する多様な細菌は共通して合成が困難なタンパク質のアミノ酸配列(難翻訳配列)を持つことが明らかになりました。さらに、難翻訳配列は、細菌のタンパク質内部にはほとんど見られないことが明らかになりました。これは、細菌が進化の過程で難翻訳配列の使用を回避する方向に適応してきたことを示唆しています。
一方で、比較的小さなタンパク質の末端(カルボキシ末端)付近に、難翻訳配列がしばしば存在することが分かりました。生物情報学的解析により、こうしたタンパク質群が、外部環境の変化に対して細菌が適応する上で、重要かつ多様な機能を果たしている可能性が示唆されました。
このように、一見すると細胞にとって不都合に思える難翻訳配列ですが、一般的にはタンパク質から進化的に排除される一方で、細菌はその特性を逆手に取り、特定の遺伝子発現の調節システムとして利用することで、環境変化へ適応するために役立てていることが明らかになりました。
本研究は、国立遺伝学研究所の藤原圭吾特命助教(JSTさきがけ研究員)がこの難翻訳配列の探索アルゴリズムを開発し、京産大チームと協力して研究を進めました。
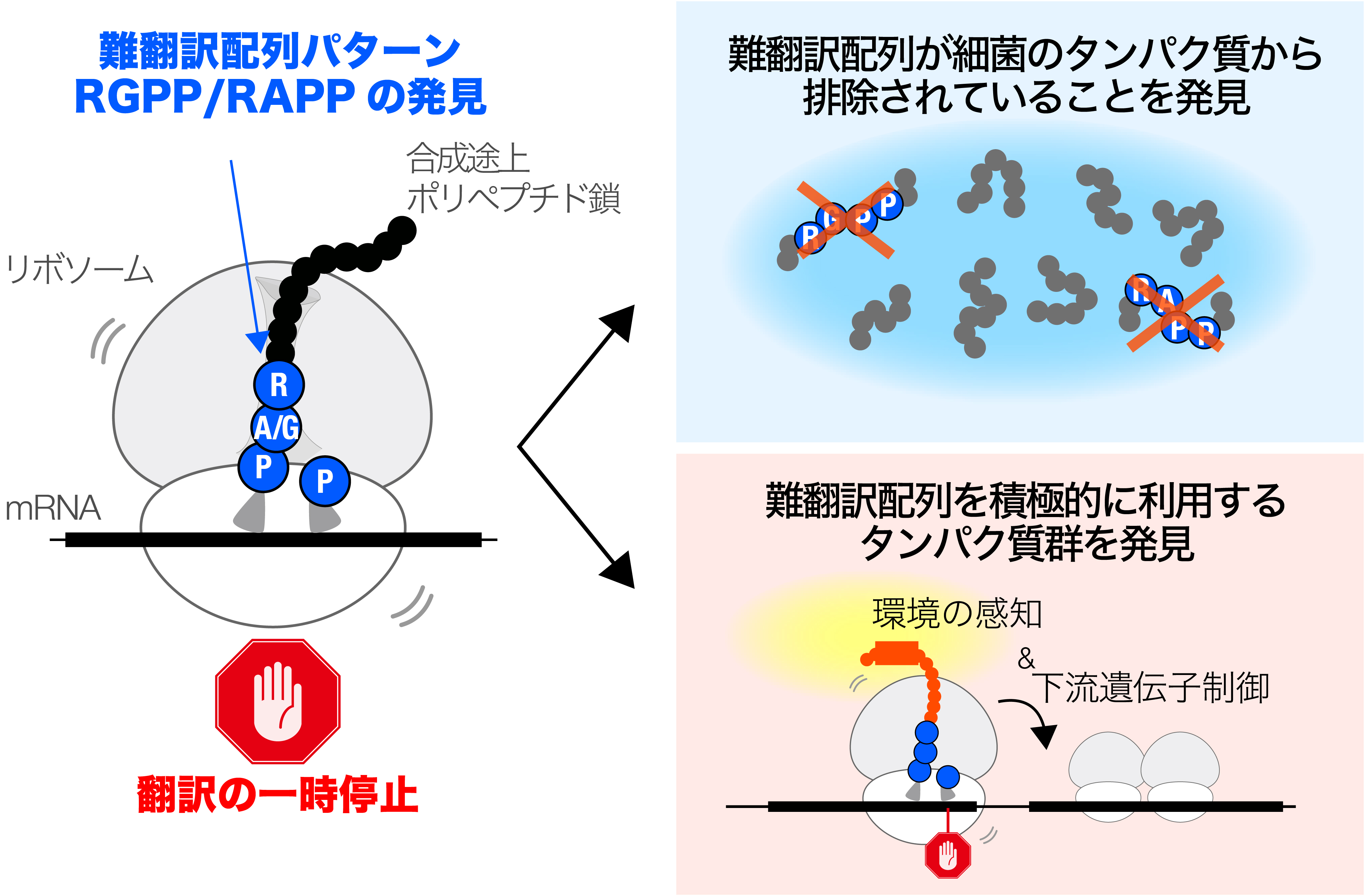
図:多くの細菌において「タンパク質の合成を止めてしまうアミノ酸配列」のパターンを発見(左)。そのような配列パターンは、一般的には、進化の過程で排除されるが(右上)、一方で、細菌は、合成困難性を細胞の機能維持に役立てるユニークなしくみを進化させることもある。
サクラ野生2種、高精度ゲノム公開──進化の「設計図」を解読
Chromosome-scale genomes of two wild flowering cherries (Cerasus itosakura and C. jamasakura) provide insights into structural evolution in Cerasus
Kazumichi Fujiwara, Atsushi Toyoda, Toshio Katsuki, Yutaka Sato, Bhim B Biswa, Takushi Kishida, Momi Tsuruta, Yasukazu Nakamura, Takako Mochizuki, Noriko Kimura, Shoko Kawamoto, Tazro Ohta, Ken-Ichi Nonomura, Hironori Niki, Hiroyuki Yano, Kinji Umehara, Chikahiko Suzuki, Tsuyoshi Koide
DNA Research(2025) , DOI:10.1093/dnares/dsaf031
日本の自然景観と文化を象徴するサクラ。その多様性や進化の歴史を理解するうえで基盤となるのが、野生種の高精度なゲノム情報です。しかし、日本に自生する主要な野生サクラの染色体レベルの網羅的なゲノムはこれまで存在せず、種間関係の精密な比較や園芸品種の起源解明には制約がありました。今回、国立遺伝学研究所と森林総合研究所を中心とする「サクラ100ゲノムコンソーシアム」は、日本を代表する野生サクラであるエドヒガン(Cerasus itosakura)とヤマザクラ(C. jamasakura)について、初の染色体スケールの高品質ゲノムを完成させました。
得られた2種のゲノムは、どちらも極めて高い精度と完全性を備え、サクラ属の染色体構造を詳細に再現しています。比較解析の結果、両種は全体として強い遺伝子配列の保存性を示す一方で、重要な種間差として、エドヒガンの第8染色体に約1.84 Mb の大型逆位構造が存在することが明らかになりました。このような大規模な構造変化は進化の重要な手がかりであり、エドヒガン系統に固有の歴史的な染色体再編成を反映している可能性があります。また、rRNA 遺伝子クラスターの位置やコピー数の違いなど、染色体上の特定領域における種間差も確認され、サクラ属における染色体進化やゲノム多様性の形成過程について新たな視点を提供しました。
さらに、この新規データを用いて、日本を代表する園芸品種「ソメイヨシノ」のハプロタイプを再構築したところ、それぞれがエドヒガン系統とオオシマザクラ(C. speciosa)系統と高い類似性を示し、長年支持されてきた雑種起源説を染色体レベルで裏付ける結果が得られました。特に、従来のアセンブリでは不十分だった領域も高精度に比較できたことで、ソメイヨシノの遺伝的背景をより精密に描き出すことが可能となりました。
本研究は、サクラ属の進化、分類、形質多様性の理解を大きく前進させると同時に、園芸品種の育種研究や保全戦略の立案においても重要な基盤となるものです。日本の自然と文化の象徴であるサクラを科学的に支える、新たな大きな一歩となりました。
本プロジェクトには、遺伝研の藤原一道 特任研究員、豊田敦 特任教授、川本祥子 准教授、佐藤豊 教授、小出剛 准教授に加え、森林総研の勝木俊雄 博士、鶴田燃海 博士ら、多様な専門領域を持つ研究者が結集し研究を推進しました。

A、B: エドヒガンの花と樹木。C、D:ヤマザクラの花と樹木。E: エドヒガンとヤマザクラの染色体レベルゲノム構造。F: エドヒガンの8番染色体にみられる大型の逆位構造。この逆位は他のオオシマザクラ、ヤマザクラ、カンヒザクラにはみられない。
本研究は情報・システム研究機構戦略的研究プロジェクトの支援を受けて実施されました。
タンパク質合成失敗への対処機構を発見 神経変性疾患など、さまざまな疾患の発症理解へ

●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●
●●●●●● ●●●●●●, ●●●●●● ●●●●●●, ●●●●●● ●●●●●●, ●●●●●● ●●●●●●
●●●●●● ●●●●●● (20●●) ●●, e●●●●●●●●●●●● DOI:●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●
●●●●●● ●●●●●●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●●●●●●● ●●●●●●● ●●●●●●

図:画像の説明です。画像の説明です。画像の説明です。画像の説明です。画像の説明です。画像の説明です。
画像の説明です。画像の説明です。画像の説明です。
動画の説明です。動画の説明です。動画の説明です。動画の説明です。動画の説明です。動画の説明です。
動画の説明です。動画の説明です。
大学院見学会を2月18日(水)に開催(申込〆切:2月17日)
ヒト細胞ゲノム、DNA複製開始位置とタイミング解明──新技術を開発
プレスリリース
Regulated TRESLIN-MTBP loading governs initiation zones and replication timing in human DNA replication
Xiaoxuan Zhu, Atabek Bektash, Yuki Hatoyama, Sachiko Muramatsu, Shin-Ya Isobe, Chikashi Obuse, Atsushi Toyoda, Yasukazu Daigaku, Chun-Long Chen and Masato T. Kanemaki
Nature Communications 2025 DOI: 10.1038/s41467-025-66278-7
細胞が増えるとき、ゲノムDNAは正確に二倍に複製されます。このDNA複製の異常は、ゲノムDNAの変化を引き起こし、細胞老化やがん、遺伝性疾患などに関与します。そのため、「細胞がどのようにDNAを複製しているのか」を理解することは、生命現象や疾患、さらには進化を理解するうえで極めて重要です。
これまでDNA複製の研究は、大腸菌や酵母などの微生物を中心に進められてきました。これらの生物では、DNA複製が始まる場所(開始領域)はDNA配列によって決まっています。しかしヒトを含む多くの真核細胞では、どのDNA配列から複製が始まるのかがDNA配列によって決まっておらず、ヒトゲノムのどこで複製が開始し、どうやってその位置が選ばれるのかは長年の謎でした。
そこで、国立遺伝学研究所の筆頭著者・朱考軒(Xiaoxuan Zhu)博士研究員を含む鐘巻将人教授らの研究チームは、ヒト細胞ゲノム中のDNA複製開始領域を高精度に検出する新しい技術「LD-OK-seq法」を開発しました。さらに、この領域に結合するタンパク質を解析することで、ヒト細胞がどのように複製開始位置を決めているのか、その基本原理を明らかにしました。
その結果、ヒト細胞は転写している遺伝子領域を除けば、ほぼどこからでもDNA複製を開始できる能力を持つことがわかりました。この能力は、DNA複製に必要なMCMヘリカーゼという酵素がゲノム全体に広く結合していることに由来します。一方で、S期の初期には、転写している遺伝子の間の領域(遺伝子間領域)で複製が頻繁に開始されており、その場所はTRESLIN-MTBPというMCMヘリカーゼを活性化するタンパク質の結合によって決定されることがわかりました。さらに、TRESLIN-MTBPのMCMへの結合を調節する拮抗的な制御システムも発見しました。
これらの成果は、「ヒト細胞がどのようにゲノムDNAの複製を開始するのか」という根本的な疑問に答えるものであり、DNA複製異常によって起こるゲノム不安定性疾患(細胞死、がん、老化、遺伝疾患など)や、ゲノム変化を通じた進化の理解に新たな視点を与えます。また、将来的には、人工的にDNA複製を制御する新しい技術開発の基盤となることも期待されます。
本研究は国立遺伝学研究所の鐘巻将人教授の研究グループ、同研究所の豊田敦特任教授、大阪大学の小布施力史教授、公益財団法人がん研究所の大学保一グループリーダー、仏キュリー研究所のChun-Long Chen教授による国際共同研究によりおこなわれました。研究遂行にあたり、科研費(JP23H02463、JP21H04719, JP23H04925, JP25H00979)、先進ゲノム支援(JP22H04925 (PAGS))、JST FOREST(JPMJFR204X)、JST CREST(JPMJCR21E6)、AMED ASPIRE(JP25jf0126015)による支援を受けました。

図: ゲノムDNAにはMCMヘリカーゼが転写している遺伝子以外の領域に広く結合しており、MCMヘリカーゼのリン酸化はリン酸化酵素DDKと脱リン酸化酵素RIF1-PP1により拮抗的に制御されている。リン酸化されたMCMヘリカーゼにTRESLIN-MTBP複合体が呼び込まれることで、複製開始する場所が決定される。

















