Archive
- Home
- 2026/2
- 2026/1
- 2025/12
- 2025/11
- 2025/10
- 2025/9
- 2025/8
- 2025/7
- 2025/6
- 2025/5
- 2025/4
- 2025/3
- 2025/2
- 2025/1
- 2024/12
- 2024/11
- 2024/10
- 2024/9
- 2024/8
- 2024/7
- 2024/6
- 2024/5
- 2024/4
- 2024/3
- 2024/2
- 2024/1
- 2023/12
- 2023/11
- 2023/10
- 2023/9
- 2023/8
- 2023/7
- 2023/6
- 2023/5
- 2023/4
- 2023/3
- 2023/2
- 2023/1
- 2022/12
- 2022/11
- 2022/10
- 2022/9
- 2022/8
- 2022/7
- 2022/6
- 2022/5
- 2022/4
- 2022/3
- 2022/2
- 2022/1
- 2021/12
- 2021/11
- 2021/10
- 2021/9
- 2021/8
- 2021/7
- 2021/6
- 2021/5
- 2021/4
- 2021/3
- 2021/2
- 2021/1
- 2020/12
- 2020/11
- 2020/10
- 2020/9
- 2020/8
- 2020/7
- 2020/6
- 2020/5
- 2020/4
- 2020/3
- 2020/2
- 2020/1
- 2019/12
- 2019/11
- 2019/10
- 2019/9
- 2019/8
- 2019/7
- 2019/6
- 2019/5
- 2019/4
- 2019/3
- 2019/2
- 2019/1
- 2018/12
- 2018/11
- 2018/10
- 2018/9
- 2018/8
- 2018/7
- 2018/6
- 2018/5
- 2018/4
- 2018/3
- 2018/2
- 2018/1
- 2017/12
- 2017/11
- 2017/10
- 2017/9
- 2017/8
- 2017/7
- 2017/6
- 2017/5
- 2017/4
- 2017/3
- 2017/2
- 2017/1
- 2016/12
- 2016/11
- 2016/10
- 2016/9
- 2016/8
- 2016/7
- 2016/6
- 2016/5
- 2016/4
- 2016/3
- 2016/2
- 2016/1
- 2015/12
- 2015/11
- 2015/10
- 2015/9
- 2015/8
- 2015/7
- 2015/6
- 2015/5
- 2015/4
- 2015/3
- 2015/2
- 2015/1
- 2014/12
- 2014/11
- 2014/10
- 2014/9
- 2014/8
- 2014/7
- 2014/6
- 2014/5
- 2014/4
- 2014/3
- 2014/1
- 2013/12
- 2013/11
- 2013/10
- 2013/9
- 2013/8
- 2013/7
- 2013/6
- 2013/5
- 2013/4
- 2013/3
- 2013/2
- 2013/1
- 2012/12
- 2012/11
- 2012/10
- 2012/9
- 2012/8
- 2012/7
- 2012/6
- 2012/5
- 2012/4
- 2012/3
- 2012/2
- 2012/1
- 2011/12
- 2011/11
- 2011/10
- 2011/9
- 2011/8
- 2011/7
- 2011/6
- 2011/5
- 2011/4
- 2011/3
- 2011/2
- 2011/1
- 2010/12
- 2010/11
- 2010/10
- 2010/9
- 2010/8
- 2010/7
- 2010/6
- 2010/5
- 2010/4
- 2010/3
- 2010/2
- 2009/12
- 2009/11
- 2009/10
- 2009/9
- 2009/8
- 2009/7
- 2009/6
- 2009/5
- 2009/4
- 2009/3
- 2009/2
- 2009/1
- 2008/12
- 2008/11
- 2008/10
- 2008/9
- 2008/7
- 2008/6
- 2008/5
- 2008/4
- 2008/3
- 2008/2
- 2008/1
- 2007/12
- 2007/11
- 2007/10
- 2007/9
- 2007/8
- 2007/7
- 2007/6
- 2007/3
- 2007/2
- 2007/1
- 2006/12
- 2006/11
- 2006/10
- 2006/9
- 2006/7
- 2006/6
- 2006/5
- 2006/4
- 2006/1
- 2005/6
- 2005/5
- 2005/4
- 2005/2
- 2004/12
- 2004/11
- 2004/10
- 2004/8
- 2004/7
- 2004/6
- 2004/5
- 2004/2
- 2004/1
- 2003/12
- 2003/8
- 2003/7
- 2003/6
- 2003/5
- 2003/3
- 2003/1
- 2002/12
- 2002/10
- 2002/5
- 2002/4
- 2002/2
- 2002/1
- 2001/8
- 2001/5
- 2001/4
- 2001/2
- 2000/8
- 2000/7
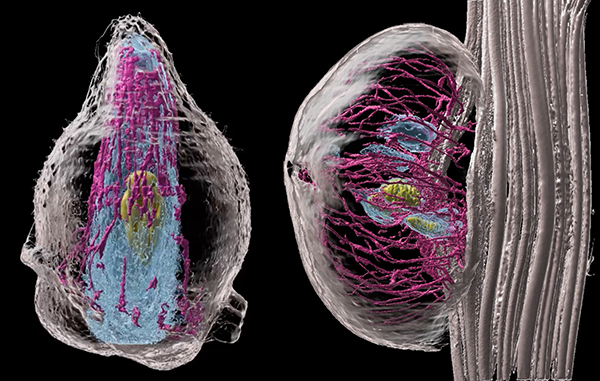
なぜこれだけ多くの仕組みが必要なのか?
〜ゲノム刷り込みの多層的な制御機構の解明〜
プレスリリース
Multi-layered epigenetic control of persistent and stage-specific imprinted genes in rice endosperm
Kaoru Tonosaki, Daichi Susaki, Hatsune Morinaka, Akemi Ono, Hiroki Nagata, Hiroyasu Furuumi, Ken-Ichi Nonomura, Yutaka Sato, Keiko Sugimoto, Luca Comai, Katsunori Hatakeyama, Taiji Kawakatsu, Tetsu Kinoshita
Nature Plants 2024 July 30 DOI:10.1038/s41477-024-01754-4
横浜市立大学木原生物学研究所の殿崎薫助教と木下哲教授、農研機構の川勝泰二上級研究員、国立遺伝学研究所、理化学研究所、アメリカ・カリフォルニア大学デイビス校、岩手大学との国際共同研究グループは、イネの胚乳発生段階や細胞の種類によって異なるゲノム刷り込み(ゲノムインプリンティング)の仕組みが存在することを明らかにしました。
本研究では、イネの胚乳における時系列マルチオミクス解析から、発生の進行過程で多数の刷り込み遺伝子(インプリント遺伝子)を同定し、全てのステージに持続的なインプリント遺伝子と、発生ステージ特異的なインプリント遺伝子が存在することを突き止めました(図)。さらにシングルセル解析から、細胞の種類によってもインプリント遺伝子の制御が異なることを示唆する結果を得ることに成功しました。極めて複雑に制御されるゲノムインプリンティングの制御メカニズムの全容解明や、植物の種子形成過程におけるインプリント遺伝子の機能解明に関する研究への進展が期待されます。
本研究は、科研費補助金学術変革領域(A) 「挑戦的両性花原理」、若手研究、横浜市立大学学長裁量事業 第5期戦略的研究推進事業「研究開発プロジェクト」などの支援を受けて実施されました。
本研究成果は、「Nature Plants」誌に掲載されます(英国夏時間2024年7月30日10時)。
図: 胚乳発生過程で異なる発現様式を示すインプリント遺伝子とその制御機構。
持続的インプリント遺伝子とステージ特異的インプリント遺伝子ではエピゲノム状態が異なる。
遺伝研オンライン大学院説明会
イネの減数分裂移行タイミングの制御に必要な細胞質RNA顆粒
Impact of protein domains on the MEL2 granule, a cytoplasmic ribonucleoprotein complex maintaining faithful meiosis progression in rice.
Manaki Mimura, Seijiro Ono, Harsha Somashekar, Ken-Ichi Nonomura
New Phytologist 2024 Jul 24. DOI:10.1111/nph.19968
RNAとタンパク質で構成されるRNA顆粒は、ストレス刺激などに応答してダイナミックに離合集散する細胞内構造体として知られています。代表的なRNA顆粒のひとつとして、動物の生殖細胞運命を決定する生殖顆粒の研究が進んでいます。しかし、植物の生殖細胞発生に寄与するRNA顆粒の報告はほとんどありませんでした。
植物細胞遺伝研究室の三村真生研究員(現在は東京大学大学院助教)と野々村賢一准教授らの研究グループは、減数分裂への適切な移行タイミングを制御するイネRNA結合タンパク質MEL2が、(1) 減数分裂移行前の生殖母細胞で細胞質RNA顆粒(MEL2顆粒)を形成し、ストレス環境下でRNA代謝を司るストレス顆粒やmRNA分解・翻訳制御に関わるPボディ顆粒と共局在すること、(2) その後MEL2顆粒が適切に消去されることが減数分裂の正常な進行に不可欠なこと、(3) 顆粒の形成・消失に重要なMEL2機能ドメインを明らかにしました(図)。
イネMEL2と類似の構造をもつタンパク質(MEL2L)は多くの陸上植物で保存されており、ゼニゴケやヒメツリガネゴケのMEL2Lは減数分裂を行う胞子体で特異的に発現していました。陸上植物の祖先といわれる車軸藻類でも保存されていたことから、MEL2が陸上植物の生殖進化に重要な役割を果たした可能性が推察されました。
本研究は、学術振興会科研費、遺伝研共同研究事業(NIG-JOINT)、遺伝研博士研究員制度、学術振興会特別研究員制度(PD)の支援を受けて実施しました。
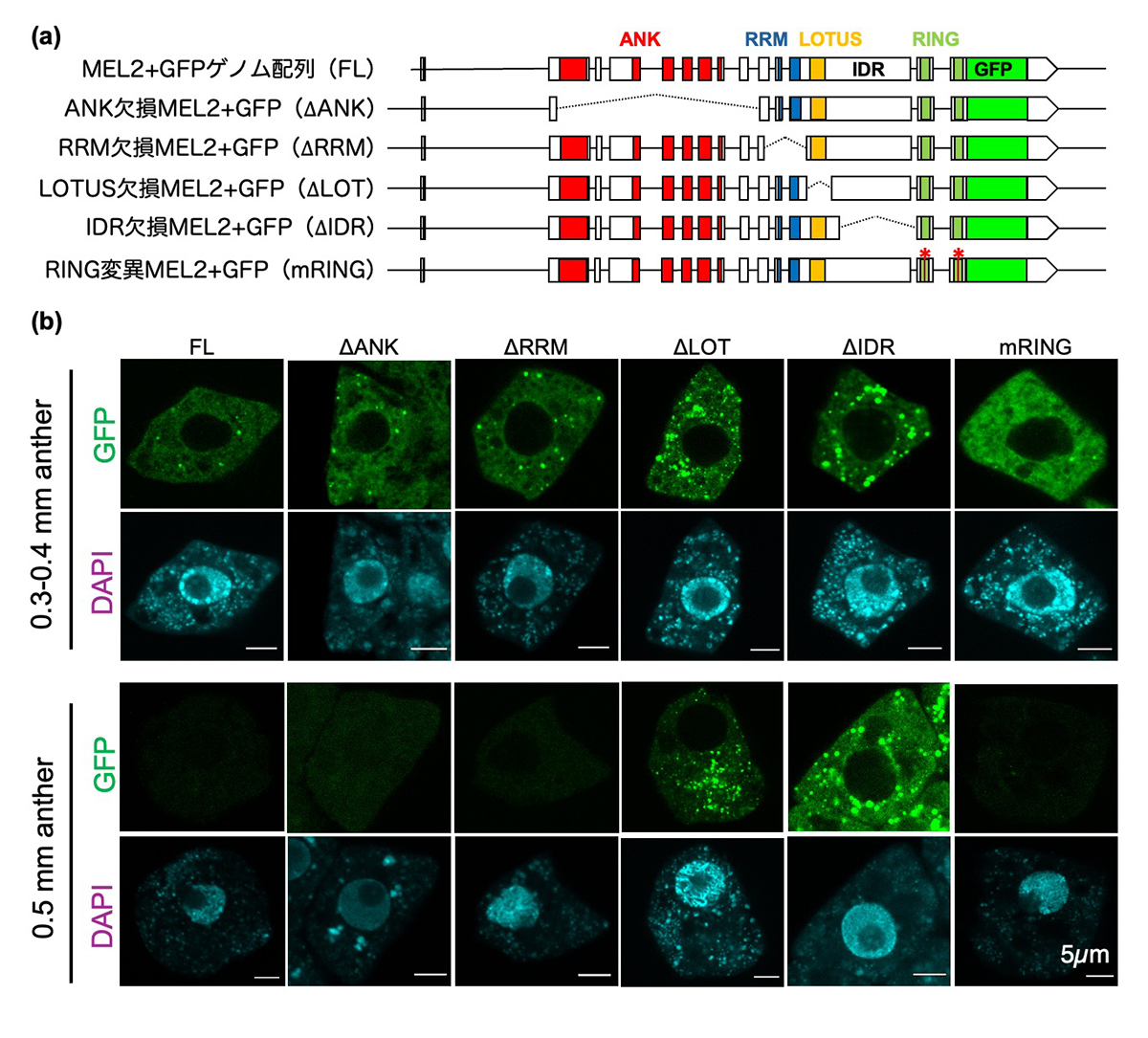
図:MEL2機能ドメインの顆粒形成における役割
(a) MEL2ゲノム配列と蛍光タンパク質GFPの融合遺伝子(FL)、および各機能ドメインに欠失(点線)・塩基置換(星印)が入った変異型MEL2融合遺伝子の構造。
(b) (a) を導入した形質転換イネの生殖母細胞質でみられるMEL2顆粒(緑)。減数分裂前(0.3-0.4mm葯)に形成される正常型MEL2顆粒は、減数分裂期(0.5mm葯)に入ると消失するが(FL)、mRING型MEL2は顆粒を形成できず、∆LOTUSおよび∆IDRは顆粒が減数分裂期まで持ち越され、減数分裂進行や花粉形成に異常をきたす。
光合成生物で初のタンパク質ノックダウン法
Development of a rapamycin-inducible protein-knockdown system in the unicellular red alga Cyanidioschyzon merolae.
Takayuki Fujiwara, Shunsuke Hirooka, Shota Yamashita, Fumi Yagisawa and Shin-ya Miyagishima
Plant Physiology (2024) kiae316 DOI:10.1093/plphys/kiae316
単細胞藻類の多くは、その細胞構造やゲノム構成が単純であるため、光合成生物に共通の現象やそのメカニズムを解明するのに適しています。しかしながら、遺伝子改変技術などの実験技法の開発が遅れているという問題があります。
光合成真核生物の進化の初期に分岐したグループである、単細胞紅藻イデユコゴメ類(綱)(Cyanidioschyzon merolae、CyanidiumおよびGaldieriaなど)は、光合成真核生物の中で遺伝子数が最小クラスであり(12-18 Mb、約4,700-8,000遺伝子)、細胞内構造も単純であるため、様々なオミクス解析を効率よく行うことができます。我々はこれまでに、本藻類の培養技術、相同組換えによる遺伝子破壊・導入技術、導入遺伝子の発現誘導系などを開発し、現在、世界各国の研究者等が、光合成、代謝、細胞周期、エピジェネティクス、生活環、タンパク質の構造、進化、産業利用など多様な分野の研究で、本藻類を用い始めています。
一方、イデユコゴメ綱の藻類は、RNA干渉機構(RNAi)を担う遺伝子群を失っているため、その機構を利用した遺伝子ノックダウンの実験を行えず、増殖や生存に必須な遺伝子の機能解析が困難でした。
これまで酵母や動物細胞を用いた研究では、RNA干渉に代わる手法として、目的遺伝子を狙ったタイミングで分解させる、誘導型タンパク質ノックダウン法が開発されてきました。その代表例である、オーキシンデグロン法(鐘巻研により開発)では、植物ホルモンであるオーキシンを細胞に添加することにより、標的タンパク質のユビキチン化が誘導され、その結果、プロテアソームによる迅速な分解が引き起こされます。本手法は、新規のタンパク質合成を阻害するRNAiとは異なり、すでに合成済みで細胞内で機能している標的タンパク質を直接分解できるという利点を有しています、しかしながら、植物ホルモンであるオーキシンは植物の成長や形態形成に影響を与え、またイデユコゴメ類に対して毒性があるため、本手法は適用できませんでした。
そこで我々は、イデユコゴメ類のC. merolae(図A)において、新たに、ラパマイシン誘導タンパク質ノックダウン法を開発しました。本手法は、化学物質ラパマイシンが、FRBタグ(ペプチド)とFKBPタンパク質を結合させる性質を利用しています(図B)。標的タンパク質にFRBを、ユビキチンリガーゼのサブユニット(CUL1)にFKBPを融合した細胞を用意し、これにラパマイシンを与えると、標的タンパク質とユビキチンリガーゼ複合体が結合し、標的タンパク質のユビキチン化とその後の分解が起こります(図B)。本手法を用いると、例えば試験用の標的タンパク質(蛍光タンパク質mVenus-FRB)は、ラパマイシン添加後、2時間以内にほとんど分解されます(図C)。また、論文中では、内在の必須遺伝子であるDRP5B(葉緑体分裂タンパク質)や、核内の細胞周期制御因子E2Fを迅速に分解できることを示しています。
本論文は、光合成生物において、タンパク質二量体誘導化合物を用いたタンパク質ノックダウン法を開発した初めての例となります。ラパマイシンはTORキナーゼの阻害剤であり、一般的には細胞に対して毒性がありますが、イデユコゴメ綱の生物に対しては毒性がありません。また、他生物においても、ラパマイシン類似体を用いることで、無毒性の誘導型タンパク質分解系を構築することができます。特にオーキシンに応答する陸上植物に対しては有用な方法になり得ます。本方法は藻類や陸上植物を含む光合成真核生物における精緻な実験系の構築を可能にし、様々な生物現象の解明に貢献します。
本研究は、科学研究費補助金(22K06396、20H00477)、JST未来社会創造事業(JPMJMI22E1)などの助成のもとに実施されました。
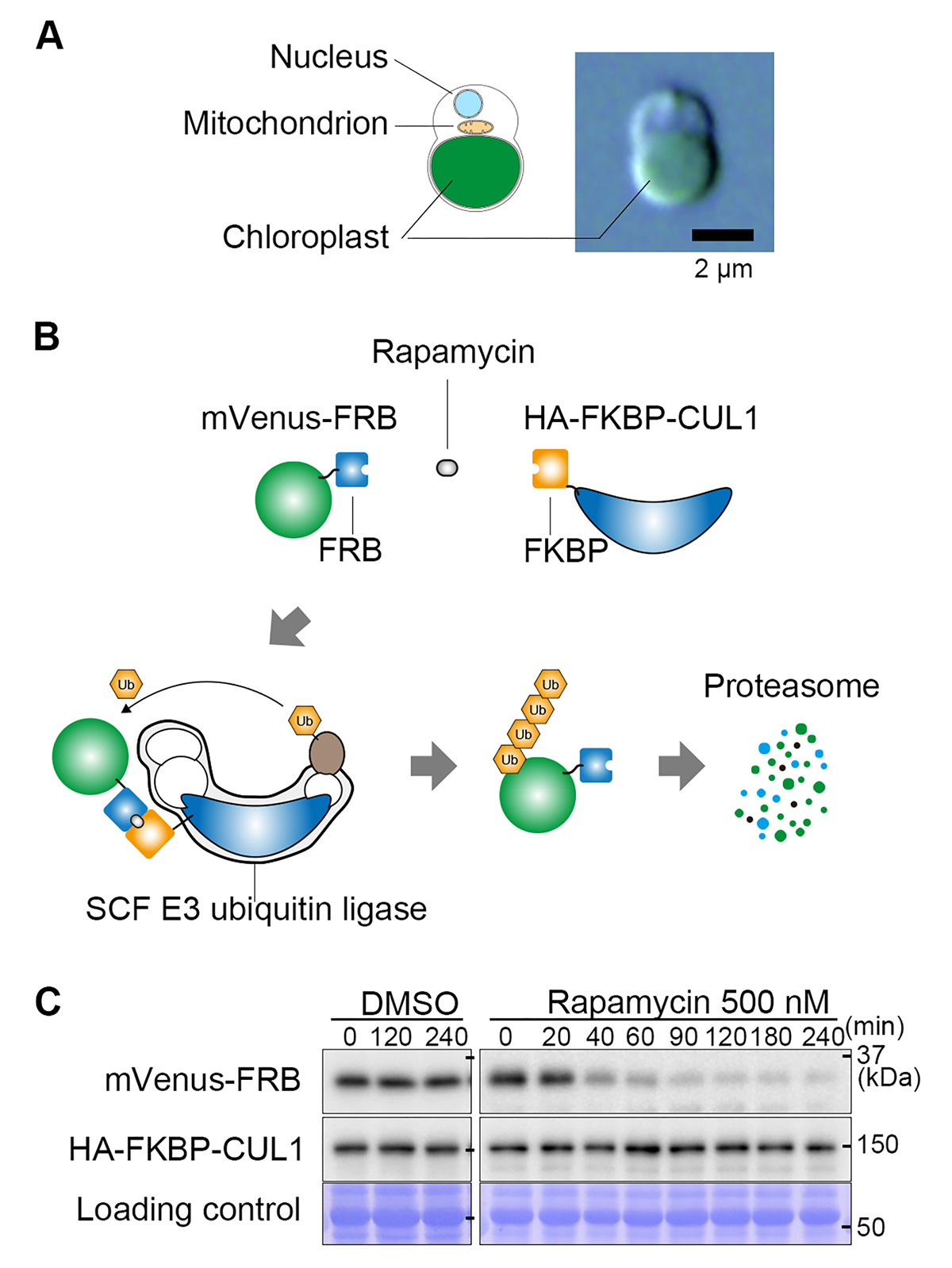
図:ラパマイシン誘導タンパク質ノックダウン法。
(A)単細胞紅藻Cyanidioschyzon merolaeの細胞内構造と微分干渉顕微鏡像。
(B)ラパマイシン誘導タンパク質ノックダウン法の仕組み。FRBタグとFKBPタンパク質はラパマイシン存在下でヘテロ二量体を形成する。FRBタグを融合した標的タンパク質(図では蛍光タンパク質mVenus)と、FKBPを融合させたユビキチンリガーゼのCUL1サブユニットは、ラパマイシン存在下で結合し、FRB-mVenusがユビキチン化される。ユビキチン化されたFRB-mVenusは速やかにプロテアソームにより分解される。
(C)ラパマイシン添加後のmVenus-FRBタンパク質レベルの減少を示すイムノブロット。
夏季休業のお知らせ(8/15-16)
本研究所は、下記のとおり夏季一斉休業を実施します。
ご不便をおかけいたしますが、ご理解とご協力のほどお願いいたします。
令和6年(2024年) 8月15日(木)~16日(金)
進化研究のモデル生物のトゲウオにおいて全ゲノムレベルでヒストン修飾を解析
Genome-wide analysis of histone modifications can contribute to the identification of candidate cis-regulatory regions in the threespine stickleback fish.
Okude, G., Yamasaki, Y. Y., Toyoda, A., Mori, S., and Kitano, J
BMC Genomics (2024) 25, 685 DOI:10.1186/s12864-024-10602-w
ゲノム中には、タンパク質やRNAをコードしない非コード領域が存在します。非コード領域の中にはプロモーターやエンハンサーなどの遺伝子発現制御に重要な領域があり、これらの領域に生じた突然変異によって進化が起こることが知られています。しかし、非コード領域の中からプロモーターやエンハンサーなどを同定することは通常なかなか困難なため、実際の野生生物の進化に重要な遺伝子発現の調節領域の変異に関する研究はなかなか進んでいません。
プロモーターやエンハンサーは、活性・抑制状態などに応じて特定のヒストン修飾を示します。そこで、ヒストン修飾を解析することで遺伝子発現の調節領域を検出することができ、ヒトやマウスなどでは全ゲノムレベルで解析されています。
このたび、生態遺伝学研究室の奥出絃太研究員と北野潤教授らの研究グループは、CUT&Tagという手法を用いて、進化研究のモデル生物であるトゲウオ科イトヨにおいて、全ゲノムレベルでヒストン修飾状態を解析しました。その結果、複数の遺伝子発現調節領域を検出し、そのうちのいくつかについては、岐阜県の河川集団(ハリヨ)と北海道のイトヨ太平洋集団の間で、転移因子による挿入・欠失変異が生じていることを見出しました。生態遺伝学研究室では、本論文で確立した手法を用いて野生生物の進化に重要な役割を果たした遺伝子発現の調節領域の変異を見出していきます。
本研究は、学術振興会、科研費、JST CRESTなどの支援を受けて実施されました。
遺伝研公式ウェブサイトの停止 (7月11日(木) 00:00~7月11日(木) 08:00)
以下の期日において、遺伝研公式ウェブサイトの断続的な停止を伴う保守作業が予定されております。
皆様のご理解とご協力をお願いいたします。
停止日時: 7月11日(木) 00:00~7月11日(木) 08:00
理由: 保守作業のため
Due to the facility maintenance, the NIG website service will be stopped several times on the following time.
Thank you for your understanding and cooperation.
Time and Date: July 11 Thu, 00:00 – July 11 Thu, 08:00.
種子が発芽する温度範囲を決める仕組みを解明
〜気候変動に対応した種子発芽制御技術への応用に期待〜
プレスリリース
The MKK3 MAPK cascade integrates temperature and after-ripening signals to modulate seed germination
Masahiko Otani , Ryo Tojo , Sarah Regnard , Lipeng Zheng , Takumi Hoshi , Suzuha Ohmori , Natsuki Tachibana , Tomohiro Sano , Shizuka Koshimizu , Kazuya Ichimura , Jean Colcombet , Naoto Kawakami
PNAS (2024) 121, e2404887121 DOI:10.1073/pnas.2404887121
種子が発芽する季節・タイミング(時季)は、種子自身が持つ休眠の状態と環境の温度の組合せによって決まります。種子成熟後の時間経過に伴う休眠性の低下は、発芽可能な温度範囲の拡大をもたらし、環境の温度がこの範囲に収まる時季に発芽します。種子は、本来と異なる時季に発芽してしまうと、個体の成長や種子生産がダメージを受けるため、生育に適したタイミングで発芽することが重要です。今回、明治大学農学部の川上直人教授、明治大学大学院農学研究科博士後期課程の大谷真彦(現:博士(農学)、明治大学研究・知財戦略機構 研究推進員)をはじめとする国際共同研究グループ(パリ-サクレー大学・フランス国立農業食料環境研究所のJean Colcombet博士、香川大学農学部の市村和也教授、国立遺伝学研究所の越水静助教ら)は、種子が発芽可能な温度範囲を決める仕組みを解明しました。この仕組みでは、酵母から植物、動物まで広く存在する細胞内情報伝達経路、MAPキナーゼカスケードが中心的な役割を持ち、発芽の適温でカスケードが活性化され、植物ホルモンの作用を調節して発芽を促し、発芽の温度範囲を決めていることを明らかにしました。このカスケードで働くタンパク質リン酸化酵素(キナーゼ)の一つ、MKK3は、コムギやオオムギでは小麦粉やモルト(麦芽)の品質を大きく左右する穂発芽関連遺伝子です。今回の研究は、種子が温度を感知して発芽の時季を制御する仕組みの解明につながるだけでなく、温暖化に対応し、安定した作物生産を可能とする、種子発芽制御技術の開発への応用が期待されます。
本研究の一部は、文部科学省私立大学戦略的研究基盤形成支援事業(S1411023)、明治大学新領域創生型研究(#229831002)の支援を受けて行われたものです。
本研究成果は、米国科学アカデミーが発行する総合科学学術誌「Proceedings of the National Academy of Sciences of the United States of America (PNAS)」に掲載されました。

図: MKK3-MAPキナーゼカスケードは種子の休眠状態と環境の温度情報を統合し、植物ホルモン代謝酵素遺伝子の発現制御を介して発芽を制御する
Pは、各キナーゼがリン酸化された活性化型であることを示す。
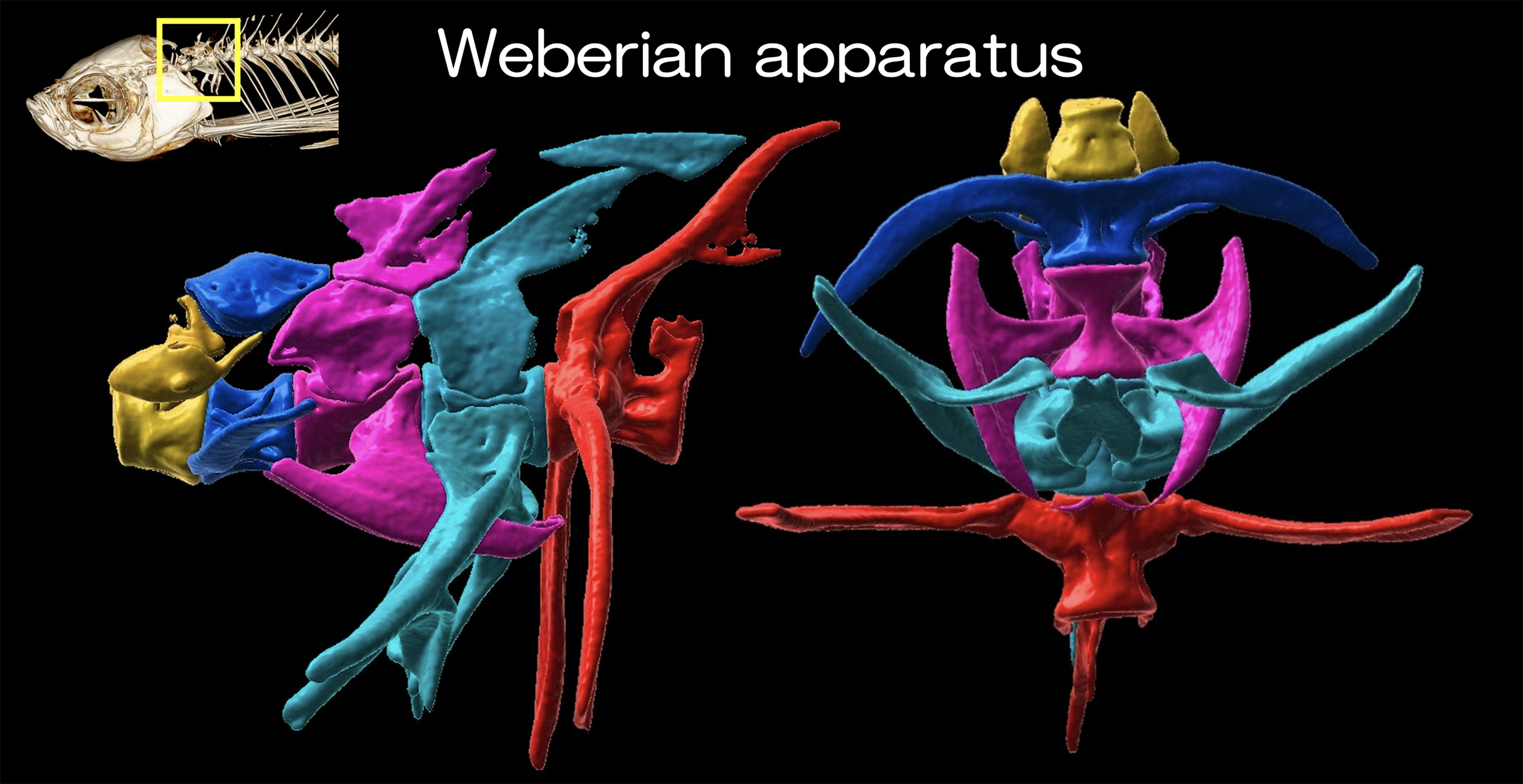
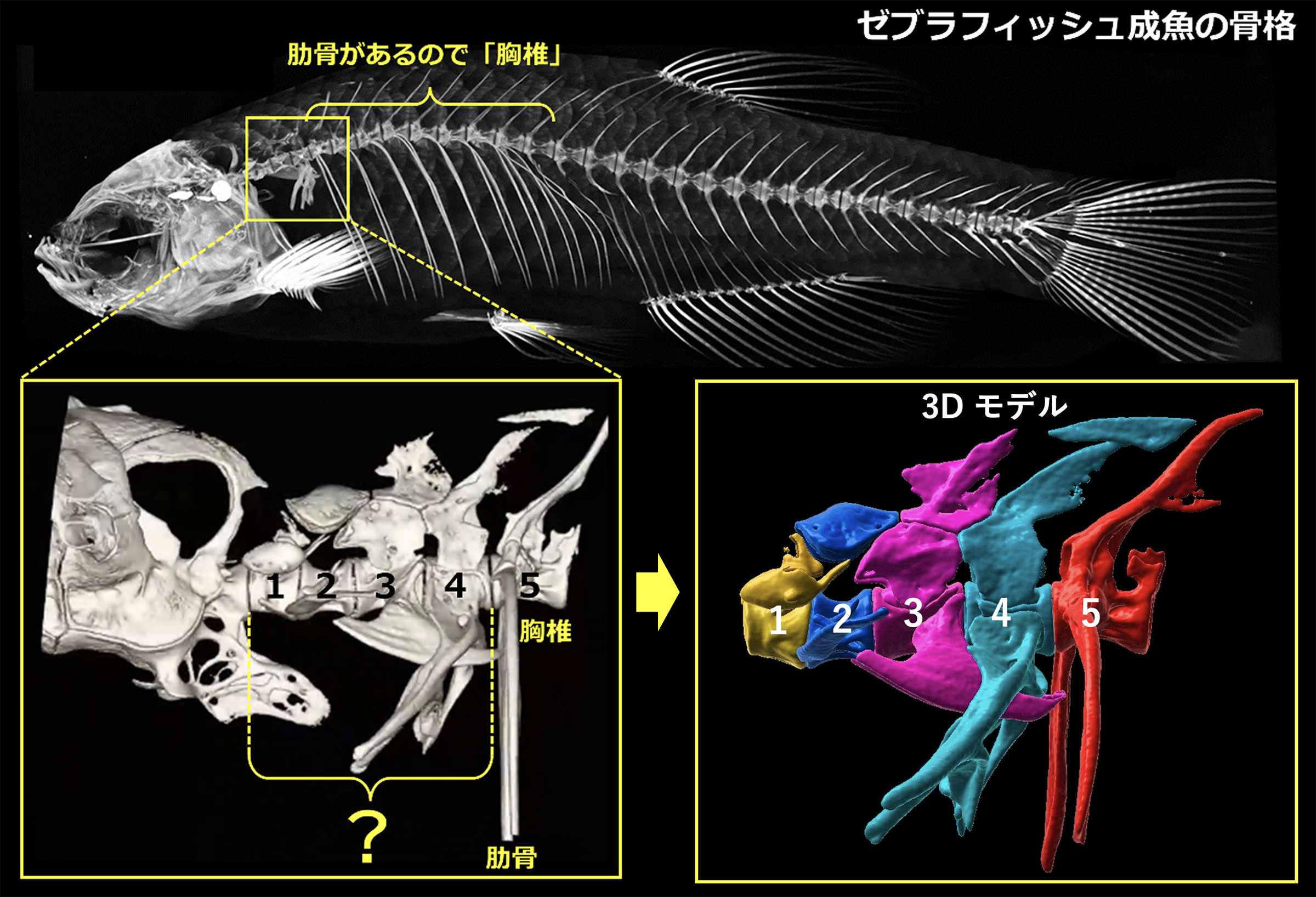
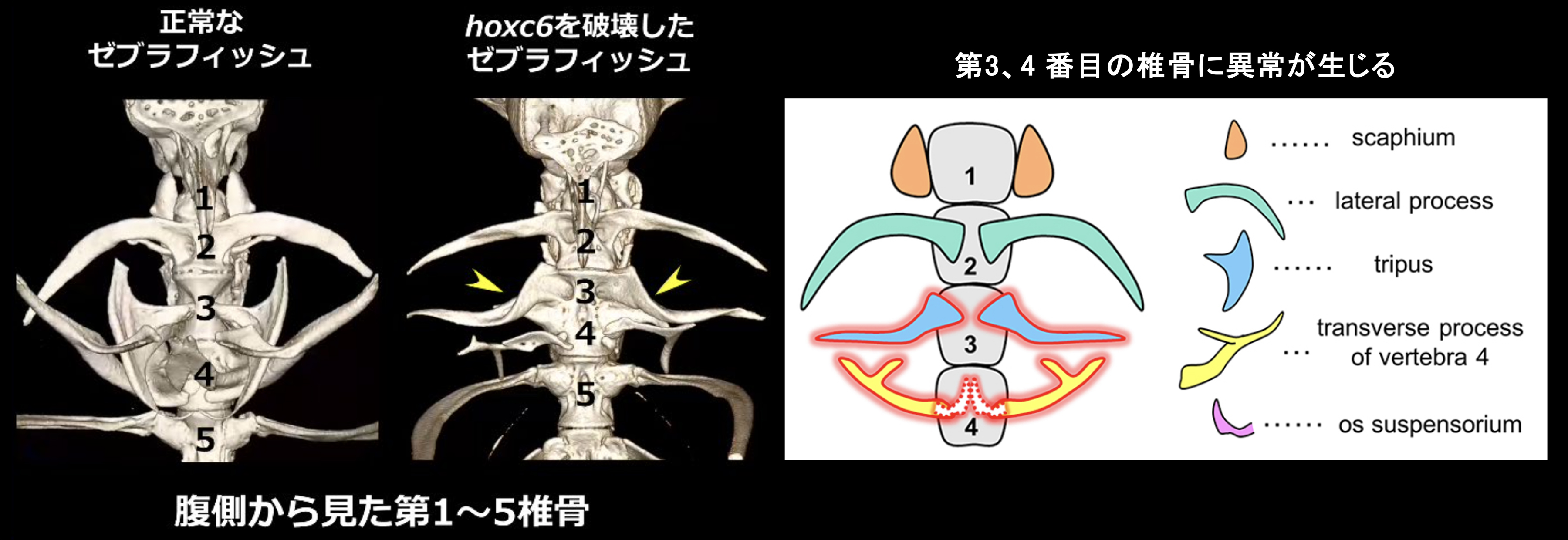
魚に首の骨(頸椎)はあるのか?
―脊椎動物の共有形質 「背骨」の進化を探る―
プレスリリース
技術課 / フェノタイプ研究推進センター / 細胞建築研究室
The Hox code responsible for the patterning of the anterior vertebrae in zebrafish
Akiteru Maeno, Rina Koita, Hidemichi Nakazawa, Renka Fujii, Kazuya Yamada, Sae Oikawa, Taisei Tani, Mizuki Ishizaka, Koumi Satoh, Atsuki Ishizu, Takumi Sugawara, Urara Adachi, Morimichi Kikuchi, Norimasa Iwanami, Masaru Matsuda, and Akinori Kawamura
Development 2024 Jun 28 DOI:10.1242/dev.202854
脊椎動物のからだに共通してみられる背骨は、椎骨が連なった繰り返し構造をしています。ヒトの各椎骨は前方から、首の骨である「頸椎」、肋骨と接続する「胸椎」、そして「腰椎」、「仙椎」、「尾椎」と分かれています。魚にも肋骨があり、魚も「胸椎」をもっていることがわかります。では、魚に首の骨、「頸椎」はあるのでしょうか。この問いは、脊椎動物の「背骨の進化」を紐解くうえで非常に重要です。
埼玉大学大学院・理工学研究科 生体制御学プログラム 川村哲規 准教授と国立遺伝学研究所 前野哲輝 技術専門職員は、宇都宮大学・バイオサイエンス教育研究センター 松田 勝 教授、岩波礼将 特任准教授らと共同で、椎骨の個性をもたらすHox(ホックス)遺伝子を壊したゼブラフィッシュやメダカを多種類作製し、どの位置の椎骨に異常が生じ、椎骨の個性を決めているかを調べました。その結果、魚の2番目の椎骨は「胸椎」と考えられ、魚の最前部の椎骨のみが「頸椎」と類似した性質をもつ可能性を示しました。本成果は、発生生物学の専門誌 『Development』に6月28日付でオンラインにて掲載されました。
本研究は、科学研究費補助金の基盤研究(C) 18K06177, 23K05790、国立遺伝学研究所公募型共同研究NIG-JOINT38A2019, 7A2020, 66A2021, 18A2022, 31A2023、成茂動物科学振興基金(2021年)によって行われました。
ヨモギにできる虫こぶの多様性を解明
プレスリリース
技術課 / フェノタイプ研究推進センター / 細胞建築研究室

Exploring the diversity of galls on Artemisia indica induced by Rhopalomyia species through morphological and transcriptome analyses.
Seiji Takeda, Makiko Yoza, Sawako Ueda, Sakura Takeuchi, Akiteru Maeno, Tomoaki Sakamoto, Seisuke Kimura
Plant Direct (2024) 8, e619 DOI:10.1002/pld3.619
京都府立大学の武田征士 准教授(細胞工学研究室)、国立遺伝学研究所の前野哲輝技術専門職員、京都産業大学の木村成介 教授(生態進化発生学研究室)らの共同研究グループは、ヨモギに作られる虫こぶの多様性を生み出す仕組みを明らかにしました。葉や茎に作られる虫こぶの形態と発現遺伝子の解析を行った結果、虫こぶでは、元の光合成器官(ソース器官)から、新たな花や実のような機能を持つ蓄積器官(シンク器官)への運命転換が行われていることが分かりました。小さな昆虫が植物の運命を大きく変えるこの能力の解明は、機能性成分を蓄積する植物器官の人為的誘導というような技術開発につながる可能性があります。
本研究は、文部科学省および日本学術振興会の科学研究費補助金(JP21K06234, JP21H02513)、国立遺伝学研究所NIG-JOINT(44A2020, 64A2021, 20A2022) の支援を受けて行われました。
本研究成果は、国際学術誌「Plant Direct」に、令和6年7月2日に掲載されました。
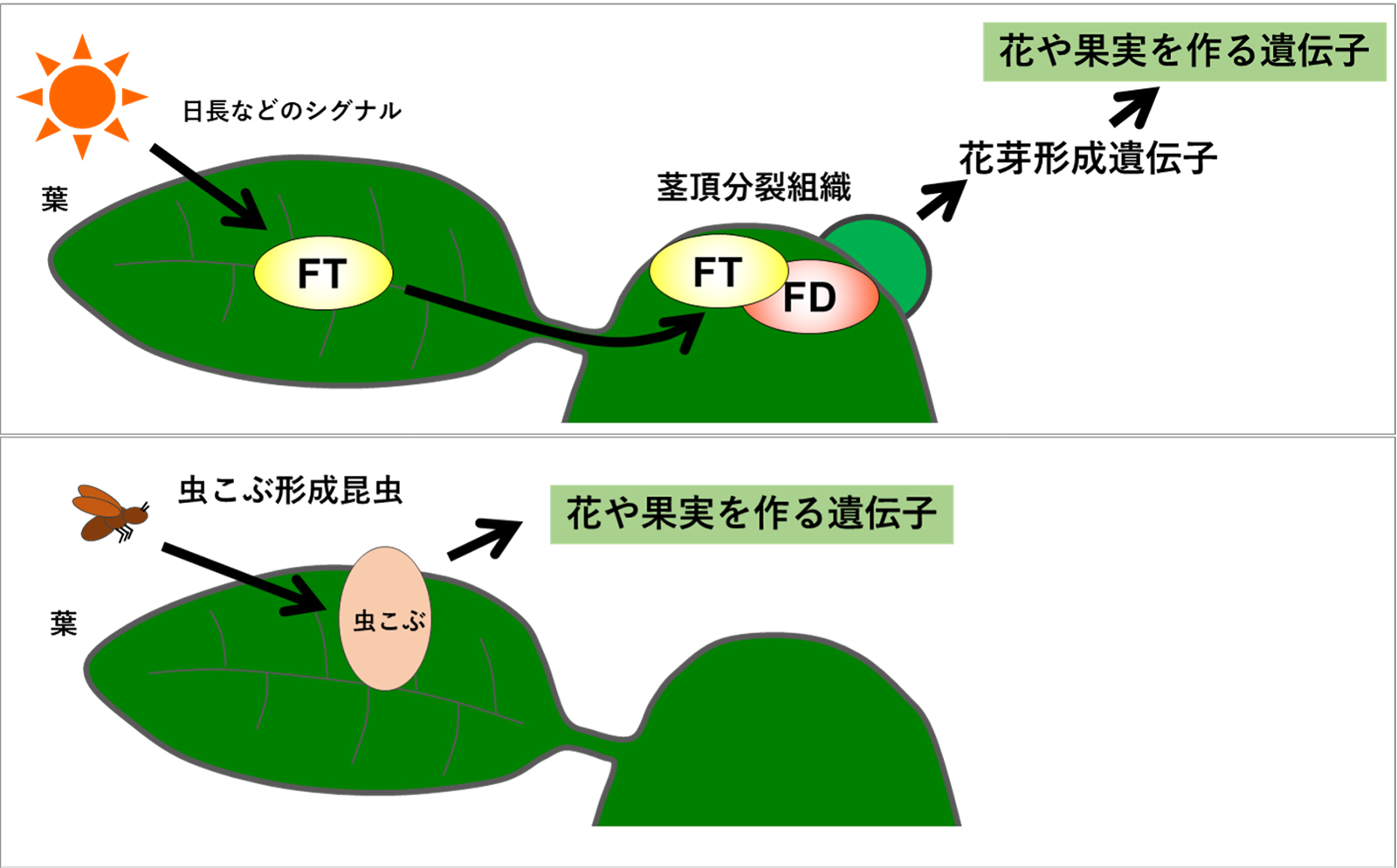
図: 虫こぶは、花や果実を作る遺伝子がいきなりオンになっている
動画: MicroCTによる虫こぶの形態観察
ヨモギにできる虫こぶの多様性を解明
プレスリリース
技術課 / フェノタイプ研究推進センター / 細胞建築研究室

Exploring the diversity of galls on Artemisia indica induced by Rhopalomyia species through morphological and transcriptome analyses.
Seiji Takeda, Makiko Yoza, Sawako Ueda, Sakura Takeuchi, Akiteru Maeno, Tomoaki Sakamoto, Seisuke Kimura
Plant Direct (2024) 8, e619 DOI:10.1002/pld3.619
京都府立大学の武田征士 准教授(細胞工学研究室)、国立遺伝学研究所の前野哲輝技術専門職員、京都産業大学の木村成介 教授(生態進化発生学研究室)らの共同研究グループは、ヨモギに作られる虫こぶの多様性を生み出す仕組みを明らかにしました。葉や茎に作られる虫こぶの形態と発現遺伝子の解析を行った結果、虫こぶでは、元の光合成器官(ソース器官)から、新たな花や実のような機能を持つ蓄積器官(シンク器官)への運命転換が行われていることが分かりました。小さな昆虫が植物の運命を大きく変えるこの能力の解明は、機能性成分を蓄積する植物器官の人為的誘導というような技術開発につながる可能性があります。
本研究は、文部科学省および日本学術振興会の科学研究費補助金(JP21K06234, JP21H02513)、国立遺伝学研究所NIG-JOINT(44A2020, 64A2021, 20A2022) の支援を受けて行われました。
本研究成果は、国際学術誌「Plant Direct」に、令和6年7月2日に掲載されました。

図: 虫こぶは、花や果実を作る遺伝子がいきなりオンになっている
動画: MicroCTによる虫こぶの形態観察
メール送受信の不具合について(7月4日12:45-13:45)
遺伝研のメールシステムのテストの影響で、7月4日12:45-13:45の間、遺伝研アカウントのメールが送受信できていない可能性があります。
ご利用の皆様にはご迷惑をおかけして申し訳ございませんが、ご理解とご協力をお願い申し上げます。
哺乳動物ゲノムのどこにDNA複製開始領域が存在するのか?(総説論文)
Replication initiation sites and zones in the mammalian genome: Where are they located and how are they defined?
Xiaoxuan Zhu, Masato T. Kanemaki*
*責任著者
DNA Repiar (2024) 141, 103713 DOI:10.1016/j.dnarep.2024.103713
ゲノムDNAがどのように複製されるかという問いは、分子生物学における重要テーマの一つであり、DNA複製異常はゲノム不安定性、遺伝疾患、細胞死、発癌に関係する。そのため、DNA複製は大腸菌や出芽酵母を材料として、これまで長年研究されてきた。これらの単細胞モデル生物では、DNA複製開始領域はDNA配列で規定されており、特定のコンセンサス配列を持つゲノム領域からDNA複製が開始する。一方で、ヒトを含む哺乳類動物細胞では、複製開始領域にコンセンサス配列は存在せず、ゲノム上のどこから開始するのかいまだに理解が進んでいない。そこで、本総説論文では、これまで報告されている大規模シーケンシングに基づく複製開始領域の探索成果を取りまとめて比較議論した。さらに、出芽酵母と哺乳動物細胞のDNA複製機構の共通性と違いに注目し、哺乳動物細胞において複製開始領域がどのように決定されるのか、その仮説メカニズムを提示し、将来のDNA複製研究の方向性と課題を議論した。
筆頭著者の朱孝軒博士(Zhu, Xiaoxuan)は遺伝研博士研究員として支援を受けました。
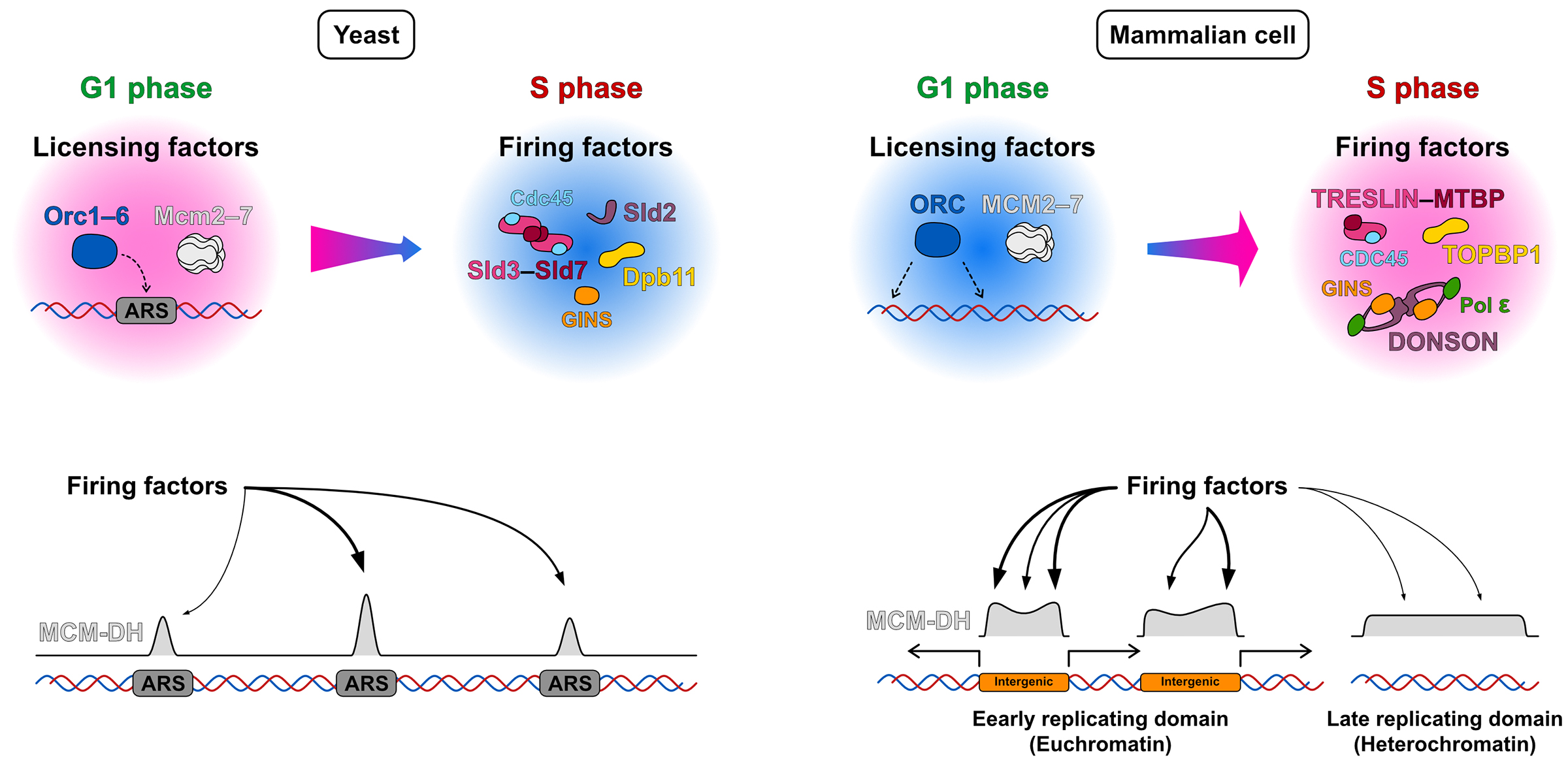
図:出芽酵母と哺乳動物細胞における複製開始領域選択メカニズムの違い。酵母においてはARS(autonomously replicating sequence)というコンセンサス配列を持つ部位にライセンシング複製因子が集積しDNA複製が開始する。一方、哺乳動物細胞ではライセンシング因子はゲノム上に広く分布しており、その後に集積する発火因子が制御を受けて特定の場所に集積することが、複製開始領域を作るのに重要ではないかと考えられる。
令和6年度 法人職員採用試験について 締め切りました
事務職員1名を募集します。
第一次試験「東海・北陸地区国立大学法人等職員採用試験」
第一次試験 7月 7日(日)
第一次試験合格者発表 7月25日(木)9時30分
■機関説明会■
第一次試験に合格された方を対象に、以下のとおり機関説明会を実施します。
(実 施 日) ①8月1日(木) 13:00~(1時間程度を予定)
②8月2日(金) 13:30~(30分程度を予定)
(会 場)①国立遺伝学研究所(https://www.nig.ac.jp/nig/ja/about-nig/access_ja)
②オンライン
(内 容)①機関紹介、質疑応答、職場見学を予定
②機関紹介、質疑応答を予定
(参加方法) 参加を希望される方は、7月30日(火)正午までに、以下URLよりお申込みください。
お申し込みいただいた方には、
個別に詳細をお知らせいたします。
(申込フォーム)https://forms.gle/q5c94mEBr8zRqL9BA
第二次試験(1次選考・最終選考)
■1次選考■
(対象者) 令和6年度東海・北陸地区国立大学法人等職員採用試験(事務)の第一次試験合格者
(日 時) 令和6年8月8日(木) 予備日:9日(金)
(会 場) 情報・システム研究機構国立遺伝学研究所 本館2階応接室
(https://www.nig.ac.jp/nig/ja/about-nig/access_ja)
(実施方法) 個別面接
(事前に送付する物) ・訪問カード(履歴、志望動機等)
※所定の様式をダウンロードして記入の上、8月7日(水)10時までに、人事・労務係( )宛てに送付願います。
(持参する物) ・第一次試験合格通知のメール(写)
■最終選考■
(対象者) 1次選考の合格者
(日 時) 令和6年8月21日(水)から8月22日(木)
(会 場) 情報・システム研究機構本部事務局
東京都港区虎ノ門4-3-13ヒューリック神谷町ビル2階
(https://www.rois.ac.jp/about/access.html)
※最終選考は、情報・システム研究機構本部で実施します。
【 申 込 方 法 】
令和6年8月5日(月)15:00までに
「受験番号、氏名、メールアドレス、電話番号」を人事・労務係( )宛ご連絡下さい。
*機関説明会及び面接にお越しいただく交通費は、自己負担となりますのでご了承願います。
問い合せ先:管理部総務企画課人事・労務係(担当:山口)
TEL: 055-981-6716 E-mail: