細菌共通の「合成困難なアミノ酸配列」確認──逆に利用するタンパク質群も発見
プレスリリース
Evolutionary Adaptation of Bacterial proteomes to Translation-Impeding Sequences
藤原圭吾、辻奈緒子、崎山歌恋、仁木宏典、千葉志信(Keigo Fujiwara, Naoko Tsuji, Karen Sakiyama, Hironori Niki, and Shinobu Chiba)
The EMBO Journal (2025)
細胞は、リボソーム上でアミノ酸をつなぎ合わせてタンパク質を合成します。細菌は短時間で多くのタンパク質を合成できるため、効率的に生育することが可能です。一方で、合成途中に現れる特定のアミノ酸配列が、リボソームを一時的に停止させる「難翻訳配列」として働く場合があり、大腸菌や枯草菌などの一部の細菌では、特殊な遺伝子発現制御系の中でいくつかの例が報告されています。
本研究では、ゲノム上に存在するタンパク質配列の中で、特定のアミノ酸配列の出現頻度を計算する独自のアルゴリズムを開発し、5万種を超える細菌を対象とした網羅的なゲノム解析を行い、難翻訳配列の探索を行いました。
その結果、地球上に生息する多様な細菌は共通して合成が困難なタンパク質のアミノ酸配列(難翻訳配列)を持つことが明らかになりました。さらに、難翻訳配列は、細菌のタンパク質内部にはほとんど見られないことが明らかになりました。これは、細菌が進化の過程で難翻訳配列の使用を回避する方向に適応してきたことを示唆しています。
一方で、比較的小さなタンパク質の末端(カルボキシ末端)付近に、難翻訳配列がしばしば存在することが分かりました。生物情報学的解析により、こうしたタンパク質群が、外部環境の変化に対して細菌が適応する上で、重要かつ多様な機能を果たしている可能性が示唆されました。
このように、一見すると細胞にとって不都合に思える難翻訳配列ですが、一般的にはタンパク質から進化的に排除される一方で、細菌はその特性を逆手に取り、特定の遺伝子発現の調節システムとして利用することで、環境変化へ適応するために役立てていることが明らかになりました。
本研究は、国立遺伝学研究所の藤原圭吾特命助教(JSTさきがけ研究員)がこの難翻訳配列の探索アルゴリズムを開発し、京産大チームと協力して研究を進めました。
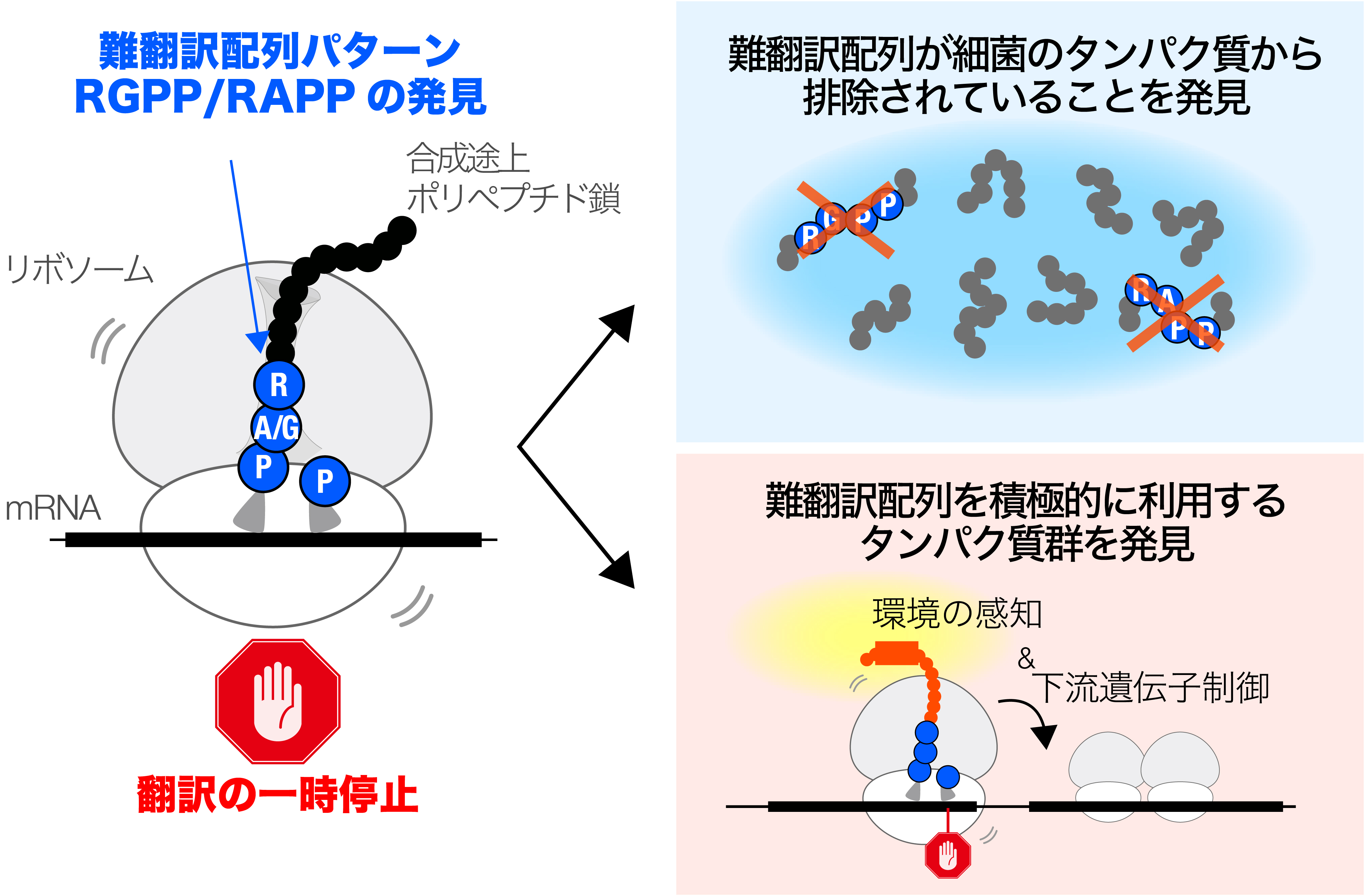
図:多くの細菌において「タンパク質の合成を止めてしまうアミノ酸配列」のパターンを発見(左)。そのような配列パターンは、一般的には、進化の過程で排除されるが(右上)、一方で、細菌は、合成困難性を細胞の機能維持に役立てるユニークなしくみを進化させることもある。















