ゲノム構造変異とリピート変異を配列識別して高精度に検出するソフトウェアTRsvを開発
プレスリリース
TRsv: simultaneous detection of tandem repeat variations, structural variations, and short indels using long read sequencing data.
Shunichi Kosugi, Chikashi Terao
Genome Biology 26, Article number: 246 (2025) DOI:10.1186/s13059-025-03718-z
個人間のゲノム配列の違いは、病気の罹りやすさを含めた様々な形質の違いを表しています。配列の違いを生み出すものの中で繰り返し変異(繰り返し配列のコピー数変異)は神経筋疾患や量的形質の要因となり、構造変異・インデル(欠失や挿入など)は神経発達障害や癌などの疾患要因となっています。しかし、これらを正確に区別して検出する解析手法はこれまでに存在しませんでした。
本研究では、ロングリードデータを用いて繰り返し変異と構造変異・インデルを配列識別によって正確に区別して同定するソフトウェア(TRsv)を開発しました。TRsvは既存のツールと比較してより高い繰り返し変異検出精度・感度を示し、繰り返し変異と構造変異をより高い精度で識別しました。さらに、160人のロングリード全ゲノムシーケンスデータを用いた解析において、TRsvは遺伝子発現、疾患、量的形質に関連する繰り返し変異を実際に検出できることを証明しました。今後ロングリードの活用が増す中で、TRsvはゲノムの繰り返し変異、構造変異・インデルを検出するツールとして広く活用されることが期待されます。
本成果は、情報・システム研究機構国立遺伝学研究所、同機構データサイエンス共同利用基盤施設、静岡県立総合病院、理化学研究所によるものです。
情報・システム研究機構国立遺伝学研究所 同機構先端ゲノミクス推進センター 特任准教授、データサイエンス共同利用基盤施設 ゲノムデータ解析支援センター 特任准教授および静岡県立総合病院 研究員 (研究当時)の小杉俊一と、 静岡県立総合病院 免疫研究部長、理化学研究所 生命医科学研究センター ゲノム解析応用研究 チームリーダーの寺尾知可史との共同研究成果です。
本研究は、日本学術振興会(JSPS)科研費(JP17K07264, JP21K06130)の支援を受け行われたものです。
本研究成果は、国際科学雑誌「Genome Biology」に2025年8月20日(日本時間)に掲載されました。
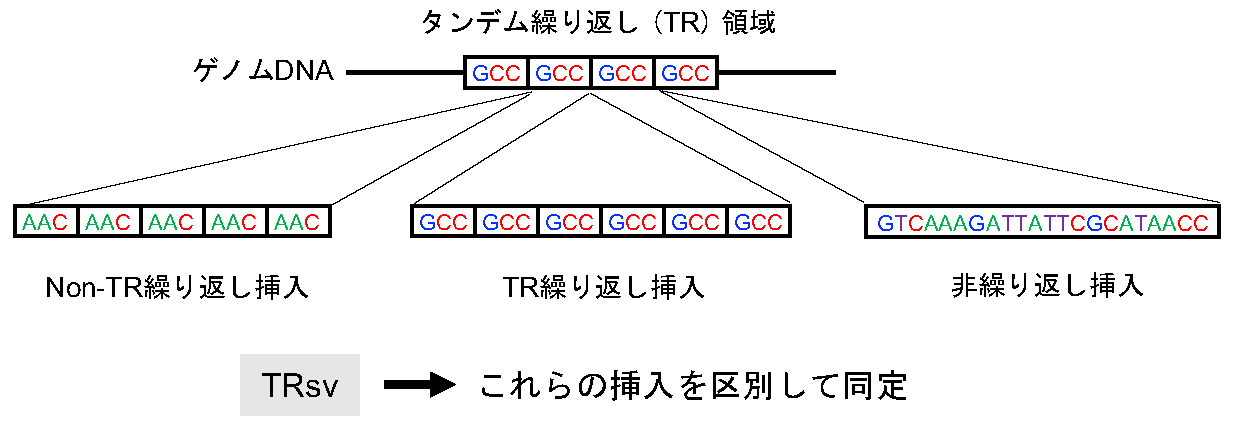
図1: TRsvはタンデム繰り返し領域で観察される異なるタイプの挿入を検出する
タンデム繰り返し(TR)領域内では、TR領域の繰り返し単位(図の例ではGCC)と同じ繰り返し単位からなるTR繰り返し挿入が観察されることが多いが、異なる繰り返し単位からなる挿入(Non-TR繰り返し挿入)や、繰り返しを持たない挿入(非繰り返し挿入)がしばしば観察される。TRsvは、これらの異なるタイプの挿入を区別して同定する。
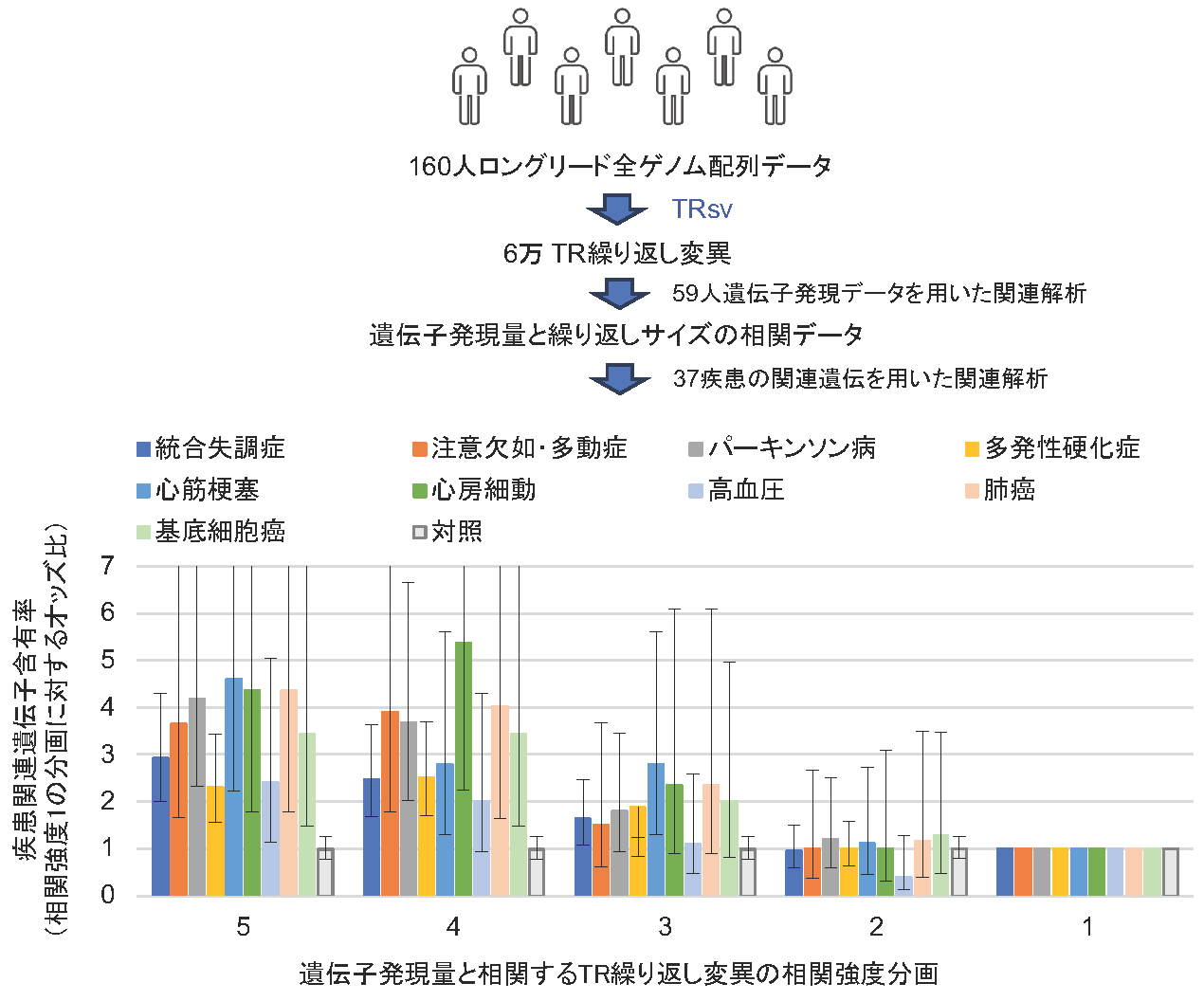
図2: 疾患関連遺伝子とTR繰り返し変異の関連がTRsvを用いて明らかにされた
160人のロングリード全ゲノム配列データからTRsvを用いて検出された6万個のTR繰り返し変異、および対応する59人分の遺伝子発現データを用いて、遺伝子発現量とTR繰り返し変異のサイズの相関が調べられた。その相関強度が強い順に5から1まで相関遺伝子セットを5等分に分画した(横軸)。各分画に含まれる疾患関連遺伝子の割合が計算され、相関強度1の分画の疾患関連遺伝子含有率に対する各分画の含有率の比(オッズ比)が各疾患毎にプロットされた(縦軸)。対照として、ヒト2万遺伝子から無作為に選別された800遺伝子セットのオッズ比が示される。バー上の黒線は標準誤差を示す。この結果は、多くの疾患に関わる遺伝子の発現がTR繰り返し変異によって制御されることを示すと共に、疾患の発症がTR繰り返し変異によって影響を受けることを示唆している。















