膨大なメタゲノムデータの相同性検索を可能にするシステム「PZLAST」
プレスリリース

PZLAST: an ultra-fast amino acid sequence similarity search server against public metagenomes
H. Mori, H. Ishikawa, K. Higashi, Y. Kato, T. Ebisuzaki, K. Kurokawa
Bioinformatics 2021 July 7 DOI:10.1093/bioinformatics/btab492
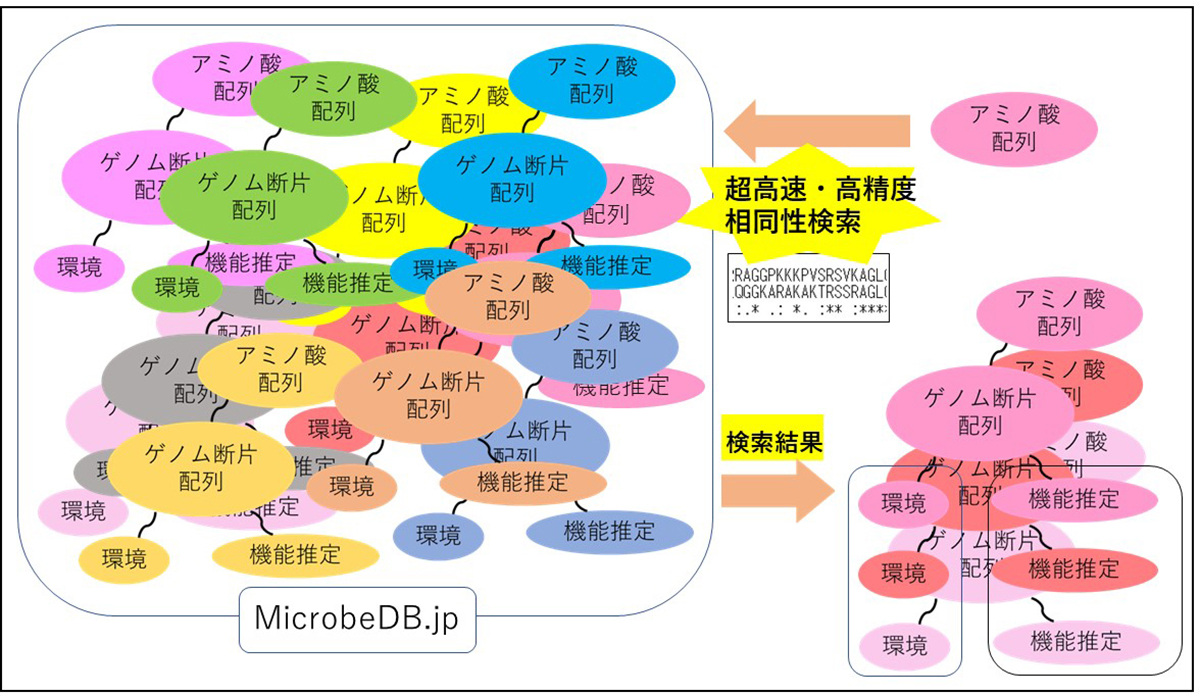
「環境」中の細菌集団の研究すなわち「マイクロバイオーム研究」が急速に進展したことにより、DDBJなどの公共データベースにはヒト腸内や土壌、河川、海洋など多様な環境に生息する細菌集団のゲノム断片(メタゲノム)データが大量に登録されています。これらのゲノム断片のデータは「遺伝子の宝の山」と言われていますが、その情報量があまりにも莫大であるため、「宝の山」を「発掘」するための解析技術の適用が困難で、「似た配列」を探し出す相同性検索すら難しい状態でした。
情報・システム研究機構 国立遺伝学研究所、理化学研究所、株式会社PEZY Computingの共同研究グループは、公開中の膨大なゲノム断片から予測したアミノ酸配列データをもとに極めて高速かつ高精度にアミノ酸配列の相同性検索を可能とするWebサービス「PZLAST」を開発しました。「PZLAST」を利用することで「遺伝子の宝の山」に埋もれている新たな遺伝子をその遺伝子が存在する環境の情報などとともに容易に「発掘」することが可能になったのです。
膨大なゲノム断片のデータが検索可能になったことで、薬剤耐性因子、病原因子やウイルスなど特定の遺伝子の環境中での動態や、新たな機能を持つ遺伝子の発見、遺伝子と環境の関係性の解析、創薬など、さまざまな研究の発展に寄与することが期待できます。
本研究は文部科学省高性能汎用計算機高度利用事業「ヘテロジニアス・メニーコア計算機による大規模計算科学(代表:姫野龍太郎)」の支援によりおこなわれました。
本研究成果は、国際計算生物学会誌「Bioinformatics」に2021年7月8日(日本時間)に掲載されました。
▶ PZLASTはこちらでご利用いただけます