微生物ゲノムの遺伝子アノテーションおよびDDBJ登録支援ツール DFAST
DFAST: a flexible prokaryotic genome annotation pipeline for faster genome publication
Yasuhiro Tanizawa, Takatomo Fujisawa, Yasukazu Nakamura
Bioinformatics 34(6) 1037-9, 2018 DOI:10.1093/bioinformatics/btx713
新型シークエンサーの利用が広がる微生物のゲノム研究においては大量のゲノムデータを高速に処理することが求められつつあります。また一方で、新規解読された塩基配列は公共塩基配列データベースに登録することが学術雑誌への論文掲載の必須条件となっており、煩雑な登録手続きは研究者の大きな負担ともなっています。DDBJ Fast Annotation and Submission Tool (DFAST) は微生物ゲノムアノテーションおよびDDBJへの塩基配列登録支援を目的に開発された解析パイプラインです。画面上の操作のみでDDBJへの塩基配列登録に必要なファイルを生成することができるウェブ版と、コマンドラインで動作し大量データ処理や解析ワークフローの自由なカスタマイズを特長とするスタンドアローン版が開発されており、https://dfast.ddbj.nig.ac.jp で利用可能となっています。典型的なゲノムサイズのバクテリアであれば5分でアノテーションを行うことができ、偽遺伝子の検出やセレノシステイン等のアミノ酸翻訳時の例外検出などの独自の機能も持っています。
実験研究者とバイオインフォマティシャンのどちらにとっても使いやすいツールを目指した本ツールにより、微生物研究におけるゲノムデータの利用を一層促進することが期待されます。
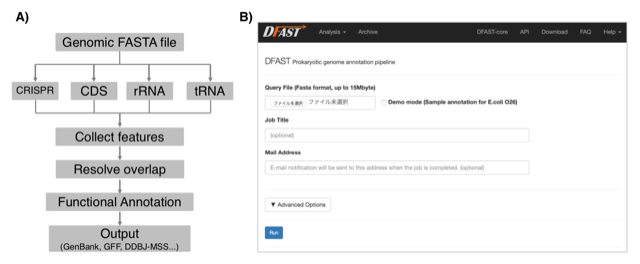
図:A) DFASTのアノテーションワークフロー。B) ウェブ版DFASTのジョブ投入フォーム















