「データ同化技術」を用いて細胞質流動の原動力を推定
Bayesian Inference of Forces Causing Cytoplasmic Streaming in Caenorhabditis elegans Embryos and Mouse Oocytes.
Niwayama R., Nagao H., Kitajima T. S., Hufnagel L., Shinohara K., Higuchi T., Ishikawa T., Kimura A.
PLoS ONE, Vol 11, e0159917 (2016). DOI:10.1371/journal.pone.0159917
国立遺伝学研究所と統計数理研究所、東北大学、理化学研究所、大阪大学、欧州分子生物学研究所の共同研究チームは、統計数理研究所で研究・開発されてきた「データ同化技術」を遺伝学に適用し、測定が困難な細胞内の力を推定することに成功しました。本研究で庭山律哉博士(総研大遺伝学専攻・日本学術振興会特別研究員、現・欧州分子生物学研究所研究員)らは、細胞内での大規模な物質の流動現象である細胞質流動に着目し、線虫(C. elegans)とマウスの細胞内の流動の方向性や速さを詳細に定量化しました。この計測データと流体力学シミュレーションモデルを統合したデータ同化によって、流動を引き起こす原動力の強さと分布を推定しました。推定された力の分布は、力の発生に重要な分子の分布と一致し、本手法の妥当性が支持されました。マウスと線虫では原動力の分布が異なっていましたが、その違いはそれぞれの細胞での流動の機能と合致していて、機能に適した力の分布をそれぞれの細胞がとっていることが示唆されました。本手法は生体内で生じる様々な流れの解析に適用可能で、今後多くの研究者に活用されることが期待されます。
本研究は情報・システム研究機構の「新領域融合センター」および「データ中心科学リサーチコモンズ」のプロジェクトとして遂行されました。新領域融合センターの合宿型会議である「クロストーク」で異分野の研究者が出会い、同センターの萌芽的研究を促進する「育成融合プロジェクト」の支援を受けて共同研究を開始しました。この間、生命科学および統計数理の両者の観点から魅惑的かつ適切な問題を探索し、取り組んできました。その後、データ中心科学リサーチコモンズ内の「データ同化」プロジェクトのサブ研究テーマとして共同研究を継続し、今回の成果発表につながりました。このプロジェクトの実施により、融合研究を開始するためのノウハウをよく理解し、高度な統計数理技術を習得した生命科学分野の若手研究者を育成し、広範な学術コミュニティに送り出してきました。長い時間はかかりましたが、情報・システム研究機構が推進する新領域創造型研究の好例となりました。この経験は「データサイエンス共同利用基盤施設」のデータ融合計算プロジェクトの運営に引き継がれています。
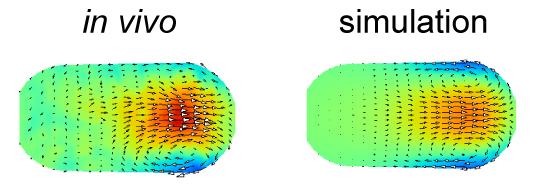
本研究で推定された原動力を用いた流体シミュレーション(右)は、実際の細胞内の流動(左)をよく再現しました。図は線虫胚での結果を示しており、色は流動の速さ、矢印は向きを表します。















