Mudi•ワンクリックで酵母の変異点を同定
大量遺伝情報研究室・中村研究室 細胞遺伝研究部門・小林研究室
Mudi, a web tool for identifying mutations by bioinformatics analysis of whole-genome sequence.
Naoko Iida, Fumiaki Yamao, Yasukazu Nakamura, and Tetsushi Iida Genes to Cells 28 APR 2014 DOI:10.1111/gtc.12151遺伝学は、変異株を単離することで未知の生命現象の分子機構や遺伝子機能を明らかにできる強力な手法です。しかし従来のクローニング法は、手間がかかり網羅的な変異同定が困難でした。近年、次世代シークエンサー(NGS)の進歩により、実験室単位での全ゲノムシークエンスが可能になってきました。NGSによる全ゲノムシークエンスとバイオインフォマティクス解析を取り入れた変異点同定法は、高速で強力なアプローチです。
一般の実験研究者自身が、全ゲノムシークエンス解析を行う際、バイオインフォマティクス解析の敷居が高いという問題があります。今回、私たちは、「Webからワンクリックで変異同定解析が出来るバイオインフォマティクスツール”Mudi”」を構築•公開しました。 (Mudi; http://naoii.nig.ac.jp/mudi_top.html)
Mudiは、戻し交配により分離した変異株プールと野生型株のゲノムリシークエンスデータをWebサイトからアップロードすれば、ワンクリックで解析が実行でき、短時間で変異点リストを得ることができるシステムです。現在、Mudiはゲノム情報がよく整備されたモデル生物として、出芽酵母(S. cerevisiae)と分裂酵母(S. pombe, S. japonicus)を対象に公開していますが、他生物への応用も可能です。このシステムによって、遺伝学的解析の高速化が期待されます。
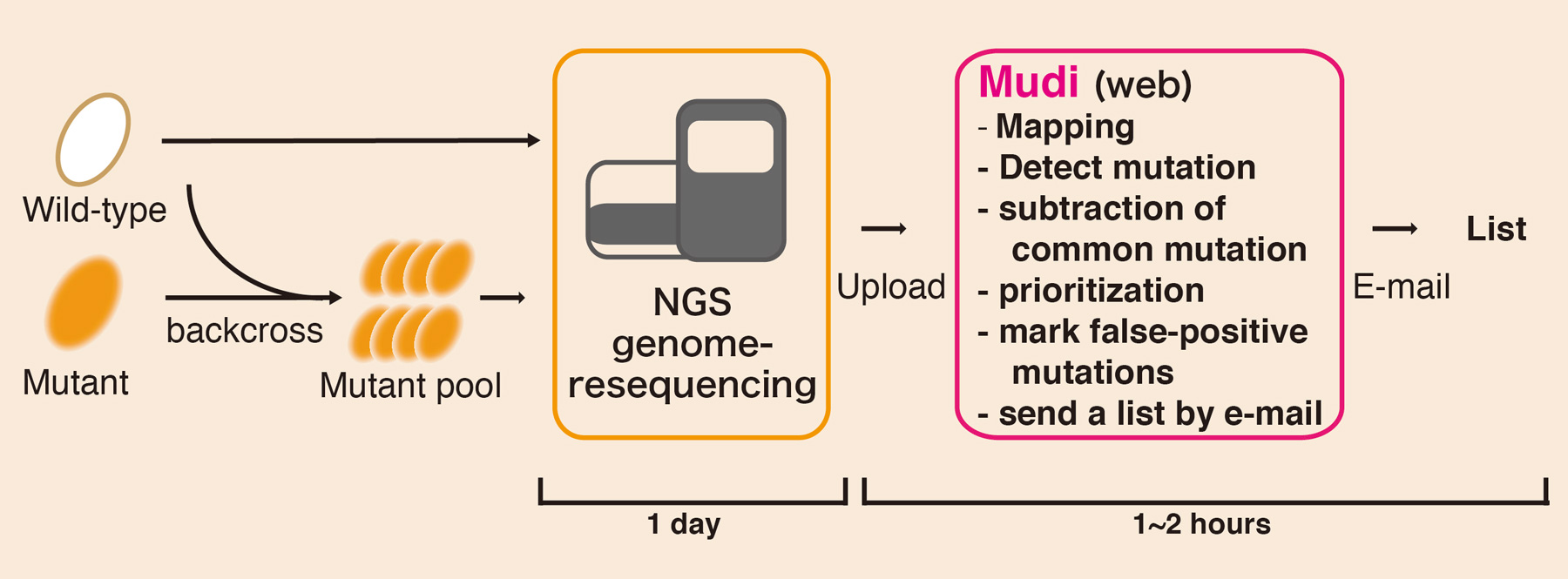
Webツール”Mudi”による変異点同定。戻し交配により分離した変異体プールからサンプル調整を行い、NGSによるゲノムリシークエンスを行う。Mudiツールによるバイオインフォマティクス解析により変異点を検出する。















