マウスゲノム内の超複雑な繰り返し領域の解析(新領域融合プロジェクトの成果です)
Umemori J, Mori A, Ichiyanagi K, Uno T, and Koide T.
BMC Genomics14: 455, 2013.
コピー数多型 (Copy number variation, CNV)は、ヒトおよびマウスゲノムの10%以上を占め、個体や系統間の遺伝的な多様性を決める重要な要素であると考えられています。
CNVはしばしば集合して、コピー数多型領域(complex CNV region, complex CNVR)を構成し、ゲノムの部分重複(Segmental duplication, SD)と呼ばれる、複雑な繰り返し配列を持つ領域をつくりだしています。
しかし、その非常に複雑な領域の解析は、ゲノム配列のほとんどが解読された現在においても、困難を極めています。今回我々は、国立情報学研究所との共同研究により、この複雑な領域の新たな解析方法を確立しました。
先ず我々は、国立情報学研究所、宇野毅明准教授が開発した、高速相同性検出アルゴリズム、Similarity/Homology Efficient Analyze Procedure (SHEAP)を用いることにより、マウスのlarge SD領域をゲノムワイドに検出しました。
その結果、large SDは、それぞれが独自の「タータンチェック模様」として視覚化されました (図1)。次に、遺伝的不適合性の原因領域である、第13番染色体上のSD (SD13M)に着目し(投稿準備中)、独自に開発した画像解析プログラム、blast-based Systematic analysis of HErPlot to Extract Regional Distinction (SHEPHERD)を用いて、繰り返しのコアとなっている配列を同定しました (図2)。
さらに、この領域について比較ゲノムハイブリダイゼーション(Array-comparative genomic hybridization, aCGH)を三島の野生マウス由来の系統MSMとブルガリアの野生マウス由来系統BLG2、実験用マウス系統C57BL/6Jについて行い、それぞれのコア配列の系統間のCNVを実験的に検出しました。この結果、これらのコア配列は、CNVsを持つものと、持たないものに分けられること、これらは含有配列などの性質が大きく異なることが示唆されました。
これらの解析結果は、今後complex CNVRの進化や機能を明らかにするための足がかりになると期待されます (図3)。
本研究は、情報・システム研究機構、新領域融合プロジェクト・育成融合プロジェクトの助成のもと行われました。
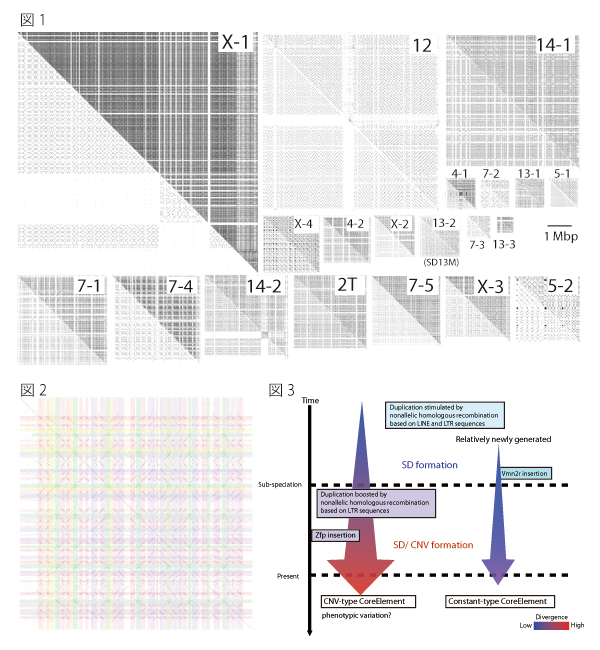
図1 SHEAPにより視覚化されたSDのタータンチェック構造
左上の数字はSDの染色体番号を示している。セルフプロットで示されたそれぞれのSDの左下はトランスポゾンなどの既知の配列をマスクしたもの、右上はマスクしていない配列を用いて解析を行なったもの。SD内の斜線は、長い繰り返し配列を表している。
図2 セルフプロットで示されたSD13Mと反復単位の抽出
斜線として視覚化された繰り返し配列について、X軸方向とY軸方向で重複するものを画像解析により間引き、コアとなる配列を選別する。
図3 SD13Mの進化および機能についての仮説
CNVsを持つコア配列は、LTR型のレトロトランスポゾンの割合およびDivergenceが高い。これは、CNVsを持つコア配列は古い年代からそのコピー数を増加させ、LTR型のレトロトランスポゾンの挿入による非アリル性の相同組換えが、そのコピー数の変動を加速させた可能性を示唆している。















