数理モデルからクロマチンの構造を知る
![]()
Dynamic nucleosome movement provides structural information of topological chromatin domains in living human cells
Soya Shinkai, Tadasu Nozaki, Kazuhiro Maeshima, Yuichi Togashi
PLOS Computational Biology. 12(10), e1005136 DOI:10.1371/journal.pcbi.1005136, 2016
真核生物における長大なゲノムDNAは、ヌクレオソーム、さらにはクロマチンを形成することによって極小な細胞核に収納されています。国立遺伝研の野崎慎(元・特別共同利用研究員/慶応大)、前島一博教授らは、生きている細胞のヌクレオソームを1分子レベルで観測することで、クロマチンの動的な性質の理解に努めてきました(図左)。ヌクレオソームの動きはブラウン運動のように、主に熱ゆらぎによって支配されていますが、実際に観察された動きはブラウン運動から予想される動きよりもはるかに小さく、これまで理論的に説明するモデルがありませんでした。今回、広島大学の新海創也助教、富樫祐一准教授らは、このヌクレオソームの動きを説明するための数理モデルを考案し、その動態を表現する理論式を導出しました。この理論式には、クロマチンの構造を表すパラメータが含まれているため、ヌクレオソームの動きからクロマチンの構造を理解できることが、理論的に明らかになりました。(図右)。そして、これまで観察されていたヌクレオソームの動きの違いは、クロマチン構造による拘束の違いに起因することが分かりました。さらに、実際に理論式と実験データを比較することによって、生細胞のヌクレオソームの動きからクロマチンの構造情報を得ることにも成功しました。
本研究での数理モデルに基づく解析を展開することで、クロマチンの「構造」と「動態」の関係性、さらに両者が支配する遺伝子発現調節機構の解明につながることが期待されます。
本研究は広島大学大学院理学研究科・クロマチン動態数理研究拠点との共同研究です。新学術領域「少数性生物学」のサポートを受けました。
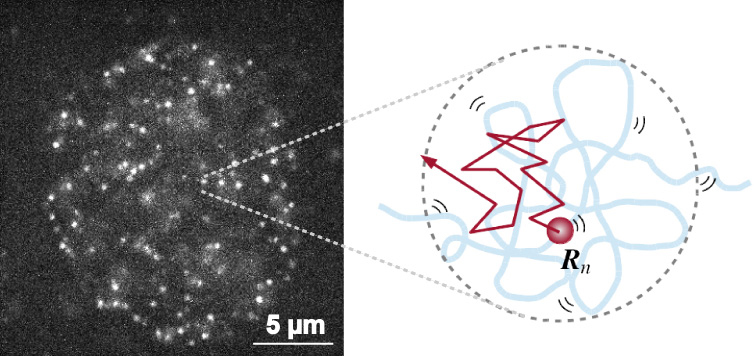
生きた細胞の核内のヌクレオソームを1分子レベルで観察した(左)。ヌクレオソームはクロマチン構造によって制限された中、ゆらいでいる(右)。ヌクレオソームの動きに対して、数理モデルを適用することで、クロマチン構造情報を得ることができる。















