両親由来のゲノム配列を個別に決定する新手法-ゲノム多様化領域に起因した生命現象の解明へ-
![]()
Platanus-allee is a de novo haplotype assembler enabling a comprehensive access to divergent heterozygous regions
Rei Kajitani, Dai Yoshimura, Miki Okuno, Yohei Minakuchi, Hiroshi Kagoshima, Asao Fujiyama, Kaoru Kubokawa, Yuji Kohara, Atsushi Toyoda & Takehiko Itoh
Nature Communications 10, Article number: 1702 (2019) DOI:10.1038/s41467-019-09575-2
東京工業大学、国立遺伝学研究所、東京大学の共同研究グループは、真核生物のゲノム配列決定において、両親由来の配列を区別し、高精度にそれぞれを決定する、新しい情報解析手法の開発に成功しました。
ヒトなど真核生物のゲノム情報は、両親から受け継いだ情報を持ち合わせていますが、今までは両親由来ゲノムの差異を無視して配列決定を行うことが一般的でした。しかしながら、この差異の大きな領域は、種々の昆虫の表現型(紋様)との関連や、ヒトでの免疫型の決定、さらには疾患との関連も報告されるようになっています。そのため、簡便に両親由来の配列を区別して解析できる手法が求められていたのです。
研究チームが開発した「Platanus-allee(プラタナス アリー)」と呼ばれる新しいプログラムは、特殊な装置や前処理を必要とせず、現在の主流になっている次世代シークエンサーの大規模な断片配列データのみから、両親由来の配列を高精度に再構築できる画期的なものです。
本成果は、2019年4月12日付けの「Nature Communications」に掲載されました。
研究支援
本研究は、文部科学省科研費「新学術領域研究『学術研究支援基盤形成』」先進ゲノム解析研究推進プラットフォーム(16H06279)および、16H04719, 15H0597などの支援を受けて行われました。
国立遺伝学研究所の貢献
比較ゲノム解析研究室および先端ゲノミクス推進センターは、両親由来の配列を区別して高精度にゲノム配列を再構築できる新たな手法の開発にあたり、さまざまな生物種から高分子かつ高品質なゲノムDNAを調製するとともに、現在主流となっている次世代型シーケンサを用いて、開発・評価用にショートリードやロングリードの配列決定を実施しました。
- Platanus-allee:http://platanus.bio.titech.ac.jp/platanus2
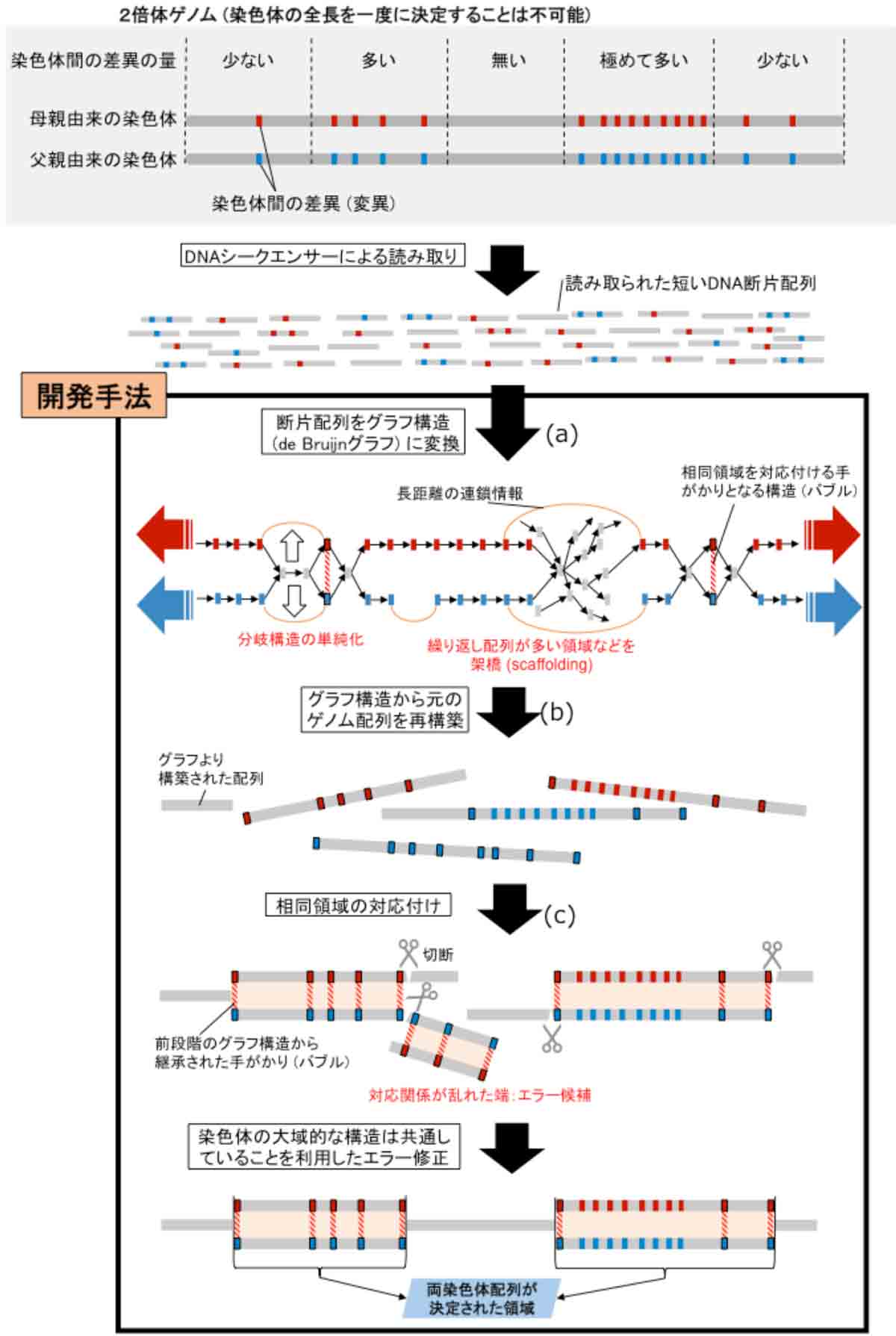
図:新たな情報解析プログラム「Platanus-allee」のアルゴリズムの模式図















