タマネギの品種育成の効率化に役立つ画期的なDNA多型分析手法を開発
プレスリリース
Development of a genome-wide marker design workflow for onions and its application in target amplicon sequencing-based genotyping
D. Sekine, S. Oku, T. Nunome, H. Hirakawa, M. Tsujimura, T. Terachi, A. Toyoda, M. Shigyo, S. Sato, H. Tsukazaki
DNA Research (2022) 29, dsac020 DOI:10.1093/dnares/dsac020
これまで、病気に強い、収量が高いなどの望ましい形質をもつ野菜を選び出すには、たくさんの個体の栽培、形質の調査、有望な個体の選抜を繰り返すことが必要であり、新しい品種の育成には多くの労力と長い時間を費やしてきました。品種育成の効率化には、DNA型の違い(DNA多型)を検出するDNAマーカーを開発し、活用することが有効です。特に、特定の形質と関連したDNAマーカー(選抜マーカー)は、DNA多型によって特定の形質が優れた個体を苗の段階で判別できるため、様々な形質について開発が望まれています。選抜マーカーの開発には、まず染色体全体のDNA多型を調べ、それらと形質データを照らし合わせて、目的の形質と関連するDNA多型の位置を特定する必要があります。近年は次世代シーケンサーの登場により、大量のDNA情報を安価に解読できるようになったため、染色体全体でのDNA多型の効率的な分析が可能になり、多くの野菜品目において選抜マーカーの開発が飛躍的に進んでいます。
しかし、タマネギでは染色体全体でのDNA多型の分析は容易ではありません。生物によって染色体全体のDNA情報であるゲノムサイズは、大きく異なります。タマネギのゲノムサイズは野菜の中でも最大級であり、その大きさはトマトの16倍もあります。そのため、トマトなど他の品目で使われてきた手法を用いて、タマネギの染色体全体でのDNA分析を行う場合、解析に要する費用や時間が大幅に増加することが予想されます。この手法でのタマネギのDNA分析は困難であり、タマネギでは染色体全体でのDNA分析法が確立していないため、選抜マーカーの開発や育種利用が遅れていました。
農研機構をはじめとする共同研究グループは、タマネギにおいて、染色体全体のDNA多型を効率的に分析する方法の開発を目指しました。まず、タマネギにある8本の染色体について、各々に圴一に配置され、染色体全体をカバーしたDNAマーカーセットを作成しました。次に、次世代シーケンサーを利用し、これらのマーカーセットの全てのDNA多型を一度にまとめて分析する手法を試みました。その結果、染色体全体のDNA多型を効率的に分析することに成功しました。この分析手法で得られた個体間のDNA多型と形質を照らし合わせれば、DNAマーカーセットの中から目的の形質と関連したDNAマーカーを特定でき、選抜マーカーとして利用できるようになります。この技術は、タマネギでの選抜マーカーの開発を飛躍的に進め、育種の効率化および新品種の早期育成に貢献することが期待できます。
本研究は、戦略的イノベーション創造プログラム(SIP):スマートバイオ産業・農業基盤技術「データ駆動型育種」推進基盤技術の開発とその活用による新価値農作物品種の開発 2. 農林水産省「戦略的国際共同研究推進委託事業」、JSPS科研費JP26292020、文部科学省 科学研究費助成事業新学術領域研究「学術研究支援基盤形成」先進ゲノム解析研究推進プラットフォーム(16H06279 (PAGS))の支援を受けて行われました。
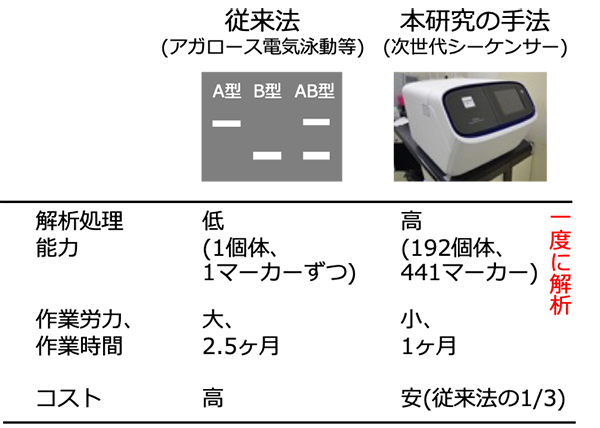
従来法では1個体、1マーカーずつを分析していたため、分析に係る作業時間や労力を多く費やしてきましたが、本研究では次世代シーケンサーの活用により、192個体、441マーカーを一度に分析できるため、分析の作業時間や労力を大幅に省力化できます。また分析に必要な試薬も少なくて済むため、分析にかかる費用も安くなります。















