従来の約100倍のサイズのゲノム編集が可能に! マウス・ラット等の遺伝子改変効率を向上させる新しい技術を開発
![]()
ssODN-mediated knock-in with CRISPR-Cas for large genomic regions in zygotes
Kazuto Yoshimi, Yayoi Kunihiro, Takehito Kaneko, Hitoshi Nagahora, Birger Voigt, Tomoji Mashimo
Nature Communications Published 20 January 2016. DOI:10.1038/ncomms10431
大阪大学大学院医学系研究科附属動物実験施設の真下知士(ましも ともじ)准教授、情報・システム研究機構国立遺伝学研究所マウス開発研究室の吉見一人(よしみ かずと)助教らの研究グループは、ゲノム編集技術‘CRISPR/Cas システム’と一本鎖オリゴ(ssODN)を利用する二つの新しい遺伝子改変技術の方法(「lsODN(長鎖一本鎖DNA)法」と「2H2OP(2ヒット2オリゴ)法」)を開発しました。
‘CRISPR/Casシステム’は、マウスやラットにおける新しい遺伝子改変技術として注目されている技術です。DNAを切断する酵素Cas9と、ゲノム上の編集箇所を見つけ出すgRNAを動物の受精卵に注入することで、特定の遺伝子を破壊(ノックアウト)したり、特定の箇所へ導入(ノックイン)することができます。しかしながらこれまで動物の受精卵では、遺伝子などの大きなDNA配列の導入効率が低く、ノックイン動物を作製することが困難でした。
本研究で開発した二つの新しい遺伝子改変技術の方法により、GFP遺伝子の効率的かつ正確なノックインに加え、これまで不可能だった大きなサイズのゲノム領域(約200 kbp)の導入、ラット遺伝子のヒト由来遺伝子への置き換え(遺伝子ヒト化動物)に成功しました。
今後、これら二つのノックイン法は、マウスやラットなどのみならず様々な生物種における遺伝子改変操作の効率を向上させ、新しい遺伝子組み換え生物の作製に非常に有用な技術になることが期待されます。また、作製された遺伝子改変動物は、創薬研究、トランスレーショナル研究、再生医療研究などへの幅広い利用が期待されます。
本研究成果は英国ネイチャー出版グループ オープンアクセス誌「Nature Communications」から公開されました。
本研究の一部は独立行政法人日本学術振興会 科学研究費助成事業(基盤研究(B))「実験用ラットにおけるゲノム編集基盤技術の開発」(課題番号:26290033、代表:真下知士)、 独立行政法人日本学術振興会 科学研究費助成事業(研究活動スタート支援)「CRISPR/Cas9を用いた多重遺伝子ノックアウトラット作製技術の開発」(課題番号:25890011、代表:吉見一人)の事業の助成を受けておこなわれました。
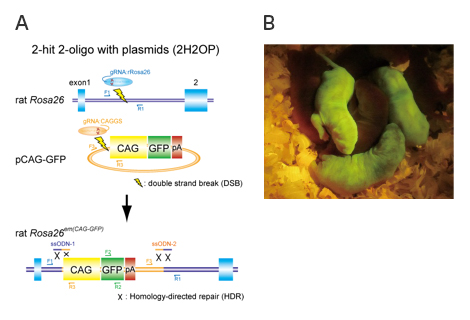
(A) 2H2OP法の略図
一本鎖DNA(ssODN)を‘のり’として利用することで、プラスミドを標的部位へ効率的にノックインできる。
(B) 2H2OP法により作製されたGFPノックインラット。
ノックイン個体は全身で緑色蛍光タンパク質を持つため、緑色に光る。















