線虫写真館
わたし、線虫(Caenorhabditis elegans)は、遺伝学では今やなくてはならない生き物なんです。そこで皆さんに、私のかわいい姿をお見せしましょう。
- 野生型
線虫(Caenorhabditis elegans)
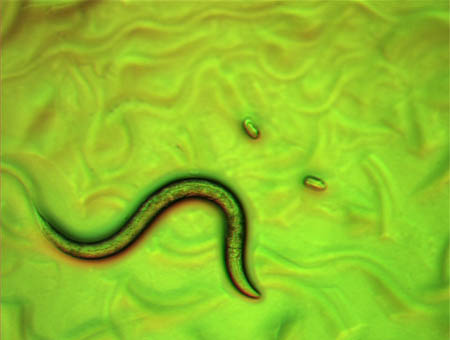
これは、線虫の幼虫の顕微鏡写真です。線虫は、ヘビのように体を波打たせて寒天の上を這い回っています(たくさんの波型は、餌の大腸菌の上をはったあとです)。1分間に30-40回ほど体をくねらせて進みます。後退することもできます。右上の楕円形の小さなものは、線虫の卵です。
原図、資料:石原健、桂勲
正常型とも言って、野生(自然)集団にふつうに見られるもののことです。この野生型と変異体とを比較することによって、遺伝学的研究を進めています。
- 雄と雌雄同体
線虫には、雄と雌雄同体がいます。雄は精子だけを作ります。雌雄同体は精子と卵を作り、個体内で自家受精することにより子孫を残します。一方、雄は雌雄同体と交尾することにより子孫を残すことができます。
雌雄同体の顕微鏡写真(右は尻尾の拡大写真)
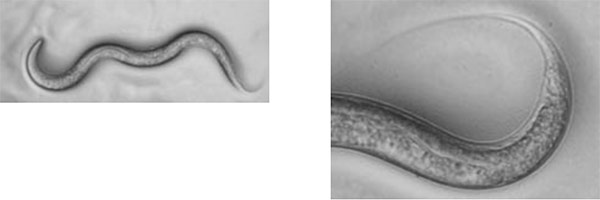
雄の顕微鏡写真(右は尻尾の拡大写真)
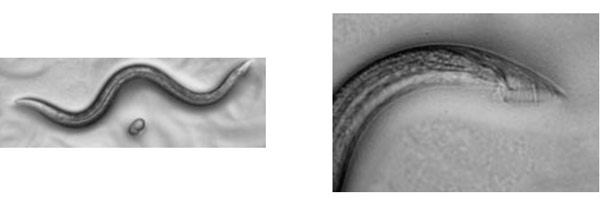
上の写真で右は、尻尾の先を拡大したものですが、雄の尻尾は交尾をするためにかぎ型になっていることがわかります。ここで、雌雄同体の体を触って、陰門を探し出します。
原図、資料:石原健、桂勲
- 変異体
これまでに、たくさんの変異体がとられてきました。ここでは、下皮に異常がある変異体を紹介します。
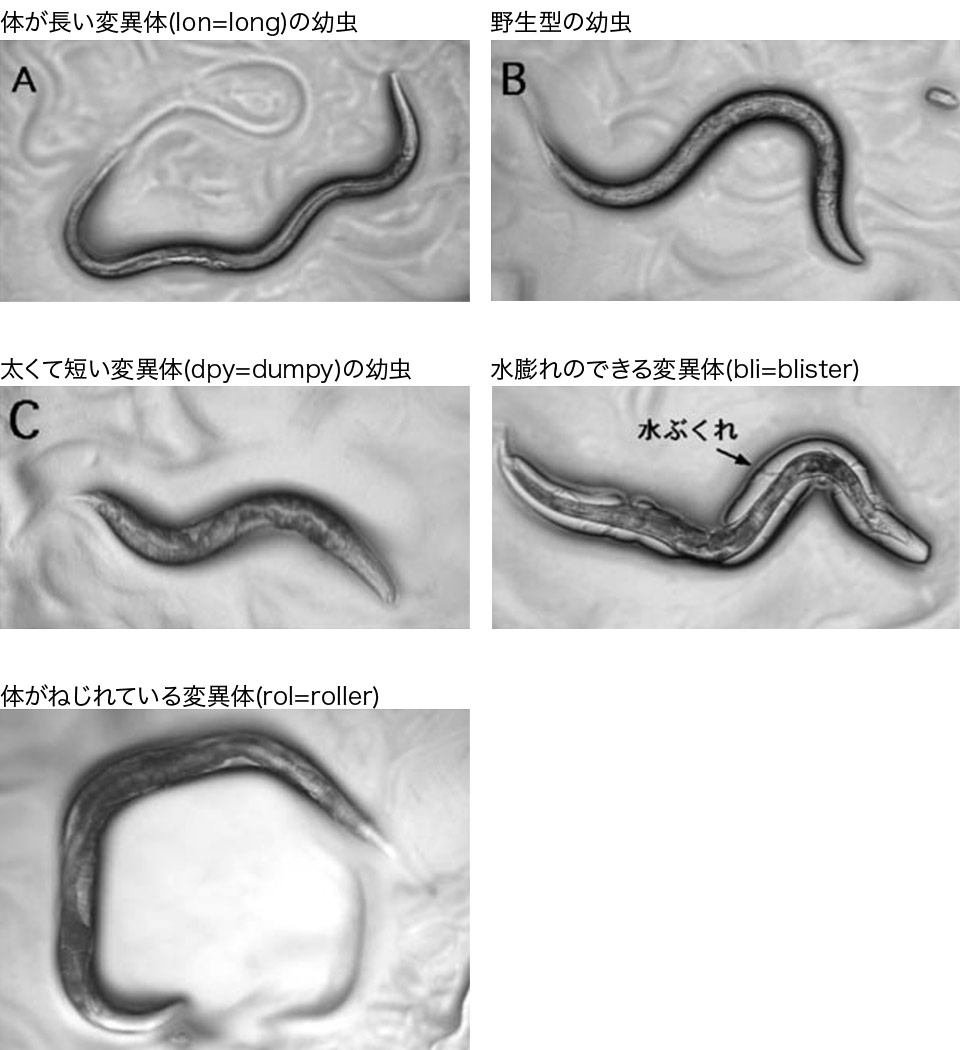
これらの変異体は、線虫の体を覆っているクチクラの構成成分であるコラーゲンなどの異常が原因で起こると考えられています。
原図、資料:石原健、桂勲
- 微分干渉像
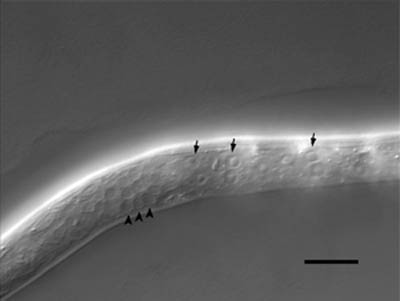
これは、一齢幼虫の微分干渉顕微鏡写真です。微分干渉顕微鏡という、特殊な装置のついた顕微鏡で線虫を観察すると、細胞の核を一つ一つ観察することが出来ます。
このことを利用して、受精卵から成虫までの全ての細胞が同定されています。また、核の形や大きさは、細胞の種類によって異なります。矢印で示した大きな核は、下皮細胞の核です。一方矢尻で示した小さめのものは、神経細胞の核です。スケールバーは10マイクロメートル。原図、資料:石原健、桂勲
- 神経に蛍光色素を取り込んだ線虫
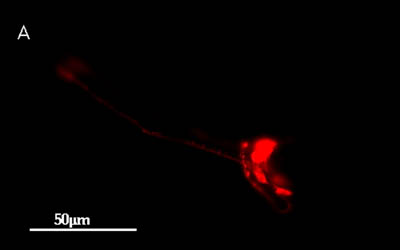
蛍光顕微鏡像
微分干渉顕微鏡像(上と同じ線虫)
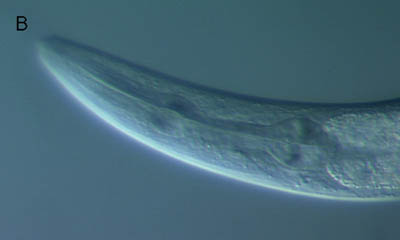
線虫を赤い蛍光色素(DiI)に浸すと、一部の神経に色素が染み込みます。上の写真は、そのようにした線虫の蛍光顕微鏡写真で、頭部では6対の感覚神経で蛍光が観察されます(下の写真は、同じ線虫の微分干渉顕微鏡写真です)。これらの神経は、味や匂いを感じる感覚神経であると考えられています。周りの化学物質を感覚するため、神経の先端が外界に露出していて、そこから蛍光色素をとりこんでいきます。これらの神経の先端が短いために味や匂いが感じられない線虫では、この蛍光色素を取り込むことはできません。
原図、資料:石原健、桂勲
線虫「1000細胞のシンフォニー」
- 線虫って何?
では、線虫というのは、いったいどんな生物なのでしょうか。線虫の種類はたいへん多いのです。たとえば、寄生虫の回虫が線虫の仲間です。また、松枯れ病の原因である松食い虫に寄生するマツノザイセンチュウも仲間です。寄生虫ですから、医学の1分野として古くから興味がもたれて研究されてきました。また、受精卵の発生が早く再現性が高いこと、細胞数が少ないことなどから、発生観察の材料として19世紀にはすでに研究されています。たとえば、ウマカイチュウの卵の研究から、生殖系の細胞と体細胞系の細胞との区別が発生初期に起きることや、体細胞系の細胞で染色体削減が見られることなど、発生学の古典的な研究が行なわれました。しかしその後、実験発生学は両生類を使って発展し、遺伝学はショウジョウバエを使って発展したため、生物学の材料として線虫は忘れ去られた存在になってしまいました。移植実験や再生実験ができなかったことや、その当時、線虫は寄生性のものが使われていたため、遺伝解析には向かなかったからです。
一方、寄生しない種類もあります。土の中に棲む線虫や、海に棲む線虫がそれです。これにもたくさんの種類がありますが、個体数は動物界でもっとも多いといわれていますし、また地球上のバイオマスの15%を占めているともいわれています。生態系の均衡にはたしている役割は、はかりしれないものと思われます。このように多種多様な線虫ですが、大きさはちがっても、その構造や発生はとてもよく似ています(本書の2章を参照)。進化的には図3に示すように、かなり古い時代に脊椎動物や昆虫と分かれています。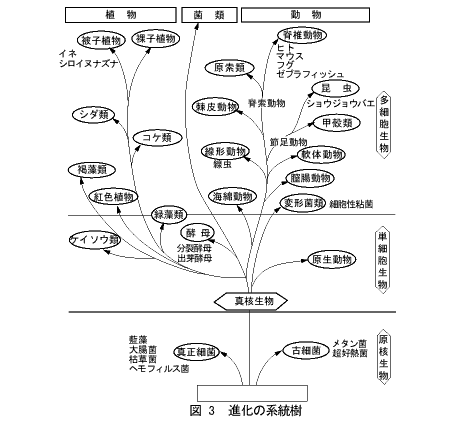
小原雄治著「線虫[1000細胞のシンフォニー]」(共立出版)より引用
- 変異から発見された遺伝子の例
変異の種類 遺伝子 [形態] からだが太く短い dpy からだが長い lon からだが小さい sma からだがねじれている rol 水泡ができる bli からだが透明 clr 核小体の形態異常 nuc 雄の形態異常 mab [行動] 運動の異常 unc 高浸透圧忌避の異常 osm 産卵の異常 egl 走化性の異常 che, tax, odr 摂食の異常 eat 胴体部の触覚異常 mec 脱糞の異常 aex, pbo, exp [発生] 細胞系譜の異常 lin 細胞死の異常 ced 耐性幼虫形成の異常 daf 致死 let 胚発生の異常 emb 生殖細胞の増殖異常 glp 孵化の異常 hch 精子形成の異常 spe 性決定の異常 tra, her, fem [薬剤耐性] レバミソール耐性 lev フッ素イオン耐性 flr アセチルコリンエステラーゼ阻害剤耐性 ric アマニチン耐性 ama [物質・酵素活性] カテコールアミン分布異常 cat コリンアセチルトランスフェラーゼ異常 cha 小原雄治著「線虫[1000細胞のシンフォニー]」(共立出版)より引用
- 分子遺伝学からのアプローチ:はじめに
1974年にブレンナー(Sydney Brenner)はC.elegansの最初の論文を発表しましたが、同じ号のすぐあとにサルストン(John Sulston、図1)のブレンナーの論文が続いています。タイトルは「C.elegansのDNA」。線虫のゲノムDNAの性質を調べたものです。そこでは、線虫DNAは大腸菌DNAの20倍の大きさであること(つまり、100Mbで、多細胞生物としては最小の部類に入る)、くり返し配列は少なく83%はユニーク配列であること、などが報告されています。あまりめだたない論文でしたが、あとあととても大きな意味をもってくる内容でした。
小原雄治著「線虫[1000細胞のシンフォニー]」(共立出版)より引用
- ゲノムの物理地図づくり
物理地図と遺伝地図のちがい
ブレンナーは最初の論文で、300余りの形態や行動異常の突然変異体を分離したこと、遺伝解析の結果6つの関連群(染色体)の約100の遺伝子に割り振った(マップした)ことを報告しました。これが遺伝地図です。遺伝地図は、遺伝子が直線状に並んでいて、遺伝子間の距離が大きいほどそのあいだでの組換えの頻度が高いことを前提に、遺伝子の並び方を決めたものです。しかし、組換え頻度は場所によって異なりますから、2点間の隔たりは実際の隔たりを必ずしも反映しません。
一方、物理地図というのは、実際の「もの」(この場合は染色体DNAですが)の地図をいいます。DNAの物理地図というと、古くは電子顕微鏡で見たファージDNAなどの変性地図をさしましたし、組換えDNA技術が現われると、制限酵素の切断点の地図というふうに変貌してきました。いずれの場合でも、2点間の距離は(精度はともかく)何らかの物理的な長さです。したがって、DNAの究極の物理地図は塩基配列ということになります。
物理地図は、遺伝地図とのあいだが関係づけられると大きな意味をもちます。発生、分化、行動など高次の生命現象にかかわる遺伝子には、たとえば酵素活性を測るといった簡便な検定法がありません。突然変異体が得られることで初めて遺伝子の存在が明らかになり、他の遺伝子との組換え実験から遺伝子の位置を決めていくのです。しかし、先のような前提がありますから、その位置は必ずしも正確とはいえません。ところが、図2に示すように、DNAの物理地図があって、近くの遺伝子がたとえば一里塚のようにマーカーとして物理地図上の位置がわかっていれば、状況はずいぶんと変わってきます。マーカー遺伝子のあいだに、目的の遺伝子の位置をしぼることができるからです。そして、もしこの間のDNAが扱いやすい形の組換え体で準備されていたら、どうでしょう。数がうんと少なくなりますから、組換え体クローンをひとつひとつ詳しく調べることができます。たとえば、DNA導入などの方法をとることが可能になるのです。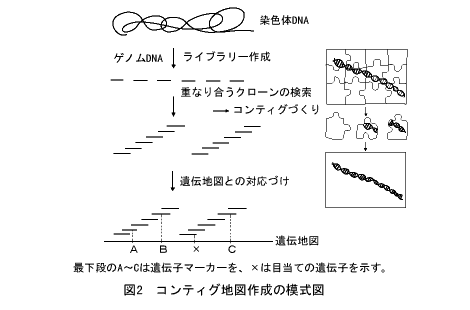
染色体を「歩く」
このやり方は1980年代に入って、まず「染色体ウォーキング法」としてショウジョウバエのバイソラックス(Bithorax)遺伝子のクローニングで成功しました。まず、スタートのマーカー遺伝子クローンの端の部分を標識します。これを用いて、ハイブリダイゼーション法によってゲノムDNAライブラリーから少しずれて重なる隣りのクローンを分離します。次いで、このクローンの端の部分を標識して、さらに隣りのクローンを分離するというやり方です。「対岸」のもうひとつのマーカー遺伝子にたどり着けば、目的の遺伝子はその間のクローンのどれかにあるはず、ということになります。染色体を一歩一歩進んでいくので「ウォーキング法」と名づけられたのです。
全ゲノムの物理地図:コンティグ地図づくり
染色体ウォーキング法は強力ですが、難点があります。手間がかかることです。一歩や二歩ならいいのですが、歩く距離が大きくなると、とたんに大変になってきます。しかし、このようにしてつくられた、たがいに少しずつ重なり合ったクローンのセットはとても便利です。このようなクローンの集まりを「コンティグ」(contig)とよぶようになるのですが、それならば最初から染色体すべてをコンティグでカバーしてしまえばよいのではないかと考える人が出てきました。サルストンもその一人で、1983年ごろからC.elegansゲノムの地図づくりを始めたのです。
ゲノムのジグソーパズル
コンティグ地図づくりはジグソーパズルに似ています。ジグソーパズルでは、ピースをその色や模様をたよりにつないでゆきます。隣りあっていればぴったりはまりますし、逆にぴったりはまりあうピースは1組しかありません。色や模様の片鱗から、ピタリとあったピースが見つかったときの快感はやった人でないとわかりませんが、これがハイブリダイゼーションによる「一歩」にあたります。空や海の単調な部分はむずかしく、なかなか進みません。高度くり返し配列の領域といえましょうか。
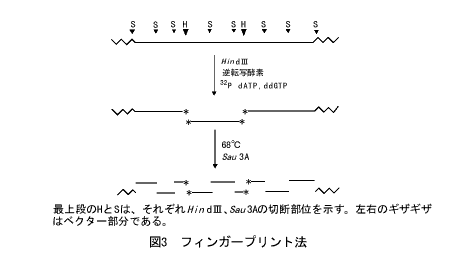
しかし、もうすこし要領よくできないものかと考えるのが科学者です。色や模様が合うように見えても、必ずしも隣りのピースとは限りません。ピタリとはまりあって初めて判定ができます。それなら、最初からピースの形を比べればよいのではないでしょうか。ウォーキング法では、ハイブリダイゼーションで候補のクローンが得られたら、必ず制限酵素地図をつくって比較し、重なっていることを確認します。それなら逆に、制限酵素地図を指紋照合のようにして、コンティグをつくっていけるはずです。フィンガープリント法
サルストンたちは、フィンガープリント法というやり方を開発しました(図3)。重なり合うクローンを制限酵素で切断すると、重なり合った部分からは同じ長さの断片ができるはずですが、これを目安にするものです。正確な判定のためには、断片の長さの精度と数が適切でないといけません。このために、サルストンたちはコスミドクローンを用い、2種類の制限酵素を使いました。コスミドは40kb程度のゲノムDNAを含みますが、これを6塩基認識のHindIIIという酵素で切断し、できた末端を32Pで標識します。次いで、4塩基認識のSau3Aという酵素でさらに小さく切断し、できた断片を塩基配列決定用のゲル電気泳動で分離して、オートラジオグラムを撮ります。4塩基認識の酵素の切断点は平均256塩基に1カ所ありますから、とても多くの断片ができますが、この方法では最初に32Pで標識した末端を含む断片のみがX線写真に写ります。平均23本のバンドが十分な精度で検出できるのです。これをデジタイザーで測定し、データベース化してコンピュータで比較し、同じ長さの断片をもつクローンを見つけてコンティグをつくっていくのです。解析クローンがふえるにしたがい、コンティグどうしがつながり、より大きなコンティグができていきます。
イギリス人のがんばり
こう書くと、いかにも自動化されて簡単なように聞こえますが、判定がむずかしい場合は人間が確認しなければなりませんし、大量のクローンの処理も大変です。しかし、イギリス人はへこたれないようです。とくに、サンガー(Fred Sanger)が引退して、腕ききのテクニシャンだったアラン・クールソン(Alan Coulson)が加わったことが大きかったようです。以来、実質的に二人三脚で進んできました。サルストンはこの仕事を始めるにあたり、コンピュータの勉強から始めたといいます(要するにコンピュータは素人だったのです)。周囲の専門家から手ほどきを受けながら、測定→判定→データベース化のプログラムをつくっていったのです。また、X線写真のデジタイザーも研究所の1階にある工作室の協力で自作しています。このあたり、イギリス人科学者の面目躍如たるものがあると思いますが、いかがでしょうか。
コスミドからYACへ:コンティグ地図の完成
1987年5月の時点で、コスミド16,000クローン(ゲノムの約6周分)を解析した結果、770のコンティグに分類されました。ゲノムの90%似上はカバーされていると推定されましたが、これ以降、新たに調べたコスミドクローンはほとんど既存のコンティグに含まれてしまい、コンティグのつながりがかなり困難になりました。コスミドでは、クローニングできにくい領域があるためです。
しかし、天の配剤といいましょうか、1987年はYAC(yeast artificial chromosome)が発表された年です。YACはアメリカ・セントルイスのオルソン(Maynard Olson)の研究室で開発されたものですが、同じフロアにボブ・ウォーターストン(Bob Waterston)がいました。彼もブレンナーの弟子で、C.elegansの筋肉遺伝子の研究者です。ウォ―ターストンは、すぐにC.elegansのゲノムDNAからYACライブラリーをつくりました。当時、セントルイスでも一番できがよかったライブラリーだそうです。そこで、YACクローンでコスミドコンティグをつなごうという話になり、YACクローン約1000個がケンブリッジに送られてきました。ちょうど私が客員研究員としてサルストンの研究室に着いてまもなくのことでした。
こまかいことですが、このとき使っていたコスミドクローンのベクターは「ロリスト」(Lorist)といって、ラムダファージがベースなので、YACベクターとは相同性はありませんでした。このため、YACクローンのDNAをパルスフィールド電気泳動のゲルから切り出して、そのまますべてを標識し、コンティグの代表コスミドを並べたメンブレンフィルターにハイブリダイゼーションするという乱暴なことが可能でした。そこで、3人で思い切ってやってみようということになり、サルストンがパルスフィールド電気泳動でYACクローンのDNAを切り出し、私が大量のハイブリダイゼーション、クールソンがデータ解析、という分担で、1,000個のYACクローンの解析を数ヶ月でやってしまいました。その結果、コンティグの数はみるみる減少し、たしか250になったと覚えています。
その後はクールソンが同じような解析を続け、6本の染色体がそれぞれほぼひとつながりのコンティグになりました。また、コンティグ地図作成の途中から、世界各地でクローン化された遺伝子がケンブリッジに送られ、フィンガープリント法などでコンティグとの対応づけが行なわれました。そのため、加速度的に遺伝地図と物理地図の対応が密になり、染色体のほとんどの部分がYACとコスミドで対応づけられたのです。小原雄治著「線虫[1000細胞のシンフォニー]」(共立出版)より引用
- 全ゲノムの塩基配列決定計画
3年間のパイロット計画
ゲノムのコンティグ地図が完成に近づいたころ、ヒトゲノム解析計画が本格化しました。紆余曲折はあったのですが、C.elegansゲノムの塩基配列決定が3年間のパイロット計画として1990年の夏から始まることになりました。アメリカの国立衛生研究所(NIH)が資金の3分の2を、イギリスのMRC(Medical Research Council、医学研究協議会)が残りの3分の1を出して、これをケンブリッジのサルストンとセントルイスのウォーターストンが折半して、2つのグループが共同で作業を進めるという、前代未聞のサポート体制でスタートしました。これは、ある意味ではうまいやり方です。大西洋をはさんで、イギリスはアメリカに絶対負けたくありませんが、研究費が不足しています。アメリカのほうでも1つの研究室ではつぶされてしまうかもしれませんが、国際協力となるとそうもいかない、といった具合いです。
それはともかく、第3染色体の中央部から両側に、それぞれのグループが塩基配列決定を始めました。ただし、当時の最長塩基配列決定は、同じケンブリッジのバレル(Bart Barrell)のグループによるサイトメガロウイルスの229kbでしたし、蛍光DNAシークエンサー(塩基配列決定機)の性能もまだ未知数が多く、最初は方法論の選択が中心課題でした。ショットガン法かプライマーウォーキング法か、シークエンサーの機種選定などが問題だったようです。そのうち、方法論もショットガン法とウォーキング法を組み合わせるという方法に落ち着き、ABI蛍光シークエンサーをずらりと並べるやり方で、2年目に800kb、3年目には3Mbと、順調に配列決定が進んでいきました。セントルイスでは、シークエンサーの1台は改良用にいつも壊されている状態で、このようななかからロングゲルや64レーン泳動が実用化されてきました。このあたり、すごいなと思います。遺伝子の予測:情報グループの活躍
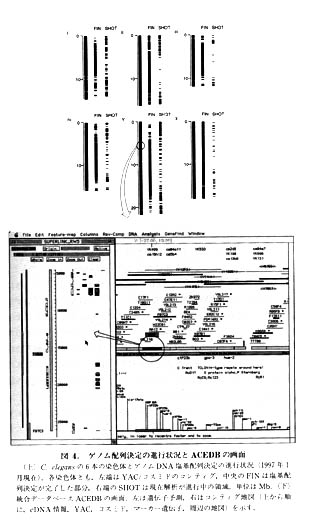
ゲノムのコンティグ地図が完成に近づいたころ情報科学(informatics)のほうも開発が進みました。レーントラッキングの補正、配列データの処理、アセンブリなどは自前のシステムが構築されました。さらに、遺伝子予測プログラムGenefinderが開発され、次々とエキソンや遺伝子が予測されていきました(図4左下)。あとで述べますが、cDNAの解析も進みましたので、遺伝子予測の能力も向上しました。この結果、C.elegansの遺伝子はだいたい5~10kbに1個の頻度で存在し、全遺伝子数は約15000と推定されました。
サンガーセンターの設立:C.elegans計画の完成へ
さて、3年間のパイロット期間が順調に終了するころ、次の計画で一悶着が起きました。サイエンティフィックでなくて申しわけありませんが、要するに研究費のめどが立たなくなったのです。1992年の初めに、あるうわさが流れました。アメリカの富豪がDNA塩基配列決定の会社をシアトルにつくる計画を立て、ついてはサルストンとウォーターストンをC.elegans計画ごと引き抜こうとしている、というものでした。サルストンに問い合わせると、多額の研究費が必要だからそのソースについてはあらゆる可能性を検討しているが、ゲノム研究はその成果がすみやかに公開されなければならないので、その会社に移るつもりはまったくない、とのことでした。しかし、「アメリカの企業がイギリスのサイエンスを買収する」ということで、イギリスではたいへんな騒ぎになったそうです。MRCはきゅうきょ対策に乗り出し、結局、イギリスの富裕な財団であるウェルカムトラストとともにDNA塩基配列決定の新しい研究所、サンガーセンターをつくることになりました。サルストンはここの所長になり、C.elegansの配列決定を続けるとともに、ヒトゲノム解析にも乗り出すことになりました。アメリカでもウォーターストンをヘッドとする配列決定センターがセントルイスにできました。
そして見事に予定通り1998年12月にほぼ全配列(97Mb)が「Science」誌に発表されました。論文の著者は総勢400人(ケンブリッジ関係150、セントルイス関係250)という大仕事でした。ここから約19,000の遺伝子がコンピュータプログラムで予測されました。ちなみにヒトゲノムの遺伝子総数は6-10万と考えられていますが、驚くべきことに、これまでにヒトでクローン化された約5,000の遺伝子のうち74%は非常に似た遺伝子がC.elegansゲノムにも見つかったのです。これまでにも「細胞死」に関する遺伝子のようにヒトとC.elegansで交換が効く遺伝子もありましたが、こういう例がもっと増えるでしょう。また、C.elegansのゲノムは多細胞生物で初めて配列決定されたわけですが、単細胞生物の酵母にもバクテリアにもない遺伝子が多数見つかっています。これらは細胞間の情報伝達に関わる遺伝子が多く、多細胞生物の本質に関わるものと考えられます。全ゲノム配列をもとに、全遺伝子の働きを調べて生物全体の遺伝子プログラムを解こうというプロジェクトがすでに始まっています。本当にちっぽけな虫ですが、C.elegansは今後明かにされるヒトゲノムの働きを解き明かすために重要な情報を提供することが期待されます。小原雄治著「線虫[1000細胞のシンフォニー]」(共立出版)より引用
- cDNAの解析
3年間のパイロット計画
cDNAは、mRNAをDNAの形に変えたものです。私たちは後で述べる発現解析のために、まず実際に生体内で使われている形の遺伝子のセットをそろえる計画を立てました。これがcDNAクローンのシステマティックな解析です(図5)。発生の全ステージをカバーするcDNAライブラリーをつくり、ここからcDNAクローンをランダムに拾います。そして、cDNAの両端から一回の作業で300~500塩基の配列をどんどん決めていきます。これをタグ配列とよびます。cDNAは全長とは限りませんので、5'側はクローンごとにまちまちですが、ポリA鎖からcDNAを合成してあるので、3'側は同じところから始まっています。したがって、3'側のタグ配列を比べれば、まるで指紋照合のように同じ遺伝子かどうかを見分けることができます。こうやって、cDNAクローンを分類してくのです。
mRNAの発現量は遺伝子によって大きくちがいますから、この方法で100%採れる保証はありませんが、簡単で有効なやり方です。1997年2月の時点で30,000クローンを調べて、約7,000の遺伝子に分類されました。全遺伝子の約半分がcDNAクローンの形で手に入ったことになります。また調べてみますと、これまでにクローン化された遺伝子(tRNAなどは除きます)の70%以上がすでにこのセットに含まれていることがわかりました。15,000の遺伝子がすべて使われているとも限りませんので、徐々に飽和に近づいていると思えます。
5'側のタグ配列からは、コード情報だけでなく、選択的スプライシングがしばしば見つかります。また、遺伝子予測プログラムの検証にもなり、文字通りゲノム解析と車の両輪として補い合っています。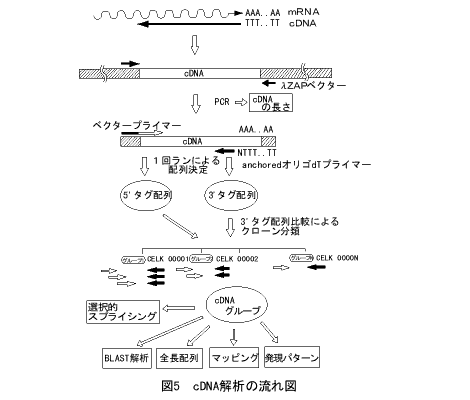
小原雄治著「線虫[1000細胞のシンフォニー]」(共立出版)より引用
- ゲノムデータベースACEDB
サルストンたちの基本姿勢は“archive and share”(情報は使いやすい形で集積して共有すべきである)という言葉に集約されます。彼はまさにこの通りに実行してきました。そのために、早くからいわゆるゲノムデータベースを構築し、その公開につとめてきました。1990年代に入って、大量の塩基配列情報を扱えるように、ダービン(Richard Durbin、現サンガーセンターの情報科学の責任者)とチエリーミグ(jean Thierry-Mieg、フランスCNRS)によって、それまでのVAX/VMS版からUNIX版の統合データベースACEDB(a.C.elegans database)に進化しました(図4参照)。現在では、いろいろな生物のデータベースがACEDBを用いてつくられており、ゲノムデータベースの定番の感があります。
ACEDBでは、コンティグ地図の情報と塩基配列の情報だけでなく、あらゆる他の情報がキーワードでリンクされています。変異体、かけ合わせ、文献、研究者、発現パターン、遺伝子破壊などの情報です。また、DNA配列から遺伝子を予測することもできます。小原雄治著「線虫[1000細胞のシンフォニー]」(共立出版)より引用
- コンティグ地図を軸とするさまざまな試み
現在では、コンティグ地図なしではC.elegans研究はありえないという状況になっています。これにもいくつかの理由があります。まず、DNAトランスホーメーションがうまくいくようになったことです。1章にも書きましたが、特別のベクターは不要で、コンティグ地図のコスミドクローンのDNAを線虫の卵巣に微量注射するだけで、突然変異のレスキュー実験ができるのです。また、遺伝子の大きさがせいぜい10kbですから、調節領域も含めてコスミドに楽々入ることもうまくいった理由です。おもしろい遺伝子が見つかったら、遺伝地図上にマップし、対応する領域のコスミドクローンをケンブリッジからもらって突然変異体に微量注射し、機能の回復を見る。これがクローニングの定石になりました。さらに、コスミドが決まったら、塩基配列は決定済みで、cDNAクローンも得られている、というのがしばしばです。まさに電話で(最近は電子メールで?)クローニングが実現しているのです。
しかし、遺伝子が何をしているのかは、塩基配列を見ただけではわかりません。まず、遺伝子がいつどこではたらいているのか、そしてその遺伝子を壊すとどうなるのかを調べていく必要があります。ゲノムの塩基配列からは出てこない情報をシステマティックに取り出して、ゲノムのはたらきの全体像にせまろうという研究が進行中です。遺伝子の発現パターン:遺伝子発現を見る
cDNAは遺伝子発現を見る方法はいくつかありますが、それぞれ一長一短があります。インシチュ・ハイブリダイゼーション法は、mRNAの存在を直接見ることはできますが、タンパク質の挙動はわかりません。レポーター遺伝子(lacZ、GFPなど)の発現を見る方法は、プロモーターや調節領域を調べることができますが、そのパターンが遺伝子のものと同じであるという保証はありません。抗体染色法は、遺伝子の機能産物であるタンパク質の挙動を直接見ることはできますが、大量の遺伝子に適用するには手間と費用がかかります。
私たちは、遺伝子発現の最初の段階である転写パターンをスクリーニングするために、インシチュ・ハイブリダイゼーション法を用いたアプローチを進めています。cDNA解析計画で分類されたcDNAグループについて、全発生ステージでのインシチュ・ハイブリダイゼーションの結果を集めるのです。さいわいC.elegansの胚を集めてくると、いろいろなステージが含まれていますし、これを切片でなく丸ごと(whole mount)の試料として処理できますので、1回の検鏡で線虫の一生のデータが得られます。
図6は、各遺伝子の各発生ステージでの発現パターンを並べたものです。これを「ゲノムの発現パターン地図」とよびます。ゲノムの全塩基配列決定が完了するまでには、この発現パターン地図もおおむね完成する予定です。これらの画像は、コンティグ地図とリンクさせた形でインターネット上で公開する予定です。また、発現パターンを細胞系譜ごとに分類する作業も進めており、調節領域の同定実験も開始しています。
イギリスのホープ(Ian Hope)は、リポーター遺伝子を使うアプローチで特異的な発現をつかさどるプロモーター/調節領域のクローニングを進めており、こちらもACEDBで公開されています。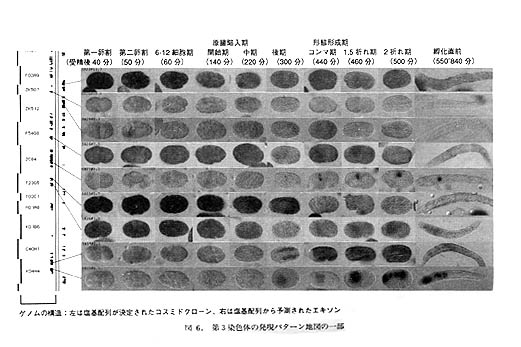
遺伝子を壊す
物理地図は、遺伝地図があってこそ生きてきます。また、遺伝子のはたらきを調べるには、最終的には突然変異体が必要です。そこで、すべての遺伝子について突然変異体を用意しよう、という考えが起こります。その一つが“saturation mutagenesis”です。領域を限って徹底的に突然変異体を分離するやり方です(方法は4章で述べましたので省きます)。致死変異をはじめとして、これまでに約2,000の遺伝子の突然変異体が得られ、遺伝地図上にマップされてきました。この数が15,000になればよいのですが、1つの変異では表現型が出ないこともあり、遺伝解析作業のたいへんさとあいまって、一挙には進みません。
それなら狙いうちで変異体をつくる方法が必要です。しかし、相同組換えによる遺伝子ターゲティングは実用化できていません。唯一の方法が、トランスポゾン高転移株を使って目的の遺伝子が転移したものをさがすことです。こちらも4章で述べていますが、オランダのプラスターク(Ron Plasterk)はこのことを利用して、あちこちにトランスポゾンが転移した線虫のバンクをつくり、ここからPCR法のスクリーニングするシステムをつくりました。C.elegansが凍結保存可能であることを最大限に利用した方法です。プライマーさえ用意すればよいので、多数の変異体がとられてきました。
てっとり早く遺伝子を不活性化するということでは、最近、アンチセンスRNA注入法が流行しています。線虫の卵巣に注射すると、そのメカニズムは不明な点が多いのですが、かなりの確率でその遺伝子を壊したときと同じ表現型を示します。cDNAクローンから簡単につくることができますので、cDNA計画からのスクリーニングのひとつとして研究者のあいだで広まっています。小原雄治著「線虫[1000細胞のシンフォニー]」(共立出版)より引用
- おわりに
「ポストゲノムシークエンス」という言葉が使われはじめました。設計図であるゲノムの塩基配列決定が、いろいろな生物で、時間の問題になってきたからです。そのあとの研究はどう進むのか、C.elegansがその先がけになりそうです(図7)。
まず、遺伝子の発現パターンです。mRNAとタンパク質の発現時期、細胞系譜、細胞内局在が系統的に明らかになってきます。ここからは調節領域の情報も次々と得られるでしょう。そして、突然変異体の系統的解析からの遺伝子の機能情報です。さらには、系統的なツー・ハイブリッド(two hybrid)法などによる遺伝子間相互作用の情報も得られてくるでしょう。これらが、ゲノムの塩基配列を軸に多次元のデータベースとしてリンクされます。発生過程の3Dグラフィックスをつくって、そこに情報をリンクさせ、たとえば、ある細胞をクリックすると、そこではたらいているすべての遺伝子の情報が得られるようなシステムを考えています。
このようなデータベースができたら、それこそ縦横の検索が可能です。ある細胞系譜で発現する遺伝子群、その順番、転写因子モチーフをもつ遺伝子、相互作用する組合せなどがわかれば、遺伝子発現カスケードが予測できるかもしれません。これの実験的検証、さらにはそのくり返しをしていくことになります。
ゲノム研究の成果でだいじなことは「遺伝子がこれで全部」ということです。関与する(可能性のある)遺伝子をすべてあげることができると、数にもよりますが、しらみつぶしの研究戦略が可能になってきます。たぶん、そのようにして初めて生物の全体像が見えてくるのだろうと思われます。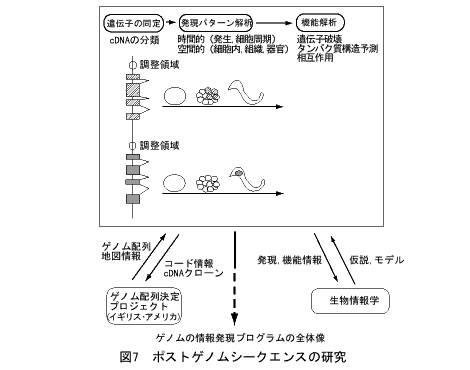
小原雄治著「線虫[1000細胞のシンフォニー]」(共立出版)より引用
線虫C.elegans実験法
- 何のためのC.elegansか?
土壌自活性線虫C.elegans(Caenorhabditis elegans)は、発生や行動の分子機構を研究するためのモデル生物として、1965年ごろに分子生物学者Sydney Brennerにより取りあげられた。Brennerは、遺伝学が使え、細胞数が少なく、体の構造に個体差が小さいという特徴をいかして、多細胞生物の発生や行動における遺伝子の論理を解明するという計画をもっていた。彼の弟子たちは、協力的な研究体制をつくって研究を展開し、全細胞系譜と全神経回路構造を解明し、データベースを整備した。そして最近では、逆遺伝学の手法も開発され、C.elegansは、ショウジョウバエと並ぶ発生遺伝学・行動遺伝学の代表的なモデル生物として世界に広まった。また、C.elegansでは、全ゲノムの塩基配列が決定されており、遺伝子破壊やGFP融合遺伝子による発現解析などの手法を組み合せて、ヒトの遺伝病原因遺伝子や癌遺伝子などの個体レベルでの機能や作用機構を調べるための「生きた試験管」として注目されている。
桂勲、石原健著「実験医学, Vol.16, No.1, 71-77, 1998」(羊土社)から引用
- C.elegansとはどんな虫か?
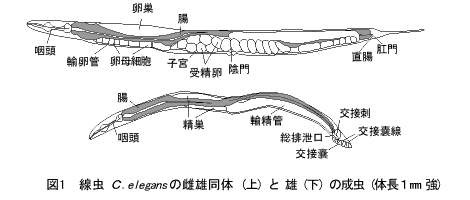
C.elegans(図1)は、非寄生性の土壌線虫で、体長約1mm、世代時間は20℃で約3日である。ゲノムサイズは約108塩基対(大腸菌の20倍、ヒトの1/30)で、性染色体(X)と5対の常染色体(I~V)をもつ。自家受精する雌雄同体(XX)と、それと交配できる雄(XO)がいる。雌雄同体は約3日間のうちにおよそ300個の受精卵を生み、卵は約半日で孵化し、1齢~4齢の幼虫を経て成虫になる。成虫の細胞数(厳密には体細胞核数)は雌雄同体が959個、雄が1,031個で、前述のように個体差がほとんどない。体や卵殻が透明なため、微分干渉光学系を用いると、すべての発生段階で、生きたまま全細胞核を観察・同定できる。このような長所をいかして、C.elegansではさまざまな実験法が開発されている。
桂勲、石原健著「実験医学, Vol.16, No.1, 71-77, 1998」(羊土社)から引用
- C.elegansの情報・クローン・変異体の入手法
C.elegansの情報・クローン・変異体の入手法を以下にまとめた。なお、頼むときには、自己紹介と使用目的を簡潔に書く。
基礎知識を得たい場合
書籍としては、日本語の入門書1)、本格的な英文総説2)3)、詳細な実験法4)の決定版が出ている。実験法は、Ambros Lab Comprehensive Protocol Collectionにも有用な情報がある。
国内のC.elegans研究者を知りたい場合
C.elegansに興味をもつ日本国内の研究者のグループ「虫の集い」がある。親睦会は毎年、日本分子生物学会年会の会期中に会場近くで開かれる。事務局のホームページは、勉強会や親睦会の情報がわかるほかに、C.elegans関係のリンクがある。mailing listにもなっている。mailing newsgroupはbionet.celegansにもある。
C.elegansの野生型や変異型の虫を入手したい場合
CGC(Caenorhabditis Genetics Center)は、現在はミネソタ大学のR.K.Herman研究室にあり、C.elegansの種々の株の蒐集と配布、Worm Breeder's Gazette(ニュースレター、WBGと略す)の発行、C.elegans関連の論文リストの作成、変異・マッピングなど遺伝学データの蒐集(この部分は英国MRC-LMBのJ.Hodgkinが責任者)を行っている。変異体が欲しいときは、CGCのGopher server(gopher://elegans.cbs.umn.edu:70/11/)にある“CGC Strain List”で存在を確認した後に、curator(現在はTheresa Stiernagle)に注文する。注文時に使用目的と研究費の出所を明記すること、論文にCGCに対する謝辞を入れ別刷を2部送る事が義務となる(詳細はGopher server中の“About CGC”参照)。(連絡先:Theresa Stiernagle, Caenorhabditis Genetics Center, University of Minnesota, 250 Biological Sciences Center, 1445 Gortner Avenue, St. Paul, MN 55108-1095, USA, E-mail : stier@biosci.cbs.umn.edu, FAX:+1-612-625-5754)
C.elegansのゲノム塩基配列やcDNA(EST)配列の情報を得たい場合
英国(Sanger Centre)、米国(Washington医大)でBLAST searchができ、コスミドなどの塩基配列も入手できる。(英国:http://www.sanger.ac.uk/Projects/C_elegans/blast_server.shtml、米国:http://genome.wustl.edu/gsc/allelegans_blast/all_ele_blast_server.html)。ESTに関しては遺伝研の小原博士のC.elegans Indexで検索などが可能である(http://www.ddbj.nig.ac.jp/htmls/c-elegans/html/CE_INDEX.html)。
C.elegansのゲノムデータベース(ACEDB)を使いたい場合
ACEDBに関しては虫の集い事務局ホームページにあるACEDBのユーザマニュアルを参照のこと。(1)wwwで直接アクセスする方法(https://www.sanger.ac.uk/science/tools/acedb)と(2)UNIX版ACEDBをワークステーションに、あるいは(3)Macace、Winace(現在β版)をダウンロードしてから使う方法がある。(1)は、通常のインターネットの端末があればすぐにできるが、検索に時間がかかる。(2)、(3)は基本的に同じだが、UNIX版の方がupdateが早いようだ。また、データ量が多いので、ダウンロードするのに非常に時間がかかる。
ゲノムDNAクローン、cDNAクローン、GFPベクターの入手先
ゲノムDNAクローン(コスミドやYACのクローン、YAC polytene filter)は、Dr.Alan Coulson(Sanger Centre, Hinxton Hall、Cambridge CB10 15A, ENGLAND:FAX+44-1223-494919,E-mail:alan@sanger.ac.uk)に、ykの頭文字のついたcDNAクローンは、小原雄治博士(国立遺伝学研究所、ykohara@lab.nig.ac.jp)に、いずれもACEDBなどでクローン番号を確認のうえ、申し込む。
[参考文献]
- 「線虫 —1000細胞のシンフォニー—」(小原雄治編;ネオ生物学シリーズ第5巻)共立出版, 1997年
- "The Nematode Caenorhabditis elegans"(W.B.Wood, ed.) Cold Spring Harbor Laboratory Press, Cold Spring Harbor, 1988
- "C. elegans II"(D.L.Riddle, T.Blumenthal, B.J.Meyer, J.R.Priess, eds) Cold Spring Harbor Laboratory Press, Cold Spring Harbor, 1997
- "Caenorhabditis elegans: Modern Biological Analysis of an Organism"(H.F.Epstein & D.C.Shakes, eds.) Methds in Cell Biology, Vol.48, Academic Press, 1995
桂勲、石原健著「実験医学, Vol.16, No.1, 71-77, 1998」(羊土社)から引用
- プロトコール
C.elegansの飼い方
分子生物学の研究室でC.elegansの研究を始めるには、とりあえず実体顕微鏡と低温インキュベーターが必要である。大腸菌が厚く生えると観察の邪魔になるので、通常は大腸菌OP50(Ura-)を餌としてNGM培地上で飼う。
C.elegansを飼うには、まずNGMプレートをつくる。餌となる大腸菌OP50の一晩培養液を1mlのメスピペットで約30μlずつ、シャーレの端にふれないようにプレートに塗る。交配用のプレートでは、中央付近に直径1cmの円状に塗る。2~3日室温に放置、または37℃で一晩培養。1)植え継ぎ方
下記A、Bのいずれかの方法で行うことが多い。
A. 実体顕微鏡下でwormpick(ガラス管の先に,先端が尖った太さ0.1mmの白金線を付けたもの.バーナーで滅菌して用いる)を使って虫をすくい取り、大腸菌の生えた新しいプレートの表面にwormpickの先端を付け、虫が這い出すのを待つ。寒天を傷つけるとあとから虫が寒天に潜るので注意。
B. 実体顕微鏡で虫がいることを確認し、滅菌した爪楊枝で寒天の小片を切り取り、大腸菌の生えた新しいプレートの上に置く。2)コンタミの除き方
虫を培養しているプレートにカビ・菌などがコンタミした場合は、端に大腸菌を生やしたプレートを用意し、反対の端に幼虫を置く。一晩後に大腸菌の所へ行った虫を新しいプレートに移す。これで除けないほどひどい場合には次亜塩素酸処理(後述)する。
3)雄の飼育
雄は常に雌雄同体と交配して維持する。最初の雄は、熱ショックにより(染色体不分離の頻度を増加させて)つくる。
(1)L4(4齢幼虫:図3参照)の雌雄同体約10匹を新しいプレートに入れ、30℃、6時間の熱ショックの後に、20℃で育てる。
(2)3~4日後に約1%の割合で育ってくる雄と雌雄同体を、交配用のプレートにwormpickを使って移す。通常、L4の雌雄同体を3匹、L4の雄を7匹(図3参照:L4の雄が少ない場合は若い成虫の雄も使う)入れて20℃で4日飼うと、子供に多数の雄が出る(交配でできた子供の1/2が雄)。これを同様に植え継ぐ。
<ポイント>
熱ショックの時間が長すぎると子供の数が減り、短すぎると雄の割合が少ないために失敗する。
さらに詳しくは、実験医学Vol.16 No.1(1月号)1998 桂 勲、石原 健を参照。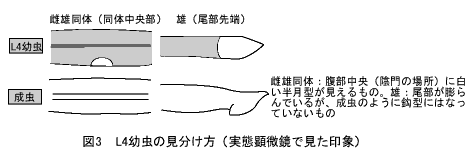
桂勲、石原健著「実験医学, Vol.16, No.1, 71-77, 1998」(羊土社)から引用
- 用語解説
土壌自活性線虫:
線虫類の中には、カイチュウなど動物に寄生するもの、マツノザイセンチュウなど植物に寄生するものもあるが、C.elegansのように地中にすみ細菌を食べて一生を送るものも多い。C.elegansは、世界各地の温帯の地中から採られており、日本の地中からも採ることができる。
細胞系譜:
受精卵が分裂して成体(ここでは成虫)になるまでの細胞の系図のこと。哺乳類では、一卵性双生児のように遺伝子が全く同じでも、個体ごとに細胞系譜が異なる。しかし、C.elegansでは生殖細胞以外では個体差はほとんどなく、1個の受精卵がどの虫でも同じように分裂して、959個の細胞からなる成虫の体ができあがる。
神経回路構造:
神経細胞はシナプスと呼ばれる部分で別の神経細胞に信号を伝えている。ここでいう神経回路構造とは、具体的にどのような神経細胞がいくつあり、それらがシナプスを介してどのようにつながっているかを意味する。C.elegansでは、電子顕微鏡で302個の神経細胞からなる全神経回路構造が解明されている。
データベース:
C.elegansに関する遺伝子地図、遺伝子クローン、塩基配列、全細胞の名前、変異体、遺伝子発現の場所と時期、参考文献、研究室名などの情報がコンピューターの中に集められ、インターネットで調べられるようになっている。
逆遺伝学:
昔からの遺伝学では、まず特定の表現型に注目して変異体を選択し、次に交雑を行なって遺伝子地図上での変異の位置を決め、さらに変異を起こした遺伝子をクローニングする。(通常はその後で、その遺伝子の変異のためになぜそのような表現型が生じるかを調べて、発生などのメカニズムを解明する。)ところが、多くの遺伝子がクローニングされている現在では、まず特定の塩基配列を持つ遺伝子(例えば特定の酵素の遺伝子)に注目し、次に生物個体内のその遺伝子に変異を導入して、どのような表現型が現れるかを調べる研究も行なわれるようになった。古典的な遺伝学では「表現型から遺伝子へ」と研究が進むのに対し、このような研究では「遺伝子から表現型へ」と進むので、逆遺伝学と呼ばれている。
ゲノム:
1つの生物個体または細胞がもつ全遺伝情報のこと。C.elegansのゲノムの大きさ(ゲノムサイズ)は、約1億塩基対である。
GFP融合遺伝子:
GFPとは、クラゲの1種が作る緑色蛍光タンパク質のことである。遺伝子発現の場所と時期を調べたい遺伝子に、GFPの遺伝子をつなげてGFP融合遺伝子を作り、生物(ここではC.elegans)に導入すると、その遺伝子の発現を蛍光として顕微鏡で見ることができる。
桂勲、石原健著「実験医学, Vol.16, No.1, 71-77, 1998」(羊土社)から引用

